

一种整合了所有在BioPlex中亲和纯化后的人类蛋白图谱免疫荧光图像,进而创建了人类细胞结构的统一层次图谱,即MuSIC。
背景介绍
真核细胞由细胞器等大组件组成,而细胞器又可分解成冷凝物和蛋白质复合体等小组件,形成复杂的多尺度结构。绘制亚细胞结构的基本技术有蛋白质成像和生物物理关联,且每一种都实现了自动化。尤其是共聚焦显微镜和免疫荧光技术的进步,使得扫描单个细胞内原位蛋白质的分布成为可能。通过将这些技术与抗体库相结合,人类蛋白图谱(Human Protein Atlas, HPA)已经开始系统地将人类蛋白质研究定位于亚细胞区室。作为一种并行的细胞定位方法,质谱(MS)与亲和纯化(AP-MS)和邻近依赖标记强有力地结合在一起,使蛋白-蛋白相互作用的快速测量成为可能。利用AP-MS, BioPlex项目正在为大多数人类蛋白质生成全面的相互作用图谱。
一个关键问题是:如何将成像和生物物理关联结合起来以展示细胞结构。图像定位蛋白质相对于细胞核等细胞标志,而生物物理关联则是将蛋白质相对于邻近蛋白进行定位。在这两种情况下,由于机器学习系统在数据中识别复杂模式的能力,使得这种定位变得越来越定量化。
主要内容
本文中,来自瑞典斯德哥尔摩皇家理工学院及美国斯坦福大学的Emma Lundberg和美国加州大学圣地亚哥分校的Trey Ideker等人演示了一种机器学习方法,将蛋白成像和生物物理关联集成在一起,以创建亚细胞组件的统一图谱(图1)。首先,研究者使用神经网络在成像或生物物理关联的基础上,将蛋白质投射到低维度。一旦确定了每个平台的蛋白质坐标后就对蛋白间的两两距离进行校准和组合,以显示不同尺度下(从小于50 nm到大于1 μ m)的蛋白质组合。相关的研究成果以“A multi-scale map of cell structure fusing protein images and interactions”为题发布在国际顶级期刊Nature上。
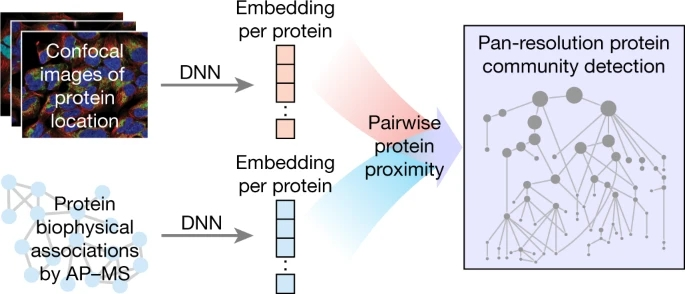
图 1. 数据融合策略概述。图片来源于Nature
两种方式的蛋白质的位置和距离
研究者收集了来自HPA的免疫荧光图像和来自BioPlex的AP-MS数据的匹配数据集。这两种资源都是基于人类胚胎肾(HEK293来源)细胞,共产生了661个蛋白质,具有兼容成像(1451张图像包括复制)和生物物理关联数据(291个蛋白质亲和标记为“诱饵”,370个蛋白作为相互作用的“猎物”) 。
接下来研究者根据免疫荧光和AP-MS数据,使用深度神经网络嵌入每个蛋白。嵌入是复杂输入的一种低维表示,其中每个数据点(这里是蛋白质)都被赋值为降维坐标。在图像嵌入中,研究者使用了卷积神经网络——DenseNet,其在捕获相对于反染色细胞标志物的蛋白位置方面具有优越的性能。类似地,使用node2vec神经网络扩展的AP-MS邻近交互来嵌入每个蛋白质。
然后,研究者分别在免疫荧光和AP-MS嵌入中计算所有蛋白对的距离。研究者组装了已知或估计直径的亚细胞组分参考集,包括了从小于20 nm的蛋白质复合物到大于1µm的细胞器。利用这些经过筛选的直径作为训练标签,作者训练了一个有监督的机器学习模型(随机森林回归),直接从免疫荧光和AP-MS嵌入的坐标中估计任意蛋白质对的距离。
亚细胞系统的多尺度图谱
研究者分析了661个蛋白质之间的所有距离以识别相互接近的蛋白群落,这些群落表明不同的组件(图2)。随着阈值的放宽,较小距离的群落被完全或部分地包含在较大的群落中,从而产生一个结构层次(图3a)。这样可以观察到广泛的群落检测参数,且与大小型群落都一致的独立数据集。最终的层次结构,即MuSIC 1.0,它包含69个蛋白质群落,代表了有87个层次包含关系的假定的亚细胞系统(图2)。16个系统包含在多个较大的系统中,表明了多个亚细胞定位或多向性。
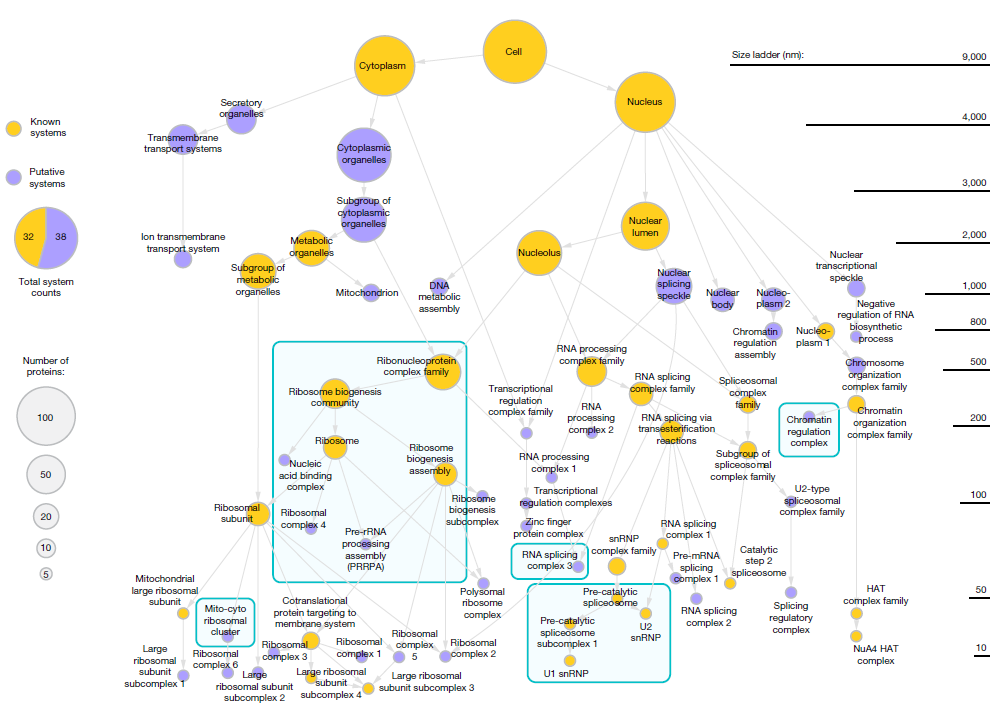
图 2. 多尺度集成单元。图片来源于Nature
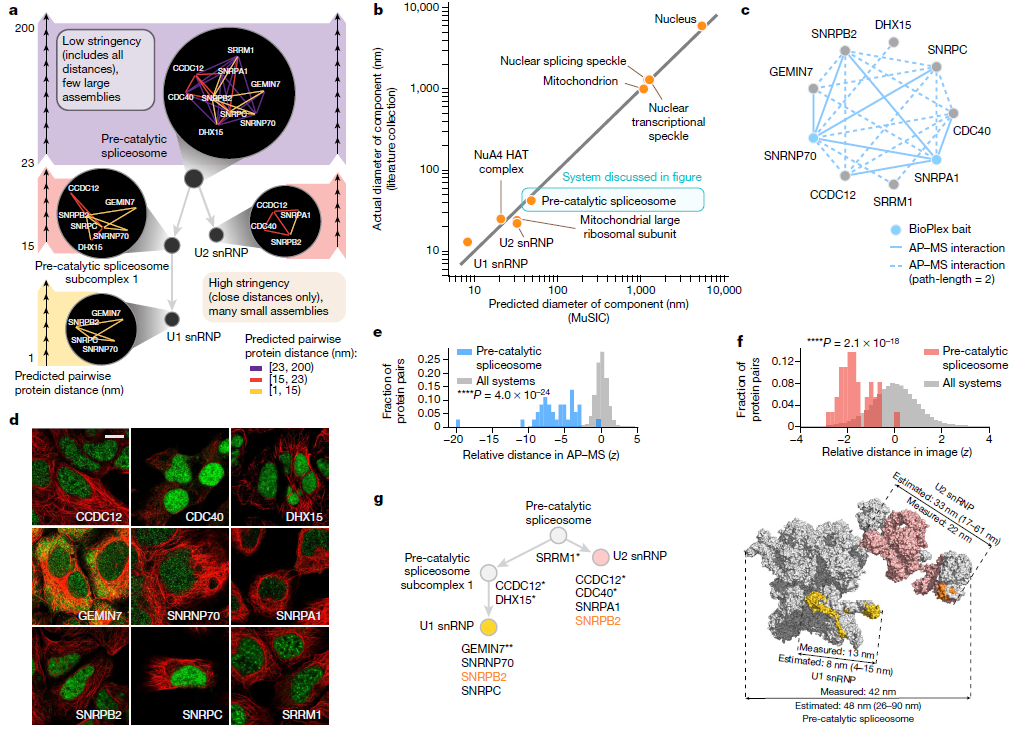
图 3. MUSIC捕获的亚细胞组件和直径。图片来源于Nature
MuSIC系统的物理尺寸是根据它们的配对蛋白质距离估算的(图2),并与9个先前未在校准中使用的特征良好的细胞组分的已知直径进行了比较(图3b)。其中一个组分是催化剪接体,免疫荧光和AP-MS数据支持(图3c-f)诱导的蛋白群落为48 nm(95%预测区间[26,90]),与其公布的直径42 nm(图3a, g)一致。在该群落内,分析解决了较小的U1和U2子单元(U1: 8 nm, 95%预测区间[4,15];U2: 33 nm, 95%预测区间[17,61]),同样与低温电子显微镜测量的排列和距离一致(图3g)。对于所有9个组件,估计的直径与文献中的实际测量值非常接近(图3b),验证了MuSIC可以捕获和测量大尺度生物系统。
MuSIC需要并告知这两种数据类型
研究者发现,大多数系统对数据的轻微破坏都是健壮的(图4a,刀切重采样)。相比之下,仅使用一种数据类型构建的替代MuSIC图谱则会丢弃许多系统。免疫荧光图谱倾向于识别大型系统,如细胞器;但对小的亚组分,如蛋白质复合体则不确定,AP-MS图谱的行为则刚好相反(图4b-d)。值得注意的是,30%的AP-MS相互作用发生在少于100个蛋白质的集中系统中(图4e),这验证并提供了相互作用的位置背景。这样的环境也增加了相互作用检测的敏感性:集中于之前BioPlex研究中没有报道过相互作用的蛋白质对,尽管如此,小系统中的蛋白质对比大系统中的蛋白质对有更强的AP-MS评分(P < 0.0001;图4f),表明了新的真实的物理交互作用。
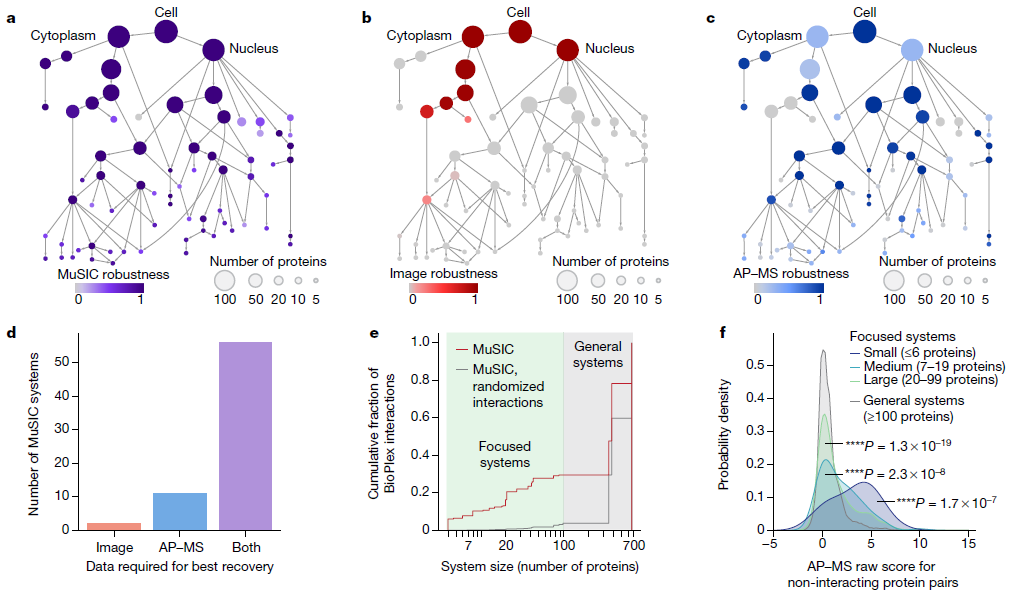
图 4. 不同的数据提供不同规模的信息。图片来源于Nature
新AP-MS对MUSIC的全局验证
在661个MuSIC蛋白中,有370个尚未在AP-MS实验中作为诱饵进行亲和标记。它们出现在被另一种亲和标记蛋白质分离出来的猎物蛋白列表中。作为验证候选系统的直接手段,亲和标记了134个前猎物蛋白质并进行了AP-MS,结果识别出了339个物理相互作用。44个MuSIC系统专门针对新的相互作用进行了改进(64%;错误发现率(FDR) < 0.1)(图5a),包括23个假定的候选项。
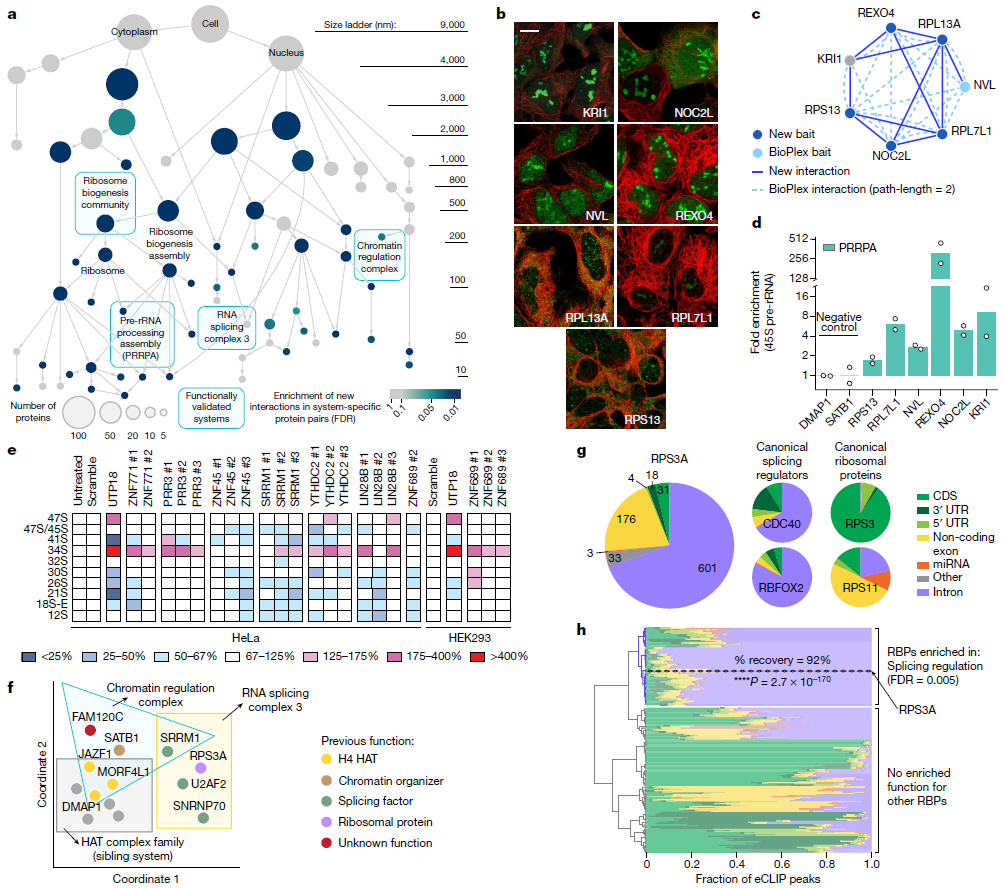
图 5. 使用物理和功能分析的MuSIC探索。图片来源于Nature
多尺度的核糖体系统
通过附加AP-MS数据验证的候选蛋白中,有7个蛋白组装,直径估计为81 nm(95%预测区间[43,151])。基于已建立的两个蛋白(NVL, RPL13A)的pre-rRNA作用、遗传筛选(KRI1, NOC2L)的支持以及酵母中的pre-rRNA因子(REXO4)的同源性,研究者初步将该系统命名为pre-核糖体RNA处理组装(PRRPA)。这些蛋白质由于图像相似、核仁定位和AP-MS网络邻域相似而形成一个系统(图5b, c)。通过针对5种PRRPA蛋白的新的亲和纯化,恢复了高度特定于该系统的相互作用伙伴(图5c)。然后,研究者使用RNA免疫沉淀和定量PCR (RIP-qPCR)时,发现这些蛋白质结合了45S pre-rRNA,再次表明pre-rRNA的处理作用(图5d)。
研究者还检测了包含PRRPA的大尺度系统,“核糖体生物起源群落”(347 nm, 95%预测区间[186,646])。该系统包含了与核糖体生物起源无关的额外蛋白质,其中7个被研究者用靶向Dicer-底物siRNAs (DsiRNA)进行了敲除。所有7个都对前-RNA处理有影响,按受影响的特定前-RNA可进行分层(图5e)。其中三个蛋白在研究者新的AP-MS实验中被定位(LIN28B, PRR3, ZNF689);每一种都能结合相同群落中的大量蛋白质。
染色质和拼接
SRRM1是一个已建立的剪接因子,除了其在RNA剪接复合体3 (71 nm, 95%预测区间[38,133])中的典型位置外,还参与到了其他意想不到的系统中。染色质调节复合物(211 nm, 95%预测区间[113,393]),包括三种组蛋白乙酰转移酶(HATs) (DMAP1, JAZF1和MORF4L1)和SATB1,它们通过HAT增长来重塑染色质(图5f)。这些功能表明,该系统中剩余的蛋白质SRRM1和FAM120C也参与调节染色质。
总结
本文开发了一种通过神经网络嵌入每个蛋白质来测量接邻近性的系统方法。这种分析还与其他类型的信息相集成,并在多个尺度上重塑组件,包括可以进行物理和功能验证的新系统。在这项工作中,AP-MS的物理交互促进了正确的分配。MuSIC中的系统覆盖了多个尺度,桥接并超越了免疫荧光和AP-MS的范围。
此外,新的蛋白系统可能伴随着附加的数据模式而出现,如邻近依赖标记、交联质谱或低温电子显微镜。探索这些平台之间的协同作用将是很有趣的工作,所有这些平台都可能用来校准从而测量分子距离,进而有助于绘制多尺度细胞的图谱。
参考文献
Qin, Y., Huttlin, E.L., Winsnes, C.F. et al. A multi-scale map of cell structure fusing protein images and interactions. Nature (2021). https://doi.org/10.1038/s41586-021-04115-9