

多核图卷积网络(GCMNK)将学习到的药物特征输入到具有三个全连接层的block中来预测DDI,AUC-ROC达到0.95,远远优于其他DDI预测方法。
背景介绍
药物-药物相互作用(DDI)是指对一种药物组合的药理和临床反应,不同于两种药物单独使用时的药效。通过预测药物与药物、疾病、靶标的相互作用来寻找新药是一种常见的CADD手段。DDI预测有助于研究人员深入了解药物的作用机制。
目前文献中有许多基于药物特征来预测 DDI的方法。有基于药物-药物相似性,利用 k 最近邻程序并采用正则化最小二乘分类器来预测潜在的 DDI,也有先用SVM 从未知样本中产生负样本并建模来预测DDI,还有使用多模态自动编码器(MDAE)预测潜在的DDI。
DDI 可用于构建 DDI 图,图的节点是药物,边代表药物之间的相互作用。Zhou在 DDI 图上使用马尔可夫聚类算法来预测潜在的药物组合。研究人员可以将药物特征与网络结构相结合来预测潜在的相互作用。Zhang在 DDI 图上使用随机游走算法,基于药物-药物相似性矩阵计算转移概率。
当前的DDI 预测方法中,研究人员将已知的 DDI 都标记为阳性样本并用于构建 DDI 图。然而,有些药物组合会增强药效,有些反而会减弱。这是当前文献未考虑的。
在这项工作中,加拿大萨斯喀彻温大学吴方向教授团队提出了用一个多核图卷积网络 (GCNMK) 来预测潜在增加的 DDI。首先分别从与增加相关和与减少相关的 DDI 构建一个增加的 DDI 图和一个减少的 DDI 图。结合两个GCN层,用这两个图和各种类型的药物特征来学习药物的低维表示向量。生成节点嵌入后,将两个药物向量连接起来作为 DDI 的向量。最后,使用具有三个全连接层的block预测。
模型搭建
GCNMK模型的架构如图1所示。首先建立了药效增强的DDI图和药效减弱的DDI图,然后将这两种图分别输入两个GCN模块中(图1A)。两个block分别组成GCN L1层和L2层,一个加和操作将两个药效增加和药效减弱的输出合并(图1B)。两个药物的特征向量拼接形成DDI的特征向量(图1C)。最后用三全连接层的网络预测药物相互作用结果。
药效增加和药效减少的GCN模块,不仅从各自输入的分子图中学习特征表示,还要吸收彼此线性变换的输出信息。在加入彼此的输入后经过激活函数输出HI1和HD1。最后由block3融合了HI1和HD1得到DDI的特征向量。
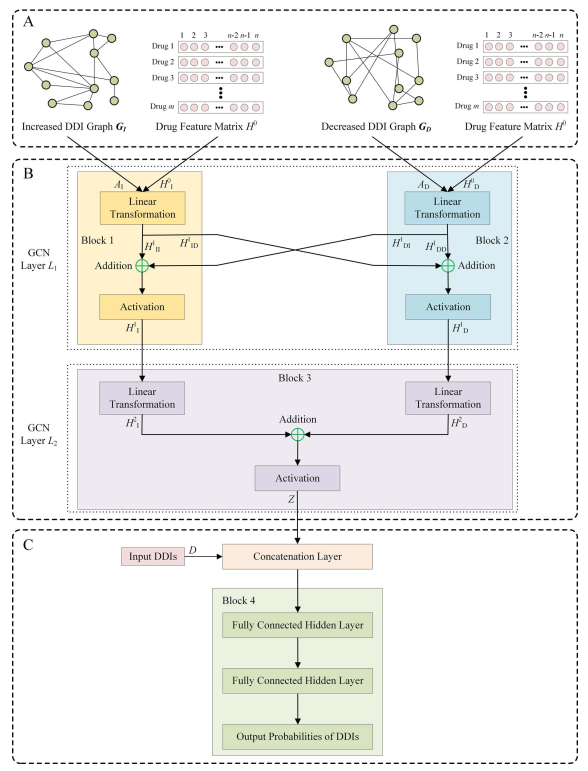
图1. GCNMK的架构。图片来源:BIB
Block 4以DDI的特征向量为输入来预测药物存在相互作用的概率,如图 1C 所示。GCNMK 模型使用交叉熵作为损失函数。另外作者还采用了L2正则化防止模型过拟合。
模型评价
在研究中作者使用 5 折交叉验证来评估GCNMK和文献方法的预测性能。以已知的 DDI (80702条)表示为正样本,未知的 DDI(106876条) 表示为负样本。
为了使训练数据平衡,作者随机选取了80702个负样本。将正样本和选择的负样本随机分为五个子集,每次选择一个正子集和一个负子集作为测试集,其余的子集作为训练集。五次后,所有子集都用作测试集,并产生预测结果。为了避免在训练过程中使用测试信息,在每次训练时从GI和GD中删除测试集中的DDI。
使用AUC-ROC和AUC-PR来衡量模型表现。值越高,模型越可靠。图2分别展示了描绘不同lr值(最佳lr为 0.002)、不同L2 正则化系数 λ(最佳λ为 0.0003)、不同嵌入大小d(最佳d为160)及不同特征类型下的AUC-ROC和AUC-PR值的直方图。node2vec和PRL 特征效果最差,其他六类特征的AUC-ROC和AUC-PR差异不大,药物的目标特征在其中取得了最好的预测效果。
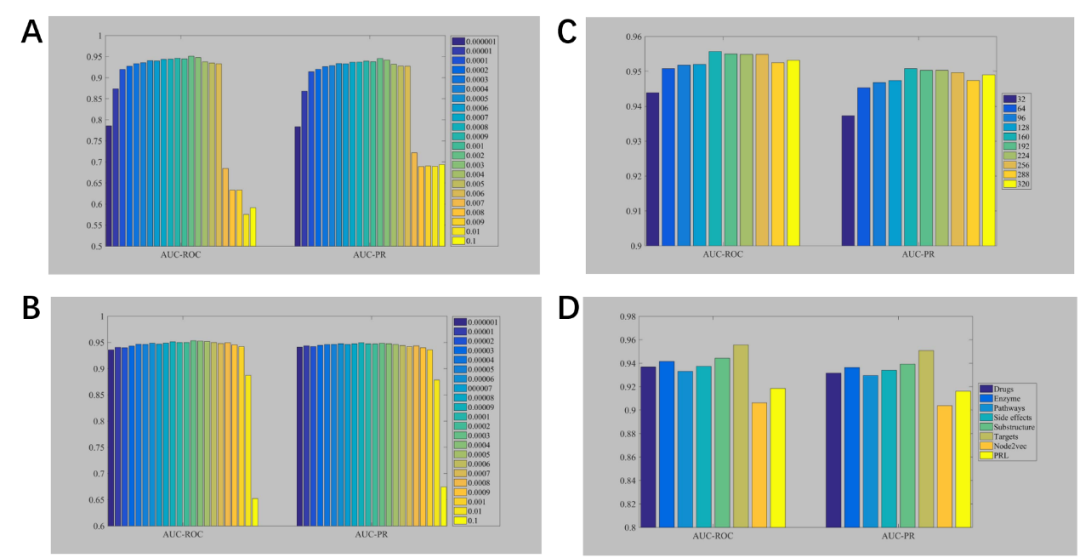
图2. 不同(A)学习率, (B)正则化系数,(C)嵌入大小,(D)特征类型对模型性能的影响。图片来源:BIB
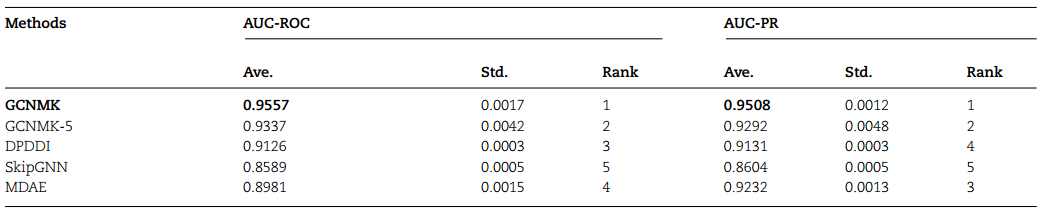
研究者比较了GCNMK和三种 DDI 方法,它们是 DPDDI、SkipGNN 和 MDAE。如他们的方法中所述,将参数设置为最佳值。测试结果如表1所示。
表1. 四种预测方法对比。表格来源:BIB
可视化分析嵌入特征
为了研究GCNMK的嵌入效果,研究人员使用 t-SNE从模型中学习到的嵌入特征来可视化 DDI。如图 3所示。绿点是已知的 DDI,红点是未知的 DDI。可以看到大部分点聚集在两个区域。已知的 DDI 位于图的下半部分,而未知的 DDI 位于图的右上四分之一,这可以很好地解释模型的效果。
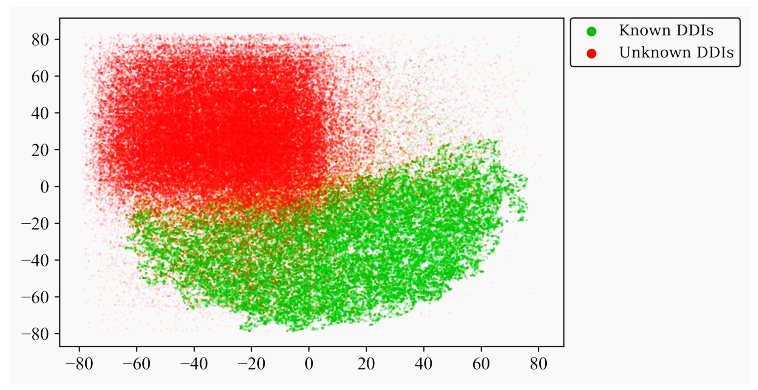
图3. 嵌入的t-SNE的可视化分析。图片来源:BIB
模型比较
为了公平比较各种类型的特征和算法,作者下载了DrugBank的 DDI数据。接着在实验中比较了8种类型的特征。相关的药物特点、酶、副作用、子结构、靶标等信息来自于DrugBank,KEGG等数据库(如表2所示)。
表2. 特征的类型、维度和来源。表格来源:BIB

作者在不同方面将 GCNMK 模型与其他模型进行比较。DPDDI方法中只有一个图核 ,GCNMK产生的AUC-ROC和AUC-RP值比DPDDI大4%左右。当使用相同类型的特征时, GCNMK 模型仍然实现了比 DPDDI 更好的性能。结果表明,使用两个 DDI 图 GI 和 GD 可以提高预测性能。
SkipGNN 中有两个图内核,GCNMK 生成的 AUC-ROC 和 AUC-RP 值比 SkipGNN 大 10%。一个可能的原因是在数据集中,Gall 中边的比例约为 43%,而在 Gskip 中接近 95%。添加这样一个几乎全连接的图并不能提高预测性能。GCNMK在 AUC-ROC 和 AUC-PR 两个指标中实现了所有竞争方法中最好的预测效果。
案例研究
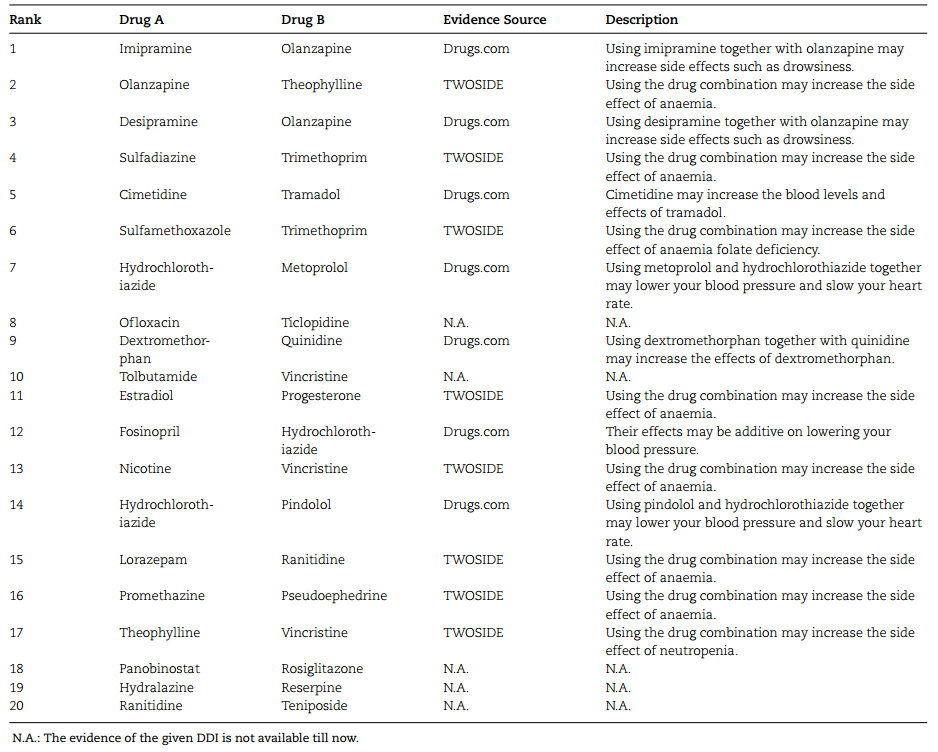
所有 106 876 个未知 DDI 都被输入到 GCNMK 模型中。两种药物的较大预测分数表明它们具有较高的相互作用概率。根据它们的预测分数生成降序排列的 DDI 排名列表。
表 3 中列出了前 20 个预测的 DDI,并从文献中收集有关它们相互作用的描述。作者在文献中确认15个DDI。结果表明, GCNMK 模型在预测新的 DDI 方面是有效的。
表3. 预测最靠前的20个DDI。表格来源:BIB
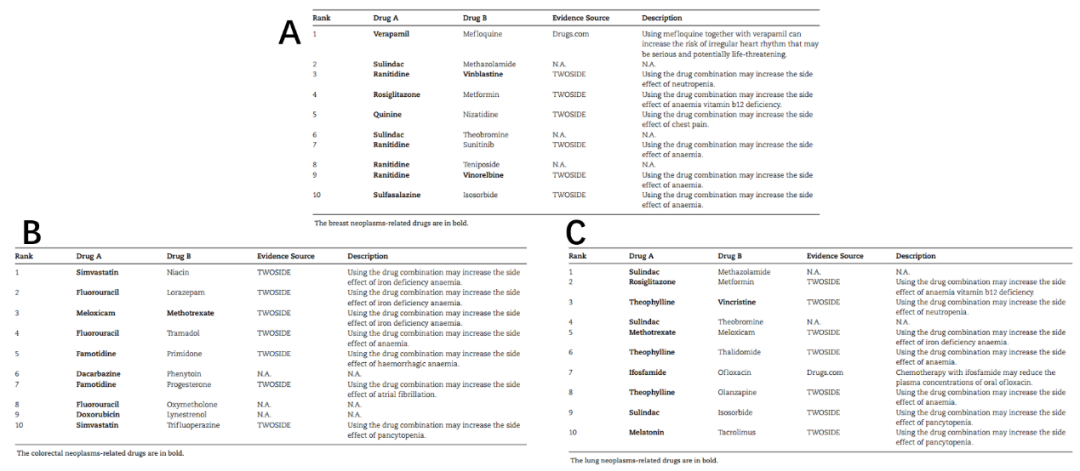
为了研究与给定疾病相关的潜在 DDI,作者从 CTD 数据库中生成与乳腺癌、结直肠癌、和肺癌相关的药物,并预测这些药物相关的未知 DDI。预测结果列于表 4。在乳腺肿瘤相关 DDI 的预测结果中,10 个 DDI 中有 7 个在 TWOSIDE 或 Drugs.com 中已被证实具有相互作用。尤其是有两种已确认的DDI,每一种均由两种乳腺肿瘤相关药物组成。在结直肠肿瘤相关DDIs的预测结果中,10个DDIs中有7个被证实在TWOSIDE中有相互作用。在肺肿瘤相关 DDI 的预测结果中,10 个 DDI 中有 8 个已在 TWOSIDE 或 Drugs.com 中被证实具有相互作用。这些与肿瘤相关的案例研究证明了GCNMK 模型在识别特定疾病相关药物的潜在 DDI 方面的有用性。
表4.(A)乳腺癌,(B)结直肠癌,(C)肺癌预测最靠前的10个DDI。表格来源:BIB
结论
在这项研究中,作者提出了一个用于预测 DDI 的 GCNMK 模型并有效地预测了三种癌症药物的DDI。在 GCNMK 的 DDI 图中,边属于同一类型。可以将其应用于任何异构网络,例如药物-疾病网络。药物-疾病关联的描述包括两种类型:治疗和标记/机制,这可能有助于采用 GCN 模型。另一个未来的方向是区分更多类型的预测DDI。根据它们的功能,每种类型的 DDI 都可以用来构建图内核,并且该模型具有识别预测 DDI 的特定类型的潜力。
参考文献
Fei Wang, Xiujuan Lei, Bo Liao and Fang-Xiang Wu. Predicting drug–drug interactions by graph convolutional network with multi-kernel. Briefings in Bioinformatics, 2021, 00,1–11.