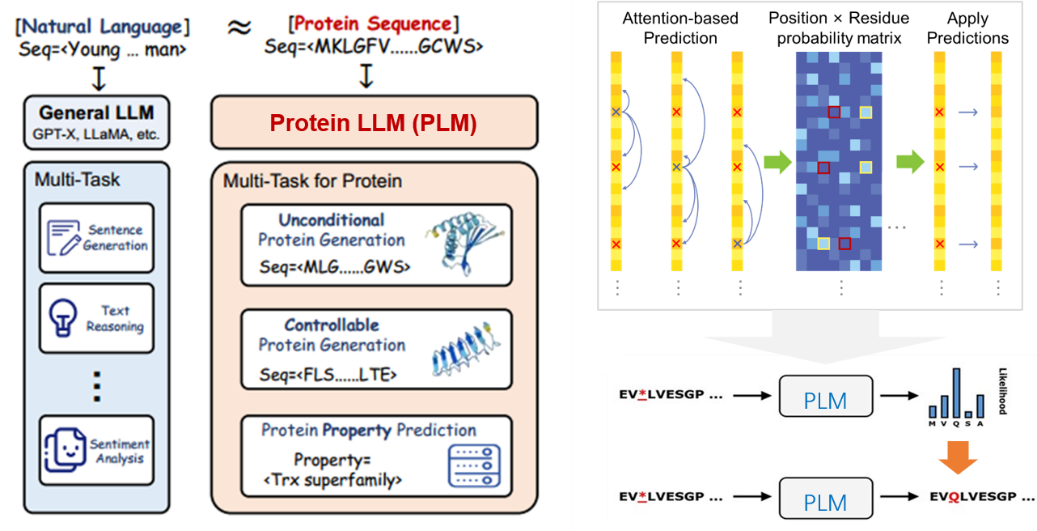
蛋白质语言模型是一类应用自然语言处理(NLP)技术来分析和理解蛋白质序列的模型。这些模型借鉴了语言模型在文本处理中的成功经验,通过将蛋白质序列视作由氨基酸组成的“句子”,来预测和生成新的序列、识别功能域、理解蛋白质结构和功能等。这种技术不仅可以帮助研究人员识别哪些突变可能是有效的,还可以预测这些突变是否会提高蛋白质的功能。进而指导蛋白质活性以及可开发性等多种性质的改造。
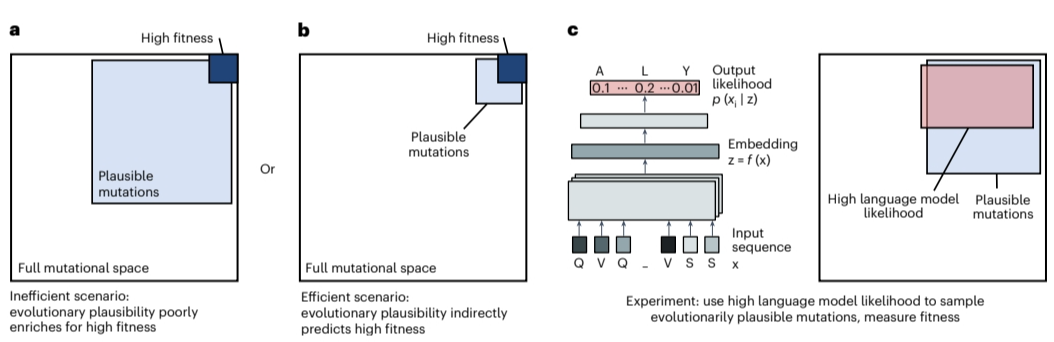
例如,斯坦福大学Peter S. Kim团队在2023年在《自然生物技术》上发表的一项研究,展示了如何利用通用蛋白质语言模型有效地进行抗体亲和力的改造。具体步骤如下:
1. 模型训练 研究团队使用了ESM-1b和ESM-1v两个语言模型,这些模型分别在UniRef50和UniRef90数据库上进行训练。这些数据库包含了数百万个自然界中观察到的蛋白质序列,涵盖了广泛的蛋白质变异。
2. 突变选择 通过语言模型计算所有单个氨基酸替换的进化可能性。选择那些进化可能性高于野生型的替换,并通过实验筛选这些替换是否能够改进抗体的结合亲和力。
3. 实验筛选 对每种抗体进行两轮筛选。在第一轮中,筛选单个氨基酸替换的变体。在第二轮中,筛选组合了多个有利替换的变体。通过生物层干涉技术(BLI)测量这些变体的抗原结合强度。
4. 亲和力测定 针对临床相关的抗体,测量它们的单价抗原结合片段(Fab)的解离常数(Kd);对于未成熟的抗体,测量双价免疫球蛋白G(IgG)的表观解离常数,并进一步测量最高亲和力变体的Fab片段的Kd值。
研究结果
研究团队通过上述方法,对七种人类免疫球蛋白G(IgG)抗体进行了优化,取得了显著成果:
-
MEDI8852:这种广泛中和的抗体结合甲型流感血凝素(HA),通过实验筛选,最佳变体的亲和力提高了七倍。
-
mAb114:这种埃博拉病毒抗体的最佳变体在结合埃博拉病毒糖蛋白(GP)时,亲和力提高了3.4倍。
-
REGN10987:对新冠病毒Beta变种刺突蛋白(Spike)的亲和力提高了1.3倍,对Omicron BA.1受体结合域(RBD)的亲和力提高了5.1倍。
-
S309:这种新冠病毒抗体的变体在不同病毒株上的亲和力均有提升,相较于目前的治疗性抗体sotrovimab,其最佳变体在与新冠病毒Wuhan-Hu-1 S-6P结合时,亲和力提高了1.3倍。
-
MEDI8852 UCA:这种抗体的最佳变体在与HA H1 Solomon结合时,亲和力提高了2.6倍,并且对某些HA亚型的结合亲和力也有显著提升。
-
mAb114 UCA:这种抗体在与埃博拉病毒GP结合时,最佳变体的亲和力提高了160倍。
-
C143:这种抗体的最佳变体在与新冠病毒Wuhan-Hu-1 Spike结合时,亲和力提高了高达23倍。
在所有抗体中,每种抗体只需要筛选20个或更少的变体,并且在两轮实验中就达到了显著的亲和力提升。
结论
本研究证明了利用蛋白质语言模型可以有效指导人类抗体的进化和优化,显著提高其结合亲和力。在本研究验证的7个抗体中,第一轮设计有14-71%的突变体提高了亲和力,第二轮设计绝大部分突变体都提高了亲和力。这种方法不仅在抗体优化中显示出巨大潜力,还可以推广到其他蛋白质家族的进化研究中,如抗生素抗性和酶活性等。
WeMol中集成了多种蛋白质语言模型
1. 通过序列生成,填充,氨基酸概率预测指导人源化,亲和力和稳定性改造
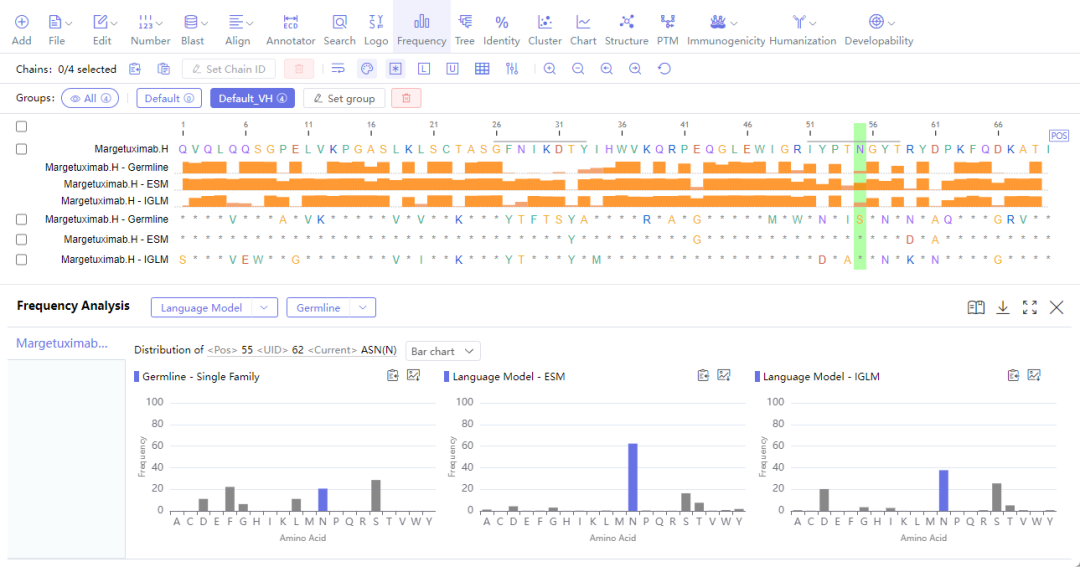
如上图,在WeMol中,利用蛋白质语言模型对整个序列的氨基酸概率进行分析,可以发现序列中出现的不常见的氨基酸。针对这些氨基酸进行突变可以帮助进行蛋白的人源化设计,亲和力改造,以及稳定性改造等多种应用。
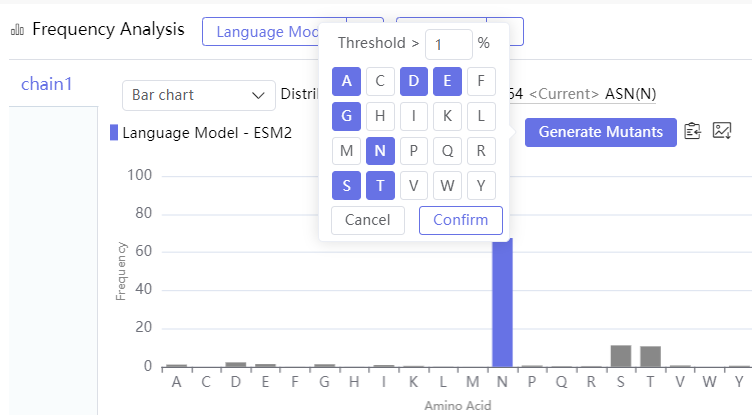
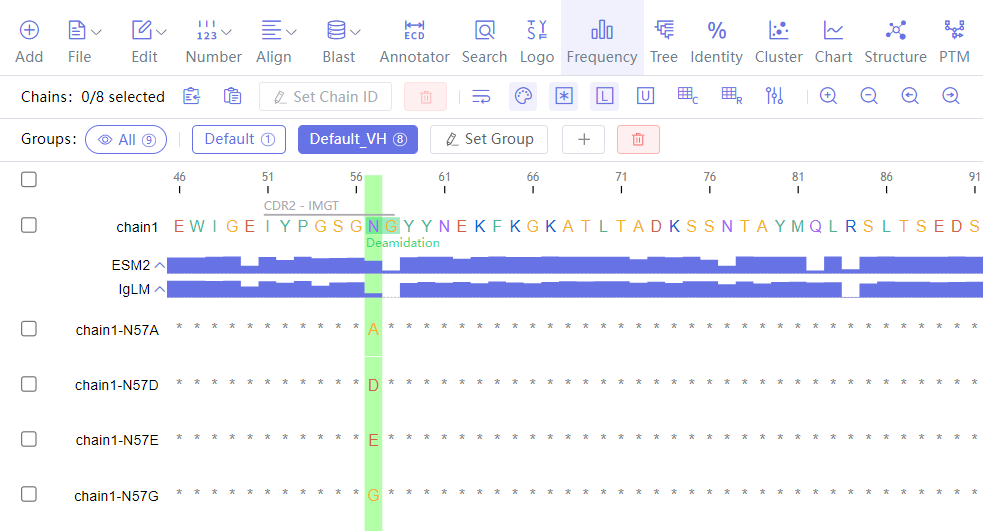
在下图针对抗体CDR区域的PTM位点改造的案例中,通过蛋白质语言模型预测该位点氨基酸的频率,(下图1)通过点击Generate Mutants按钮可以自动生成推荐的突变并显示到WeSeq的编辑器中(下图2)。
在WeMol中可以利用蛋白质自然语言模型设计的文库,针对CDR区域进行突变模拟,生成类内源抗体的大量抗体序列,在极大丰富文库的多样性的同时,优化可开发性和人源性等各方面的性质,使得筛选到的hit分子具备一步到位的成药潜力。
如果对蛋白质语言模型的其他应用或者方法开发有兴趣可以联系我们。
参考文献:
arXiv:2402.17156.
Bioinform Adv. 2022 Jun 17;2(1):vbac046.
Nat Biotechnol. 2024 Feb;42(2):275-283.
Wecomput(唯信计算)致力于”计算技术驱动创新药研发”,基于对创新药物研发流程的深刻理解,融合人工智能、生物物理、高性能计算等技术,打造了独具特色的自动化、智能化、数字化的药物分子生成、设计与模拟平台,有望革新传统药物发现方式,驱动小分子、蛋白质、抗体、mRNA等创新药物的研发进程。核心团队成员来自国际知名AI制药公司、头部药企、知名互联网公司、985高校,在制药、生命科学、人工智能、软件开发等交叉领域有丰富的经验、深刻的理解和饱满的热情。
母公司北京中大唯信科技有限公司于2015年注册成立,入选国家高新技术企业,2021年获红杉资本股权投资,并入选NVIDIA Inception计划资深会员。至今,Wecomput已服务海内外数百家客户,涵盖众多生物医药企业、高校、医院、科研机构,并倾力打造了自主知识产权的分子数字化智能计算平台WeMol。欲了解更多信息,请访问官网www.wecomput.com,或关注公众号“唯信wecomput”。
WeMol(wemol.wecomput.com)是Wecomput开发的面向生物制药、材料、化学等领域的新一代分子数字化智能计算平台,集成了计算生物学、人工智能、量子化学等领域的上百种Wecomput自研及开源的计算与可视化模块,核心算法的速度、准确度超过或媲美国外主流商业软件,尤其特色的抗体人源化设计、蛋白质免疫原性预测、虚拟亲和力成熟、高通量虚拟筛选、RNA序列设计等算法已在多家知名药企的数十个药物发现项目中得到验证和广泛应用。WeMol基于先进的流式架构,可将复杂计算流程简单化、自动化,并支持低代码定制开发和灵活扩展,是业界首款同时面向计算科学家及非计算专业的湿实验人员,旨在构建一个简单、易用、智能、可扩展、可追溯、可重现的一站式计算平台,全方位覆盖大分子生物药设计、小分子化合物设计、分子模拟、数据分析等应用场景,可对Hit->Lead->PCC各阶段进行全链条赋能。发布至今,WeMol已获得了国内外数百家药企及学术单位的青睐与好评。