

一个协同进化导向的蛋白-蛋白相互作用识别管道,将RoseTTAFold的速度和AlphaFold的精度结合,系统性地评估了830万对酵母蛋白之间的相互作用。
背景介绍
酵母双杂交(Y2H)、亲和纯化质谱(APMS)等高通量实验方法,已在酵母和其他生物中鉴定出许多对相互作用蛋白,但使用不同方法产生的数据集存在差异,且有相当高的假阳性率和假阴性率。由于蛋白-蛋白界面的残基预计会协同进化,所以任何两个蛋白相互作用的可能性可以通过pMSAs来评估,即识别和比对两个蛋白在许多不同物种中的同源序列并将其连接起来,创建成对的多重序列比对(pMSAs)。然后确定第一个蛋白的同源序列的变化与第二个蛋白的同源序列的变化协方差。氨基酸协同进化已经被用于指导伴侣蛋白结构已知的复合体的建模,并系统地识别在原核生物中相互作用的蛋白质对,比实验筛选的准确性更高。
最近深度学习在蛋白结构预测方面的进展有可能增强这类方法的能力,因为它们现在能够对蛋白质单体结构甚至蛋白-蛋白复合物进行精确建模。
主要内容
本文中,来自美国得克萨斯大学达拉斯分校的Qian Cong和 美国华盛顿大学的David Baker等人,利用蛋白质组内氨基酸协同进化分析和基于深度学习的结构建模的进展,系统地识别和建立了酿酒酵母蛋白质组内核心真核蛋白复合体的精确模型。研究者将RoseTTAFold和AlphaFold结合,对830万对酵母蛋白进行多序列比对筛选,识别出了1505对可能有相互作用的酵母蛋白,并为106个以前未识别的装配体和806个尚未描述结构特征的装配体建立了结构模型。这些复合体有多达5个亚基,在真核细胞几乎所有的关键过程中发挥作用,并提供了对生物功能的广泛见解。相关的研究成果以“Computed structures of core eukaryotic protein complexes”为题发布在国际顶级期刊Science上。
蛋白-蛋白相互作用和结构预测的准确性的评估
研究者着手将蛋白质组广泛的协同进化指导的蛋白质相互作用识别与基于深度学习的蛋白质结构建模相结合,以系统地识别和确定真核蛋白质组装体的结构(图1A)。
目前,主要存在以下挑战:
1. 首先,真核生物的基因组序列比原核生物少得多:细菌蛋白质的平均同源序列数在10,000左右,而真核生物蛋白质在1000左右。
2. 其次,真核生物通常有更多的基因,使得全面的成对分析计算量更大,背景噪声也更多。
3. 第三,真核生物的mRNA剪接进一步增加了蛋白质种类的数量,可能导致错误的基因预测和复杂的序列比对。
4. 第四,真核生物在多个谱系中经历了几轮基因组复制,很难从旁系同源中区分出直系同源,而这对检测协同进化的信号很重要,因为感兴趣的蛋白质相互作用可能保存在其他物种的直系同源中,但在旁系同源中保存的较少。
为了应对挑战,研究者以预测酿酒酵母的蛋白质复合体为起点,因为真菌基因组数量众多,基因组相对较小(共6000个基因),mRNA剪接相对较少。
对于大约6000个酵母蛋白中的4090个,研究者能够将一个单拷贝酵母蛋白分配给其他物种的直系同源蛋白,并生成了这些蛋白的4090×4089/2 = 8,362,005对组合的pMSAs。研究者专注于4,286,433对比对(包含超过200个序列)以提高预测精度,小于1300个氨基酸以加速计算。
在第一组计算中,研究者发现:即使有酿酒酵母和改进的直系同源识别的优势,之前在原核生物中使用的协同进化导向PPI筛选的统计方法(DCA)也无法有效地将768对已知相互作用的酵母蛋白对的金标准组与768000对基本不相互作用的酵母蛋白对进行区分(图1B灰色曲线,曲线下面积:0.016)。
因此,研究者探索了基于深度学习的结构预测方法RoseTTAFold (RF)和AlphaFold (AF)的应用。这种方法在区分金标准相互作用和随机对方面明显优于DCA(图1B蓝色曲线,曲线下面积:0.219),使用两个蛋白质中所有残基对的最高预测接触概率作为两个蛋白质相互作用倾向的度量。通过修正第一个蛋白质的C-端残基与第二个蛋白质的N-端残基之间的预测接触概率的过高预估,以及对一个显示与许多其他蛋白质具有枢纽状相互作用的蛋白质子集的预测相互作用,进一步提高了性能(图1B绿色曲线,曲线下面积:0.248)。
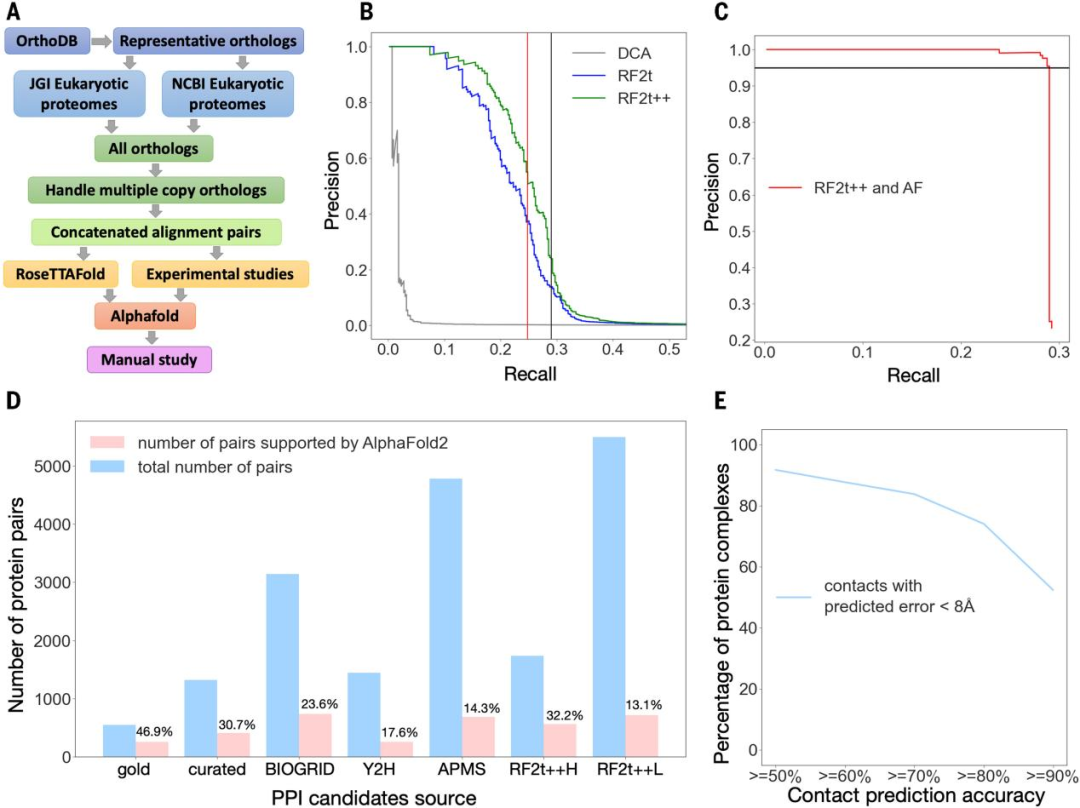
图 1. 蛋白-蛋白相互作用和结构预测的准确性的评估。图片来源于Science
研究者进一步研究了AF残基-残基接触预测,能否进一步区分相互作用和非相互作用的蛋白质对。AF太慢,无法应用到430万对pMSAs中(这将需要10万到100万图形处理单元(GPU)小时);相反,AF可以应用于具有最高RF支持的5495个蛋白质对(如图1B中的黑色竖线)。使用所有残留物对中最高的AF接触概率作为相互作用强度的测量标准,研究者发现RF和AF的组合提供了优异的性能(图1C)。几乎所有的金标准对的排序都高于阴性对照,选择的一组715个候选PPIs在AF接触概率的截止点为0.67(图1C中的黑色水平线),预期精度为95%。
鉴于AF在区分RF滤波对之间的金标准相互作用方面的优异性能,作者还将AF应用于pMSAs,PPIs来自于在文献中报道的PPIs(包括在高通量实验筛选中识别的)。研究者发现47%的黄金标准PPIs是预测可信的,根据BIOGRID的低通量实验,候选PPIs的比例较低(31和24%)。
最终,研究者从“从头RF→AF”流程中共鉴定出715对可能的相互作用对,从“混合实验集→AF”流程中共鉴定出1251对,去掉重合的共鉴定出1505个PPIs。其中699个已经进行了结构表征,700个从文献和数据库中获得了一些实验支持的数据,据目前所知,剩下的106个此前还没有实验描述过。为了评估预测的蛋白质复合体三维结构的准确性,研究者将PDB中的699对实验结构作为基准。对于92%的蛋白对,至少有50%(预测对齐误差<8 Å) AF-预测接触出现在实验结构中(图1E)。
每个功能类复合体的范例
基于以上基准结果,研究者对新的复杂交互预测和预测复合体的3D模型的准确性有了初步的信心。接下来对806个复杂复合体的结构模型进行了分析,这些复杂复合体的高分辨率结构信息目前尚无法获得。研究者根据它们的生物功能,将这些模型进行了分组,并提供了每个功能复合体的范例。
1. 第一组复合体涉及遗传信息的维护和处理:DNA修复、有丝分裂和减数分裂检查点、转录和翻译。
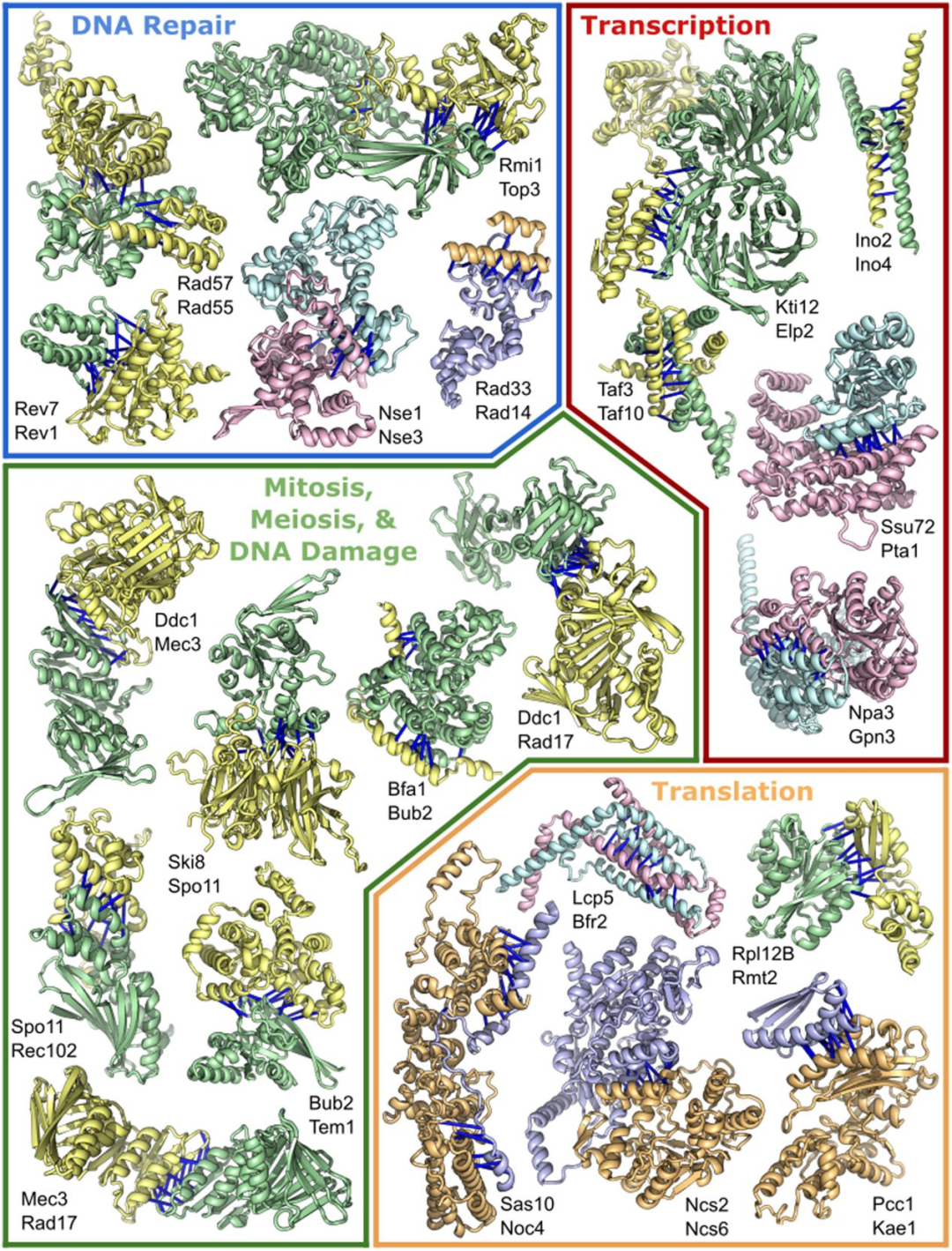
图 2. 参与转录、翻译和DNA修复的蛋白质复合体。图片来源于Science
2. 第二组复合体在蛋白质转运、通过分泌途径转运、细胞骨架和细胞器中发挥作用。
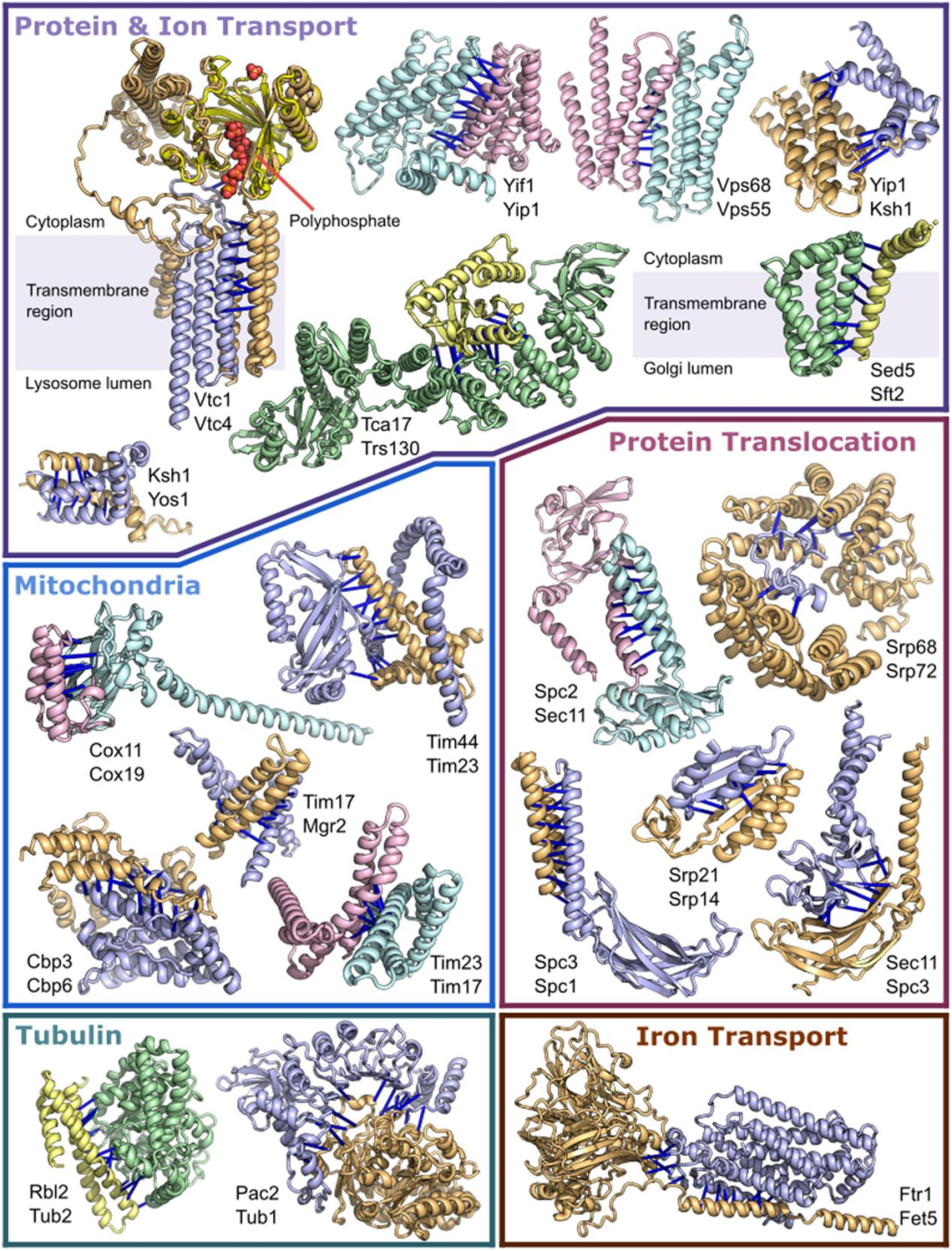
图 3. 参与分子转运、膜转运和线粒体的蛋白质复合体。图片来源于Science
3. 第三组复合体参与新陈代谢。图4显示了功能未知的蛋白与特征良好的蛋白相互作用的蛋白-蛋白复合体的范例:这些相互作用为未表征蛋白的功能提供了线索,并有助于识别先前表征的组装体的新组件。
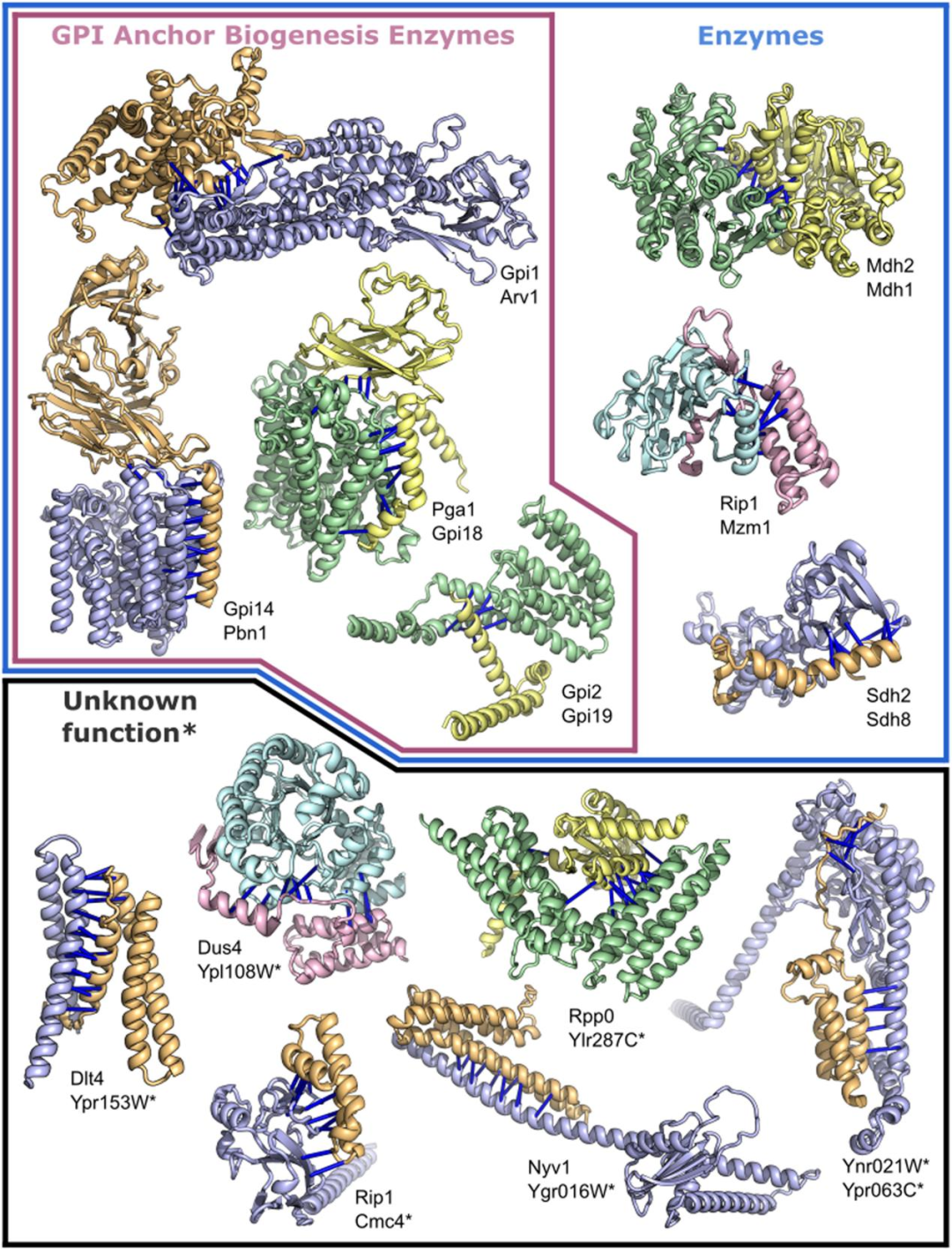
图 4. 参与代谢、GPI(糖基磷脂酰肌醇)锚定生物合成的蛋白复合体,或包括功能未知的蛋白质。图片来源于Science
在预测三个或更多蛋白质相互作用的情况下,研究者通过使用整个复合体的序列比对作为输入,生成了完整组装的模型。这些大型装配的示例如图5所示;在大多数情况下,成对的相互作用与独立构建的二元复合体非常相似,但是整个复合体的同时建模的优点是允许伴随完整组装的构象变化。
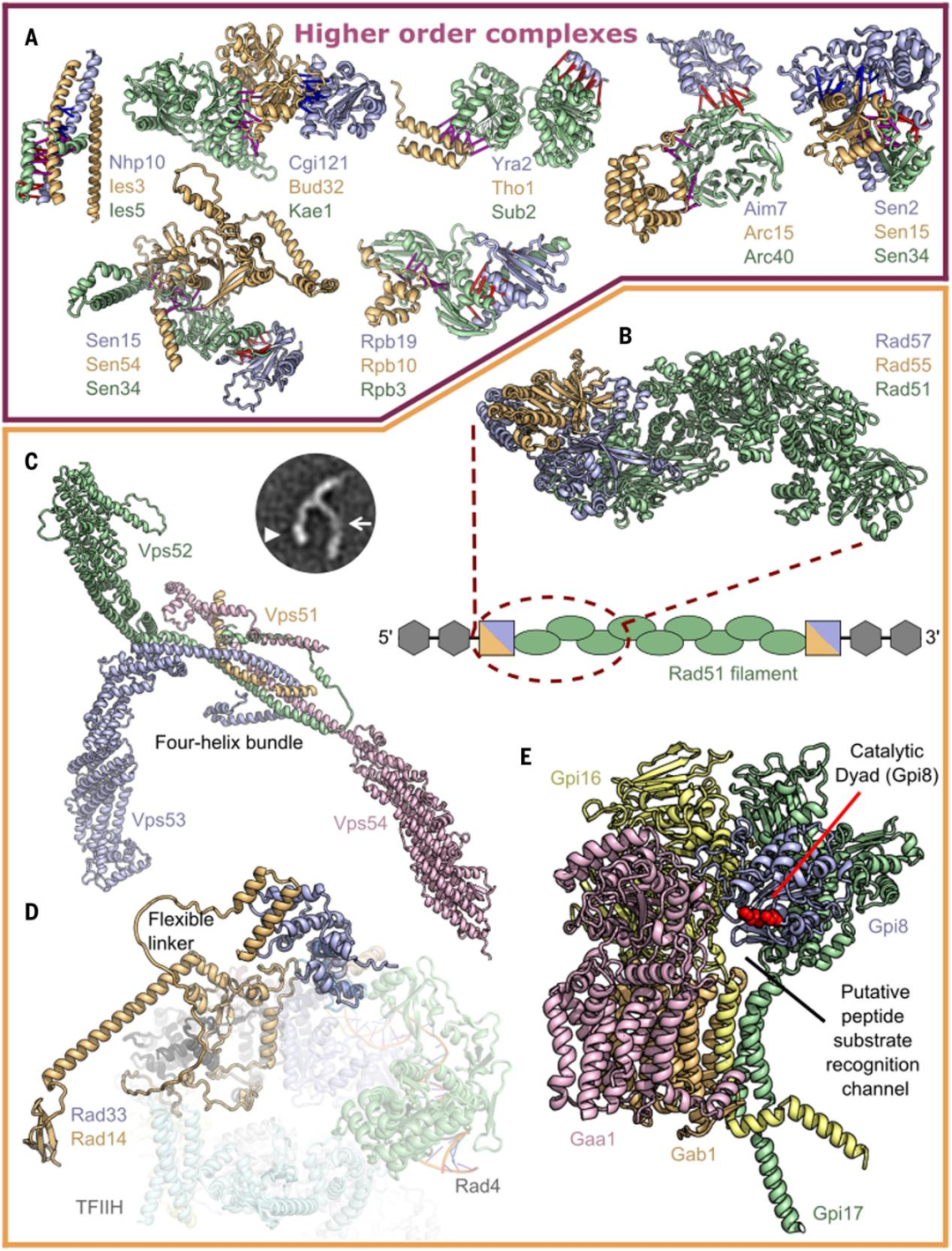
图 5. 高阶蛋白复合体。图片来源于Science
结论总结
本文提出的方法将基于深度学习的大规模结构建模的范围从单体蛋白质扩展到了蛋白质组装。正如上面的范例所强调的,对这里提出的许多新复合体的跟踪,将促进对广泛的真核细胞过程的理解,并为治疗干预提供新的靶点。
在蛋白质复合体上训练RF和AF,可以进一步提高这两种方法的性能,特别是对于同源性较少和/或相互作用较弱和较短暂的蛋白质对,并减少对同源性鉴定的依赖。结合单体结构预测的进展,该研究结果预示着一个新的结构生物学时代的到来,在这个时代中,计算在相互作用发现和结构确定中起着基础性的作用。
参考文献
Humphreys, Ian R., et al. “Computed structures of core eukaryotic protein complexes.” Science 374, eabm4805 (2021). DOI: 10.1126/science.abm4805