

一种通用且可配置的深度学习框架,可利用3D卷积神经网络进行蛋白质-蛋白质界面(PPIs)的数据挖掘。
背景介绍
高度调控的蛋白-蛋白相互作用网络协调了从DNA复制到病毒入侵和免疫防御等大多数细胞过程,蛋白以特定的方式进行相互作用或与其他生物分子相互作用。获得这些生物分子在3D空间中如何相互作用的知识是理解其功能和开发或设计这些分子用于各种靶标的关键,如药物设计、免疫治疗或设计新的蛋白等。
在过去的几十年里,各种实验方法(如X射线晶体学、核磁共振、低温电子显微镜)已经确定并积累了大量蛋白-蛋白复合物的原子分辨率3D结构。截至2020年9月2日,PDBe数据库(https://www.ebi.ac.uk/pdbe/)中已有7000个非冗余结构。许多机器学习方法已经发展到可以从这些实验3D结构中学习复杂的相互作用模式。与其他机器学习技术不同,深度神经网络有望在非迅速达到性能稳定的情况下,从数百万数据中学习,这是通过获取硬件加速器(如GPU、TPU)和并行文件系统技术进行计算处理实现的。
以图形表示蛋白界面的图形神经网络(GNN)也已被应用于蛋白界面的预测。深度神经网络在结构生物学中的应用有一个著名的案例,即AlphaFold2,这是在最新一期CASP14中在单链蛋白结构预测方面的最新突破。然而,预测蛋白复合物的3D结构仍然是一大挑战:在CASP14中,除非有已知的模板,否则没有一个单独的组装能被正确预测。这就需要开源的框架,因为它可以很容易地被社区修改和扩展以用于数据挖掘蛋白质复合物,并可以加速相关科学知识的发现。
挖掘3D蛋白复合物数据目前存在几个独特的挑战。首先,蛋白界面受物理化学规则的控制,不同类型的蛋白复合物(如酶-底物、抗体-抗原)可能具有不同的显性相互作用特征;其次,蛋白质相互作用可以在不同的水平上进行表征:原子-原子水平、残基-残基水平和二级结构水平;第三,蛋白质界面,在形状、大小和表面曲率方面具有高度多样性;最后,蛋白质的大量原子坐标文件的高效处理和特征化,在计算成本和文件存储需求方面是令人生畏的。
主要内容
荷兰乌德勒支大学的Alexandre M. J. J. Bonvin 和Li C. Xue等研究者开发了DeepRank,一种基于3D CNNs的蛋白质-蛋白质界面数据挖掘的通用深度学习平台。DeepRank将PDB中生物分子复合物的三维原子坐标计算出的原子和残留级特征映射到三维网格上。DeepRank允许使用包含数百万PPIs的数据集,高效地训练3D CNNs,并支持分类和回归。研究者展示了DeepRank在两个不同挑战上的表现:生物与晶体学PPIs的分类,以及对接模型的排序。在这两个问题上DeepRank都是具有竞争力的,甚至优于最先进的方法。这证明了其在结构生物学研究框架的多功能性。相关的研究成果以“DeepRank: a deep learning framework for data mining 3D protein-protein interfaces”为题发布在国际著名期刊Nature Communications上。
python包地址
https://pypi.org/project/deeprank/
源代码地址
https://github.com/DeepRank/deeprank
DeepRank概述
DeepRank允许对3D蛋白-蛋白复合物的数据集进行端到端训练。框架由两个主要部分组成,一个是数据预处理和特征化,另一个是神经网络的训练、评估和测试。特征化利用MPI并行化和GPU卸载,来确保在非常大的数据集上的高效计算。
数据预处理和特征化:
(1) 特性计算。从描述蛋白-蛋白复合物的3D结构的PDB文件开始,DeepRank利用pdb2sql来识别两个链之间的界面残基;默认情况下,界面残基定义为那些与任何原子在5.5 Å距离截止(可配置)的其他链的任何原子(图1A)。表1中给出的原子和基于残差的特性,在默认情况下是计算出来的,但是用户可以轻松定义新的特性,并将这些新的特性包含在他们的特性计算工作流中。
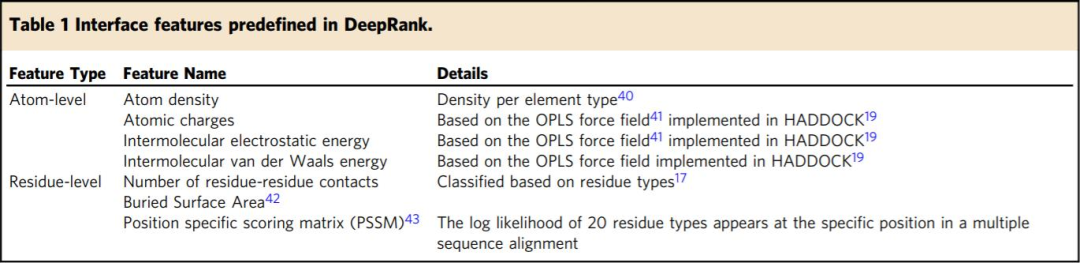
表 1. DeepRank中预定义的界面特性。表格来源于Nat. Commun.
(2) 三维网格特征映射。DeepRank利用高斯映射,将复合体界面的原子和残基特征映射到三维网格上。图1A说明了基于残基特征的映射过程。由于这种高斯映射,每个特征在三维特征网格上都具有非局部效应,从而形成大量的网格点。PPIs的这种特征映射产生了一个3D图像,其中每个网格点包含多个通道值,对应于界面的不同属性。多种数据增强和PPIs结构对齐策略可以丰富数据集。
(3) 灵活的靶标值定义和计算。用户可以很容易地为自己的蛋白结构定义特定问题的靶标值。在计算对接场景下,评估对接模型质量的标准指标是通过与参考结构的比较得到的,CAPRI(预测交互的关键评估)中使用的一些指标已集成到DeepRank中。比如配体RMSD、界面RMSD (iRMSD)、FNAT(原生接触分数)、CAPRI质量标签和DockQ评分。DeepRank能利用pdb2sql高效地执行这些计算。
(4) 高效的HDF5格式数据存储。处理数千万个具有丰富特征表示的PDB文件,对文件系统和深度神经网络的有效训练都是一个挑战。DeepRank以HDF5格式存储特征网格(图1B),特别适合存储非常大的异构数据集。
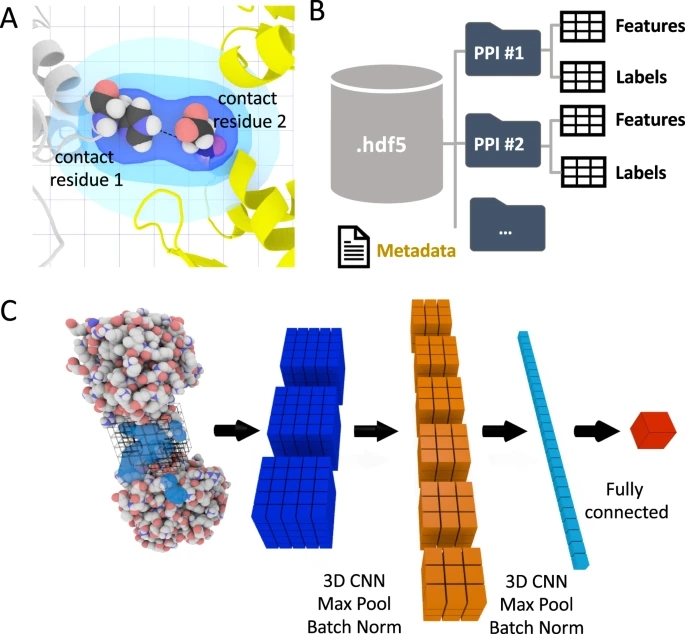
图 1. DeepRank框架。图片来源于Nat. Commun.
应用1:检测晶体构造
X射线晶体学,是测定蛋白质复合物三维结构最重要的实验方法之一(它占所有PDB已存条目的80%)。当涉及到复合物的结构时,产生的晶体通常包含多个界面,其中一些与生物学相关,而另一些仅仅是结晶过程的产物,即所谓的“晶体界面”(图2A, B)。在没有额外信息的情况下,区分晶体界面和生物界面仍然具有挑战性。
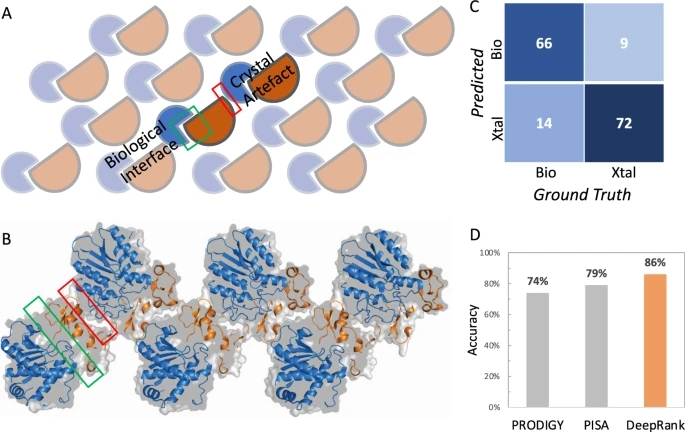
图2. 利用DeepRank对生物界面和晶体界面进行分类。图片来源于Nat. Commun.
在由2828个生物界面和2911个晶体界面组成的MANY数据集上,研究者只使用了Position Specific Scoring Matrix (PSSM)特征对指定的3D CNN进行了训练和验证。每个结构在训练前先用随机旋转(30次)增强。研究者采用验证损失的早停法来确定最优模型。训练后的网络,在包含80个生物界面和81个晶体界面的DC数据集上进行了测试。测试得知,经过训练的网络在80个生物界面中正确分类了66个,在81个晶体界面中正确分类了72个(图2C)。DeepRank的准确率达到86%,优于PRODIGY-crystal和PISA,后者分别为74%和79%(图2D)。
应用2:排序对接模型
对于单个蛋白的三维结构,蛋白界面通常是通过生成数以万计的候选构象(模型)来构建它们的相互作用模式,然后使用评分函数对这些模型进行排序,以选择正确的(接近原生的)模型(图3A)。
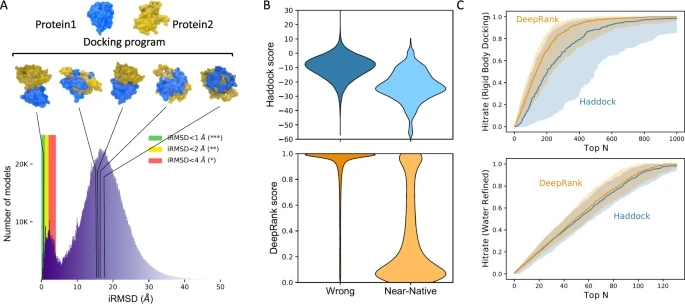
图3. DeepRank应用于对接评分问题。图片来源于Nat. Commun.
研究者使用HADDOCK为对接基准v5 (BM5)集生成了一套不同质量的对接模型,包括刚性对接、柔性对接和最终的精准对接模型。在这项工作中,研究者关注142个二聚体,这些二聚体在生成的数据集中具有接近原生模型的可用性,在生成的数据集中,排除了所有的抗体-抗原复合物。
DeepRank在刚性对接阶段生成的HADDOCK模型上表现良好。DeepRank的评分能够很好地区分接近原生的模型和错误的模型(图3B),而HADDOCK的评分在这两个类别之间呈现出显著的重叠。在排序性能方面,DeepRank通过在Top N名中选择更接近原生的模型,在刚性对接模型上优于HADDOCK(图3C顶部)。
DeepRank和HADDOCK在水-精准对接模型中的差异并不明显(图3C底部)。然而,请注意,HADDOCK要求对刚性、柔性对接和水-精准阶段生成的模型使用不同的评分函数,而DeepRank对所有阶段使用相同的评分函数。对DeepRank和HADDOCK在不同阶段的成功率进行了分析,证实了DeepRank在每种类型模型上的表现都略好于HADDOCK。这再次证实了DeepRank评分的稳健性,因为它提供的单一评分在不同的模型中都表现良好。
DeepRank的优点
1. 从用户的角度来看,该平台界面对用户十分友好。它实现了许多可以轻松调优的选项。特征化和神经网络架构的设计提供了灵活性。这使得它直接适用于一系列使用蛋白-蛋白界面作为输入信息的问题。
2. 从开发人员的角度来看,DeepRank是一个软件包,它遵循软件开发标准,包括版本控制、持续集成、文档和易于添加的新特性。这种灵活性增加了可维护性和社区对DeepRank的进一步开发,例如,允许预测单个蛋白质结构上的突变效应。
3. 计算效率:DeepRank的开发使得利用数百万PDB文件来训练模型和测试其性能成为可能。
4. 最后,在两个不同的研究问题上,它们的表现与最先进的技术相媲美,证明了DeepRank在一般结构生物学中的多功能性。
总结
本文描述了一个开源的、通用的、可扩展的深度学习框架,可用于对海量的蛋白-蛋白界面数据集进行数据挖掘。研究者证明了在结构生物学的两个不同挑战中应用DeepRank的有效性和敏捷性。DeepRank有望能够通过简化繁琐的数据预处理步骤,降低大规模数据分析可能带来的令人生畏的计算成本,从而加快与蛋白界面相关的科学研究。
DeepRank的模块化和可扩展的框架具有极大的潜力,可促进计算结构生物学社区在其他蛋白质结构相关主题上的协作开发,并将有助于在结构生物学研究中应用和发展深度学习技术。
参考文献
Renaud, N., Geng, C., Georgievska, S. et al. DeepRank: a deep learning framework for data mining 3D protein-protein interfaces. Nat Commun 12, 7068 (2021). https://doi.org/10.1038/s41467-021-27396-0