

一种基于几何结构软件和深度学习的新框架DeepPoc一套挖掘苗头片段周围的化学空间的流程,它可以找到结合模式相似且新颖的类似物。在对BRD4案例的测试中,该流程发现了新颖的活性分子,IC50 为 26 μM。ket,用3D卷积神经网络对Fpocket识别的口袋重新评分后能发现蛋白表面的空腔。
背景介绍
基于片段的药物设计已成为发现新候选药物的常用手段。优化片段的方法通常基于苗头分子,对初始片段的骨架小做修改。实际上,苗头片段周围的化学空间可以提供新的类似分子并具有更高的活性概率。这种探索片段空间的方式可以发现次优的苗头片段,揭示更传统的片段优化过程中被忽视的未探索途径。
巴塞罗那大学的Xavier Barril团队设计了如下片段挖掘流程:(1) 对化学库可扩展空间的探索;(2) 骨架跃迁探索苗头片段周围的多样性;(3) 利用结构信息增加活性分子概率;(4) 迭代逐步生长片段,提高配体效率增加效力;(5) 自动化减少人工干预。作者将该流程用在了含溴结构域的蛋白质 4(BRD4)并找到了具有新颖性的骨架。
主要内容
片段挖掘流程
如图 1 所示。该流程从已知结合模式的初始片段开始,保留基本相互作用以增加选择活性化合物的机会;接着对ZINC15子集做相似性搜索。搜索仅限于与参考配体大小相似的分子,每次迭代最多添加两个重原子。该约束保证了配体效率,并避免偏向于搜索更大分子,这种搜索方式可以使分子结构更丰富并获得更好的绝对分数。
作者将检索到的分子按以下两步过程叠加到查询分子上,确保关键的相互作用和结合模式:(i) 用原始片段识别最大公共子结构 (MCS),其中必须包括关键相互作用点;(ii) MCS 上做原子的叠加,然后进行对接,其中 MCS 受到限制,只有 MCS 外的可旋转键被对接算法采样,这样做是为了优化分子间相互作用的同时仍保留参考配体的结合模式,最终从中选择得分最高且相似度超过90%的 500 个分子。接下来再用dynamic undocking (DUck) 过滤掉对接不稳定的分子,最后选择得分最高的 50 个分子。
初始苗头片段周围的化学空间的代表性样本数量跟数据库的分子相比不值一提,在一轮迭代筛选过程后,使用筛选出的50个分子重新作为迭代起点。如果对这些分子中的每一个都进行相似性搜索,筛选出的分子数将会呈现指数性增长。为保持计算效率,所有相似苗头片段均按共同亲本的数量排序,并且仅考虑排名靠前的10万个分子,防止搜索范围过度分散。
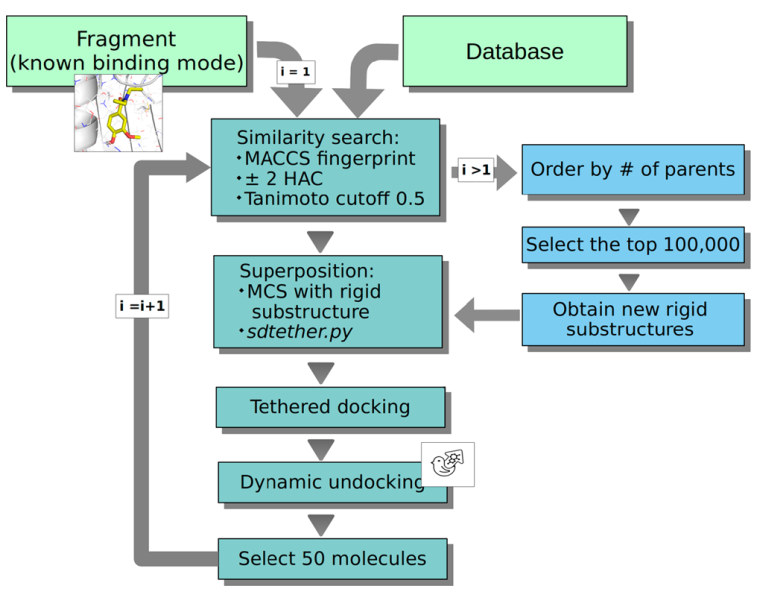
图1. 片段挖掘平台流程图。图片来源:JMC
以BRD4为例展示如何使用流程找到新颖骨架
最初选取测试片段1XA(PDB:4LR6)来研究流程是否可以从已知类型中识别出新物质。异恶唑环在BRD4抑制剂中很常见,其杂原子与 Asn140 的侧链氮原子形成氢键,剩余的环可以进入空腔底部、空腔远端和溶剂。
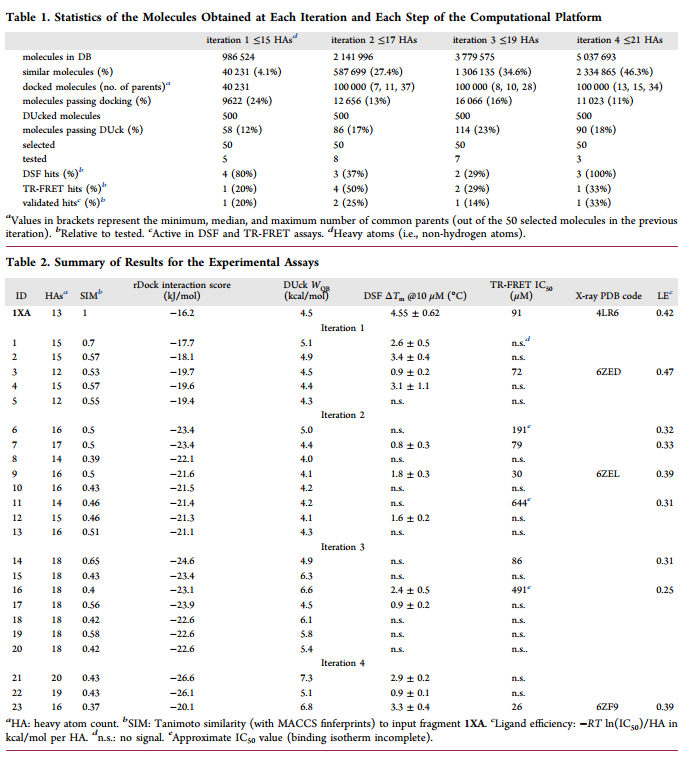
1XA 与抑制剂 (+)-JQ1 合并共同用于测试并迭代了4 次流程。在每次迭代结束时选择50 个分子。每次迭代时参数如表1所示。为了验证计算预测,测试分子样本结果如表 2所示。作者应用了单一浓度的差示扫描荧光法 (DSF) 和通过 TR-FRET 的底物置换试验测得 IC50 值。52% 的测试分子在 DSF 中给出阳性信号,35% 在 TR-FRET 测定中呈阳性。22% 在两种测定中都是阳性的分子被认为是有效的苗头片段。其中有三个片段通过 X 射线晶体学确定了它们的结合模式。
表1. 每轮迭代得到的分子信息统计;表2. 生物测试的结果。表格来源:JMC
首轮迭代
由于 1XA 有 13 个重原子 (HA),该平台筛选了重原子数在11-15的近 100 万个分子,其中只有 4% 进入下一步。叠加 MCS 和对接后,9622 个分子 (24%) 的对接相互作用评分不差于1XA (-16.2kJ/mol)。作者以 4 kcal/mol作为阈值,从55500 个分子中找出了58个分子,从供应商购买了骨架不同的5个分子。值得注意的是,3 作为母体片段 1XA (IC50 = 91μM) 是无活性的 (IC50 = 72 μM),在LE方面略好(0.47 vs0.42 kcal/mol/HA)。结合模式如图 2所示。化合物 3 保留了两个与 Asn140 相互作用的氢键受体,异恶唑被换成哒嗪;1XA 的伯胺变成吡唑环。较宽的相似性搜索要求下可能出现这种激进的骨架变化,严格的对接和 DUckfilters 可确保基本的蛋白片段相互作用。
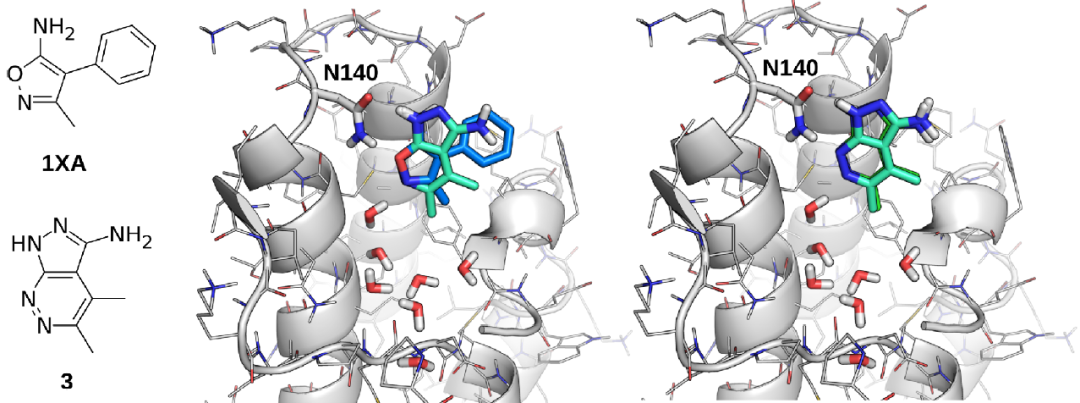
图2. BRD4与化合物3复合物的X射线结构
后续迭代
作者还前瞻性地评估了流程逐步增加虚拟苗头片段的功能。一般情况下,研究人员希望在每个周期后安排实验测试,识别活性分子并剔除非活性分子,这么做不切实际。但是多次迭代而不筛选可能产生假阳性分子,存在错误的分子迭代和效果下降的风险。迭代的分子也存在不可购买的难题(表 2 和图 3),最后一次迭代收敛到具有常见 5-苯基噻唑并[2,3-c][1,2,4]-三唑骨架的分子,但是这些分子都无法购买。因此,作者合成了代表性分子23,总共测试了 18 个分子。其中,8 个 (44%) 被 DSF 检测为有活性,7 个 (39%) 被 TR-FRET 检测到。命中率与第一次迭代获得的结果一致,证实进化过程没有偏离到假阳性。两种最有效的分子9(IC50 = 30 μM)和23 (IC50 = 26 μM),通过 X 射线晶体学确定了结构(图 4;PDB:6ZEL 和 6ZF9),与预测的结合模式一致。23与 3 一样,它代表了不剧烈的骨架跃迁,LE = 0.39,是全新的BRD4 配体。
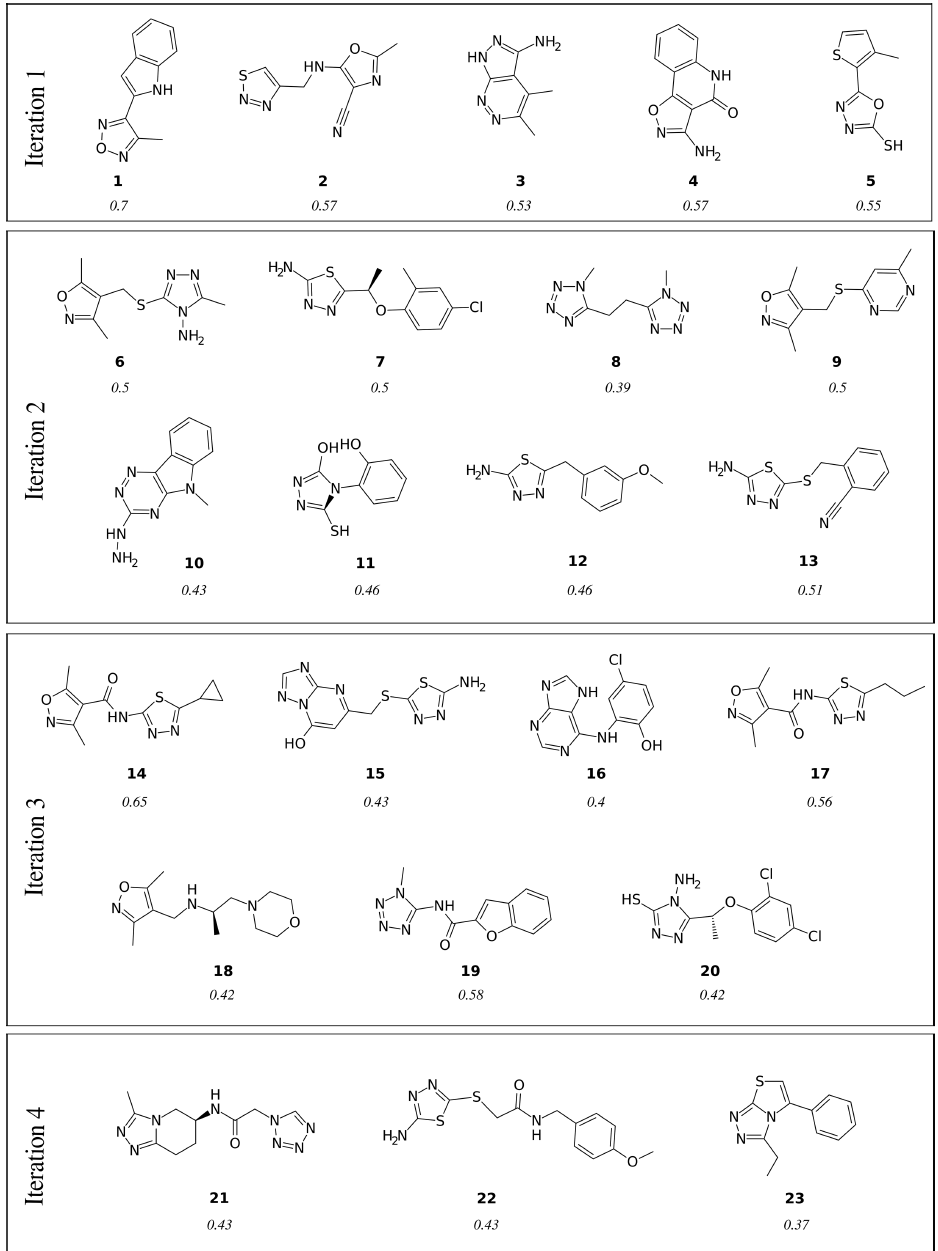
图3. 流程发现并测试活性的分子。图片来源:JMC
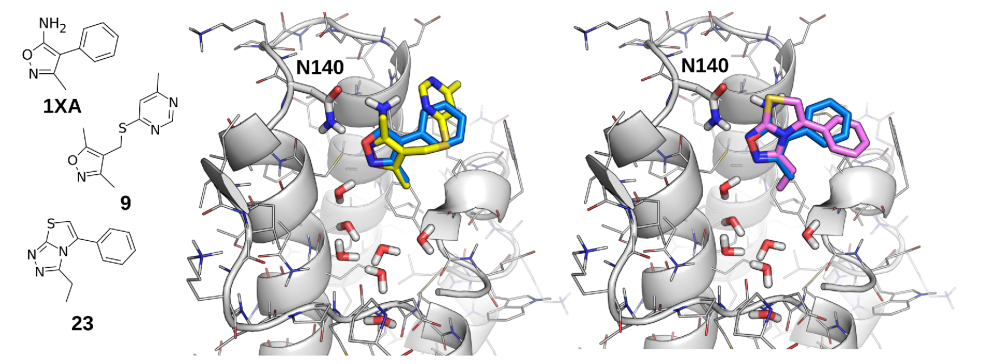
图4. BRD4与化合物9复合物,23复合物的X射线结构
骨架分析
流程的特征之一是专注于苗头片段所在空间,识别出非明显的类似物。由于描述骨架的方式不同,作者尝试了化学骨架和拓扑骨架两个角度。对于第一类,作者使用 Bemis 和 Murcko 的化学骨架算法。对于第二类,作者使用键通用支架,其中仅保留环原子并删除元素和杂交信息。
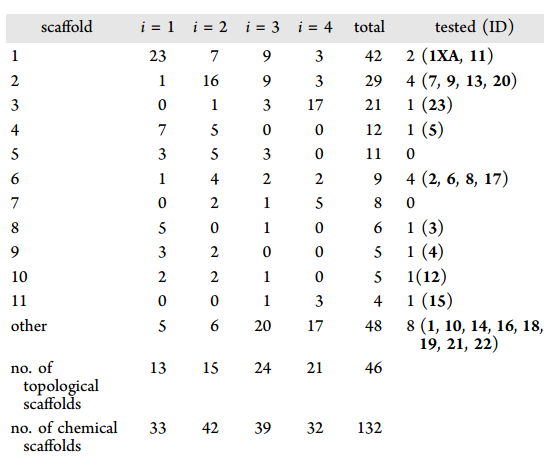
在第一次迭代中,1XA的原始骨架代表了 50 个选定分子中的 7 个分子。当作者考虑拓扑骨架时则代表了23个分子(图 5)。在这个阶段,50 个分子代表了 33 种不同的化学支架和 13 种拓扑支架(图 6;表 3)。14 个分子(28%)呈现出适度的拓扑骨架转换,剩余的 13 个分子呈现出与原始分子差异更大的拓扑支架。该分析证实,大部分所选分子在原始片段的传统搜索中无法检索到,连续的迭代能使搜索远离化学空间。第一次迭代中两个更频繁的拓扑支架(表 3 中的数字 1 和 4)保留在第二次迭代中,但频率降低(从 60% 到 24%)。在本次迭代中,中等支架变化的分子数从 13 个增加到 15 个,算法开始实质性的拓扑修改。在第三次迭代中,趋势加速,这是探索性的步骤,有 24 个拓扑支架和 39 个化学支架,骨架分散到了无关的分子中。流程只考虑那些具有大量共同亲本的分子,遏制分散的趋势以降低独特骨架的数量。但是此时流程已经找出了一个优势骨架。23 和3具有完全新颖的化学和拓扑支架。因此,尽管已经知道大量 BRD4 抑制剂,但流程能基于苗头片段的周围化学空间中发现全新的 BRD4 支架。
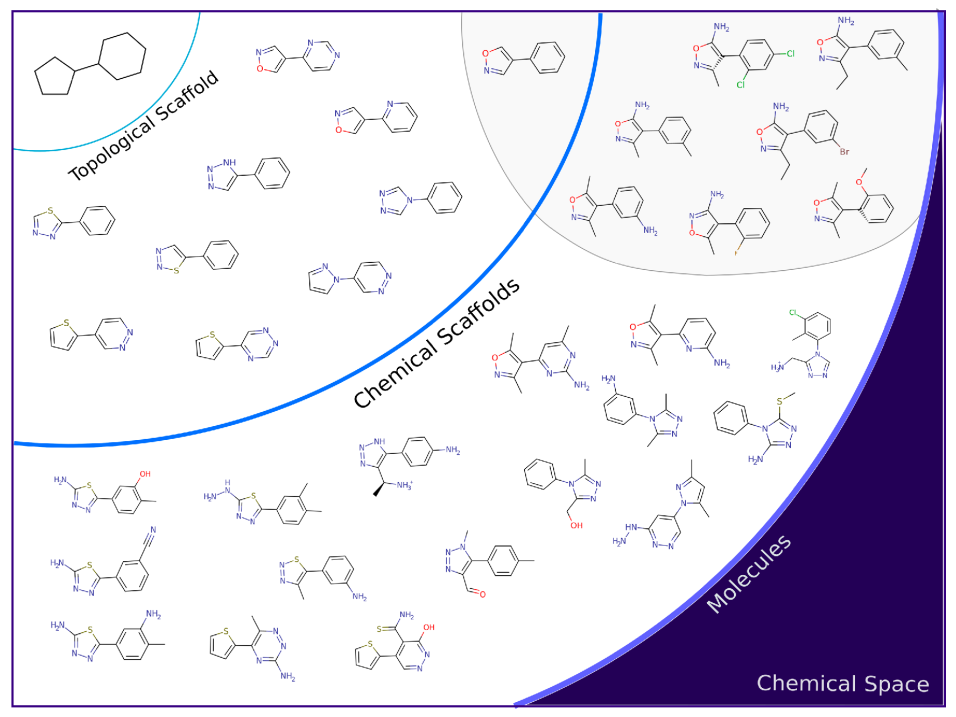
图5. 第一轮迭代的苗头化合物的骨架多样性。图片来源:JMC
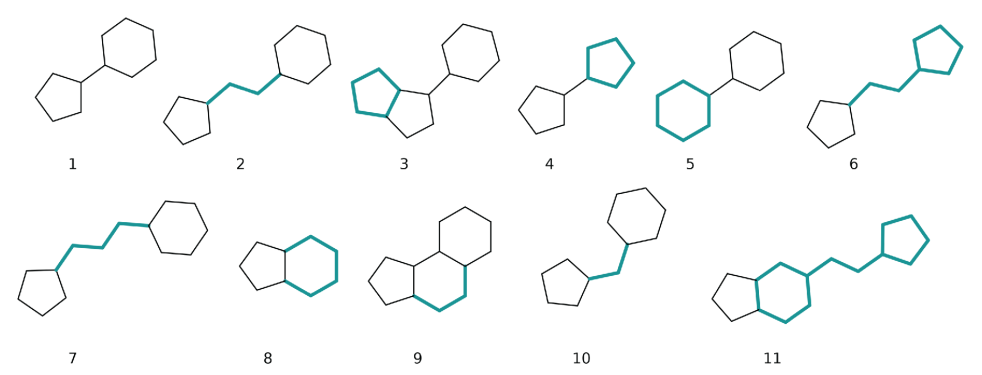
图6. 主流的拓扑骨架。图片来源:JMC
表3. 四轮迭代后,在200个苗头分子中,出现频数大于4次的骨架统计信息。表格来源:JMC
总结
本文介绍了一种计算平台,可以从特定苗头片段周围的化学空间中,寻找出结构多样的可能遗漏的片段类似物。它还可以迭代使用,逐渐偏向于具有更高分数分子的化学空间区域。此过程可产生高命中率,并且收敛于优势骨架。但应该注意的是,该平台不能用于片段生长。为了进行深入搜索更有效的类似物,需要限制化学空间探索的广度。
参考文献
Serena G. Piticchio, Míriam Martínez-Cartró, Salvatore Scaffidi, Moira Rachman, Sergio Rodriguez-Arevalo, Ainoa Sanchez-Arfelis, Carmen Escolano, Sarah Picaud, Tobias Krojer, Panagis Filippakopoulos, Frank von Delft, Carles Galdeano, and Xavier Barril. Discovery of Novel BRD4 Ligand Scaffolds by Automated Navigation of the Fragment Chemical Space, Journal of Medicinal Chemistry, 2021, 64(24), 17887-17900.