

华中科技大学宁康教授团队基于地球上4个主要生物群落(肠道、湖泊、土壤和发酵罐)的42.5亿个微生物群落序列构建了AI模型MetaSource,预测出了1,044个未知结构蛋白的可靠结构。
背景介绍
目前,蛋白三维结构预测主要有两种策略:一种是基于模板的建模,以已解决的结构为模板构建结构模型,需要在PDB中有同源模板;第二种是无模板建模(FM)方法(或从头开始建模),专注于对PDB中没有密切同源结构的硬蛋白进行建模。最有效的FM方法,包括Rosetta、QUARK和I-TASSER,以及最近的AlphaFold和trRosetta,依赖于先验空间约束,通过深度神经网络学习,从基于同源序列的多序列比对(MSAs)的共进化信息中获得。因此,要对“硬”蛋白进行三维结构建模,需要有足够数量的同源序列,以保证深度机器学习模型的准确性和后续三维结构构建的质量。
近年来,人们在利用宏基因组序列数据来增强MSA和FM模型的构建上付出了大量努力。最近,Wang等人研究了Tara Oceans微生物基因组的有效性,发现微生物基因组可以为高质量的MSA构建以及蛋白结构和功能建模提供额外的帮助。这一结果表明,微生物组序列在FM结构折叠和基于结构的功能注释中发挥了重要作用,这些序列代表着地球上最大的微生物物种之一。
尽管宏基因组辅助的三维结构建模取得了成功,但仍有成千上万的Pfam家族的建模结构不能令人满意。一个关键的原因是,尽管序列积累迅速,但目前的序列数据库还很不完整,许多FM目标的同源序列非常少。另一方面,微生物组数据库已变得非常庞大,这使得全面且平衡的数据库搜索变得越来越缓慢和困难。因此,一种具有精确MSA构造的均衡序列挖掘方法对于提高序列数据库搜索效率和后续三维结构建模具有至关重要的意义。
主要内容
华中科技大学生命科学与技术学院宁康教授团队联合美国密歇根大学计算医学与生物信息系张阳教授团队,假设在微生物生态位(生物群落)和蛋白质家族之间存在着一种内在的进化联系,依此联系建立一种有针对性的方法,可以提高MSA构建和蛋白质结构预测的效率和准确性。为了检验这一假设,研究者从EBI宏基因组数据库(MGnify)中收集了一个包含42.5亿个微生物组序列的模型库,该数据库覆盖了4个主要的生物群落。“边际效应”分析表明,特定生物群落对不同Pfam家族同源序列的补充作用存在显著差异。随后,研究者开发了一个名为MetaSource的机器学习模型来预测靶蛋白的源生物群落,该模型可显著提高接触图和三维结构模型的精度,同时节省了超过三倍的计算机内存和CPU时间。相关的研究成果以 “Decoding the link of microbiome niches with homologous sequences enables accurately targeted protein structure prediction” 为题发布在国际著名期刊Proceedings of the National Academy of Sciences of the United States of America上。
C-I-TASSER模型、Pfam家族结构未知的MSAs以及基准数据集下载地址
https://github.com/HUST-NingKang-Lab MetaSource /releases
微生物群落样本包含来自数千个属的数十亿个不同功能基因
研究者从4个典型的微生物生态位(图1A)中,采集了1705个微生物组样本。从这些生物群落中共预测了42.5亿个蛋白质序列(功能基因),鉴定出169个门8721个属,图1B-D展示了生物群落特异性的分类剖面。这些结果表明,微生物群落中存在着不同的微生物群系,进一步的研究揭示了微生物群落分类剖面与其活菌群之间的相关性:在肠道菌群中,如厚壁菌门(平均相对丰度:0.41±0.28)和拟杆菌门(平均相对丰度:0.26±0.14)为优势门。
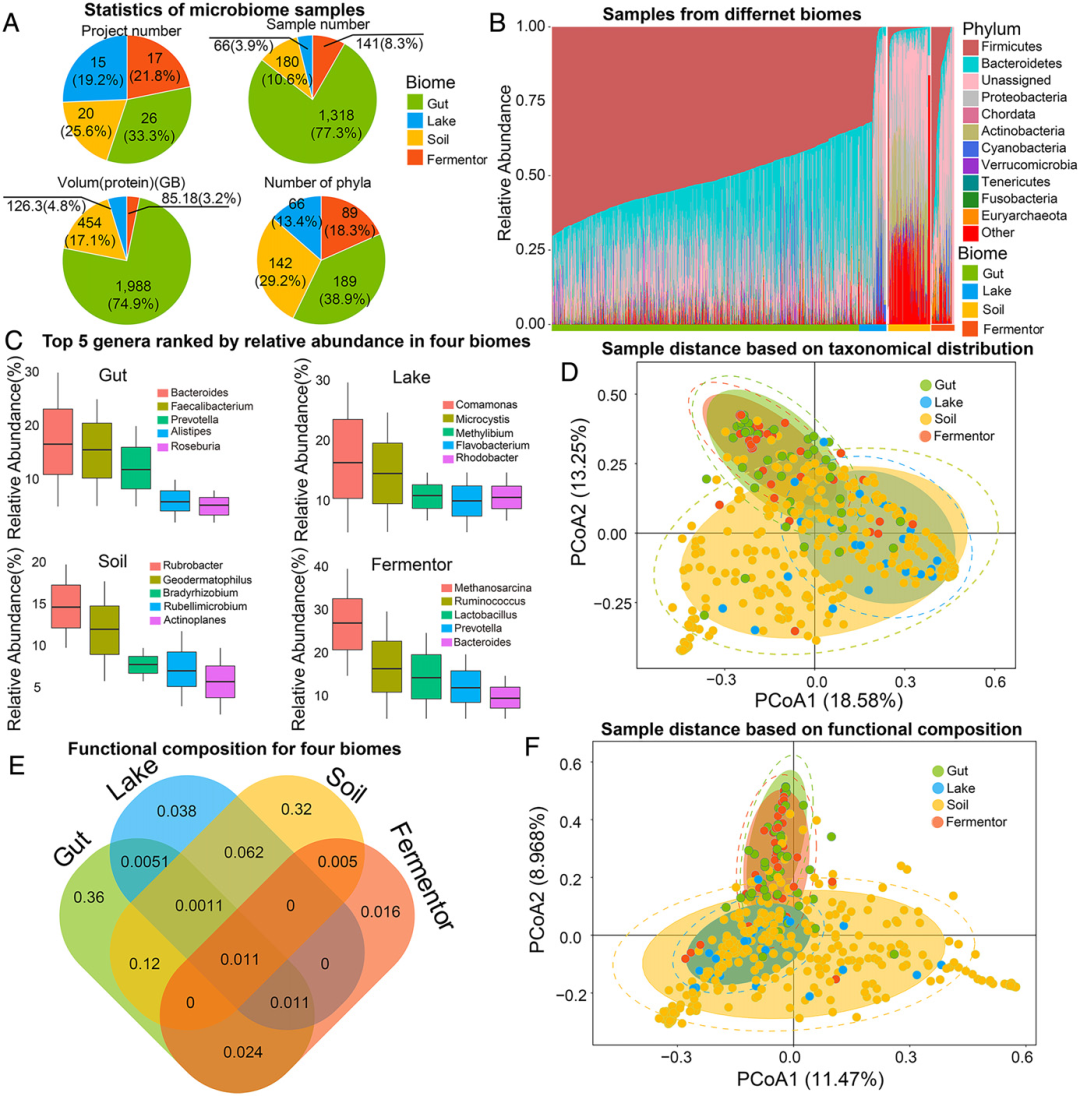
图 1. 不同微生物组样品的分类和功能分析。图片来源于PNAS
在从这4个生物群落中获得的42.5亿个蛋白质序列中,研究者观察到了生物群落特异性的功能图谱。与分类图谱相似,4个生物群落中存在不同的功能注释(图1E):仅在肠道生物群落中就检测到3.6亿个(68.4%)注释,湖泊、土壤和发酵生物群落中分别检测到0.038亿个(29.9%)、0.32亿个(62.7%)和0.016亿个(24.2%)注释。基于功能剖面的PCoA结果显示:这四个生物群落之间存在明显差异(图1F)。
宏基因组来源的蛋白助力上千蛋白家族无需同源模板即可成功结构建模
研究者从8700个结构未知的Pfam家族中,选取了2214个MSA >16 (= 24) 的Neff家族。从微生物组序列开始,将深度学习C-I-TASSER方法扩展到2214个结构未解决的Pfam家族来构建相应模型。基准测试结果显示,置信度(C-score)为-2.5的模型通常具有正确的折叠,C-I-TASSER发现Pfam家族中有47%(1044 / 2214)是可折叠的(图2A)。图2B是C-I-TASSER模型在2214个未知Pfam家族上的C-score直方图分布。图2C显示了C-score > -2.5的13个Pfam家族。虽然大多数靶标折叠正确,但有两个案例(PF3864和PF12357) TM-评分在0.5以下。
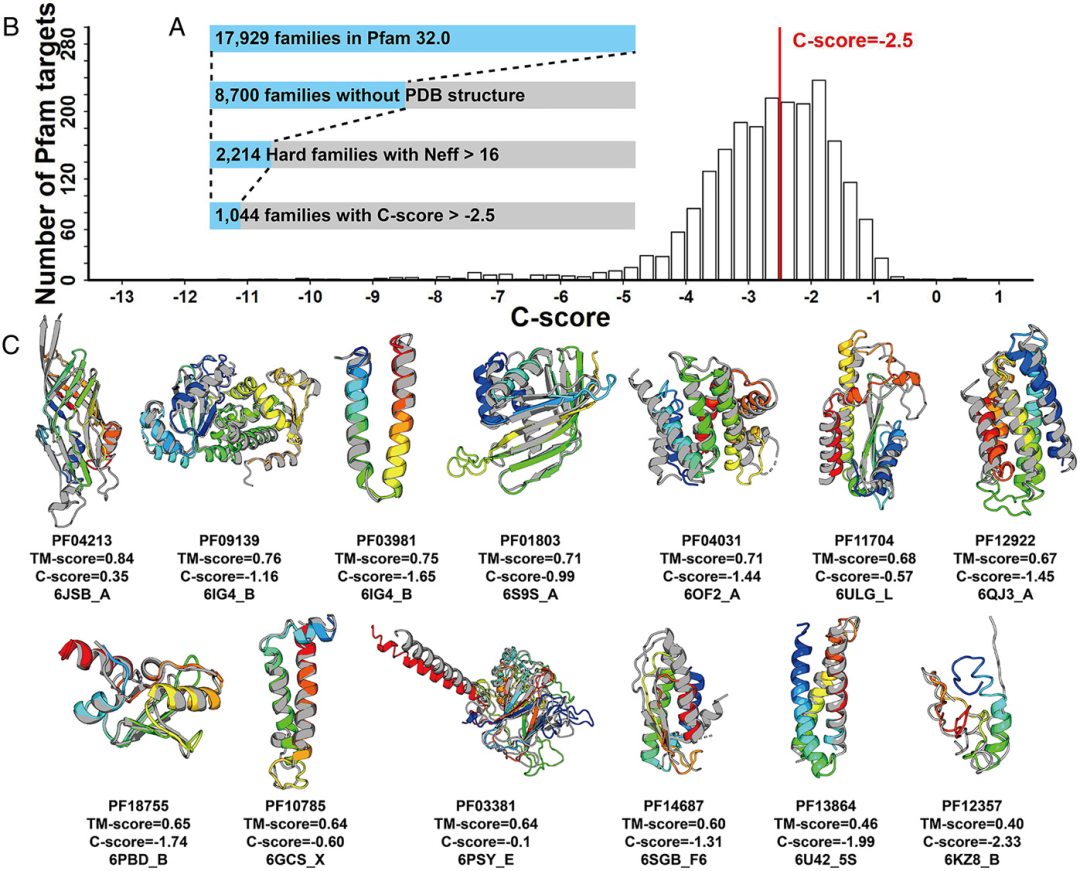
图2. 未知Pfam Hard家族的结构建模结果。图片来自PNAS
不同生物群落中同源序列的富集
C-I-TASSER预测可折叠的1044个Pfam家族中可以观察到从特定生物群系中富集的同源序列,在一个生物群系中可识别出964个Pfam家族(964/ 1044,92.3%),其Neff值大于其他三个生物群系,其中Gut为105户,Lake为116户,Soil为617户,Fermentor为126户(图3A)。
为了揭示已解决的Pfam家族在其生存环境中的重要作用,研究者对964个具有单个对应生物群系的Pfam家族分别进行了分类剖面和功能组成分析。结果表明了微生物群落序列对Pfam结构建模的特异性贡献(图3B)。与微生物群落样本相似, 964个Pfam家族的物种异质性分布反映了生物群落特异性的富集模式。此外,特定Pfam家族的优势种往往是MSA构建辅助微生物组样品的优势种。例如,在Pfam家族中标记了肠道生物群系(图1B和3B), Firmicutes和Bacteroidetes(均属于肠道)是Pfam家族的优势门(分别为0.41±0.28和0.26±0.14)和相应的源生物群系(分别为0.48±0.31和0.31±0.15),说明这种生物特异性富集模式受微生物群落组成的影响。
除结构建模外,964个Pfam家族的功能组成也为这种生物群落特异性的富集模式提供了有用的见解。例如,基于GO注释,368个Pfam家族被对齐到GO level-3 Biological Process(286)、Molecular Function(90)和Cellular Component(189)(图3D-F)。
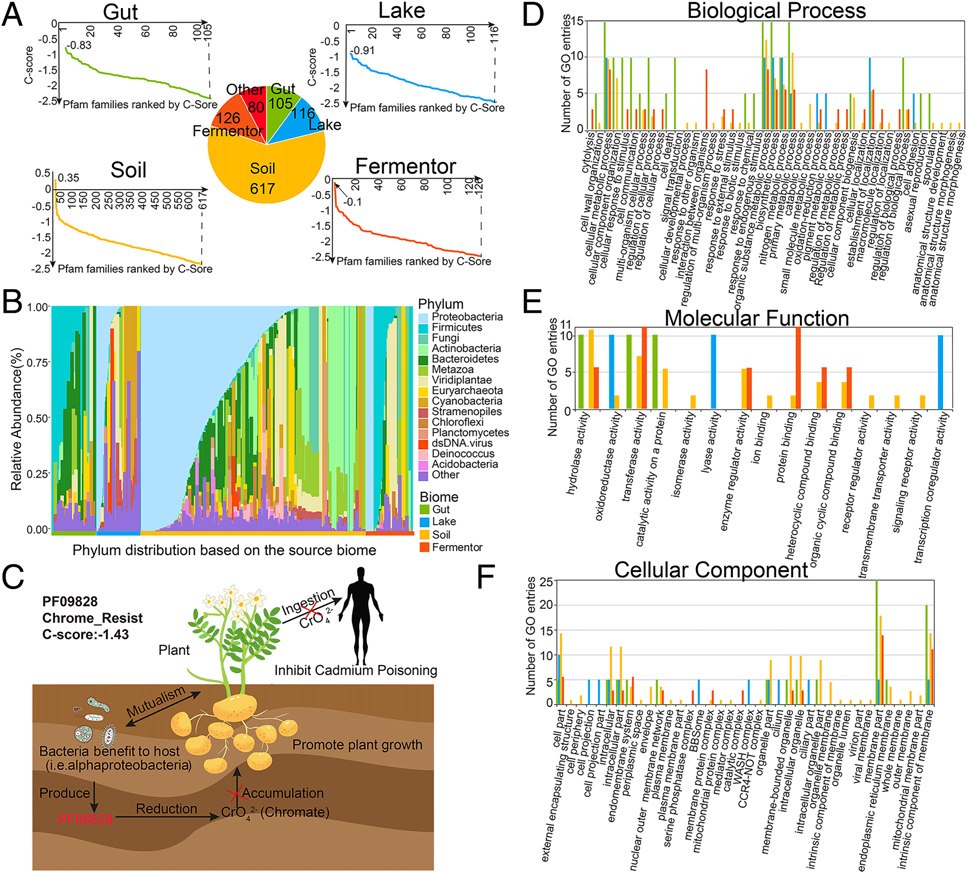
图 3. 基于C-I-TASSER的可折叠Pfam家族的分类和功能特性。图片来源于PNAS
边际效应分析揭示生物群落-序列-Pfam关系
研究者列出了四个生物群落对所有8700个未知的Pfam家族的边际效应;数据表明,不同生物群落对某一特定Pfam的贡献可能存在很大差异,这可以从它们的ME值中得到反映。在图4 A-D中,研究者展示了生物群落对PF04213、PF10785、PF13864和PF12357四个样本的MSA收集的贡献,其中微生物群落样本被随机分配到不同序列号的MSA收集中。对于不同的Pfam家族,序列同源库主要由不同的生物群落数据集组成,这再次表明在同源序列补充方面,生物群落和Pfam之间存在很强的联系。
在图4F中,研究者根据C-I-TASSER折叠结果,将Pfam家族分为两组。结果表明,C-score ≥ -2.5分家族的ME值明显高于C-score < -2.5分家族(t检验结果为5.27±3.44 vs 1.28±0.85, P值= 3.86e-26)。因此,边际效应值也与生物群系特异性宏基因组序列通过补充更多的同源序列来辅助三维结构组装模拟的能力密切相关。
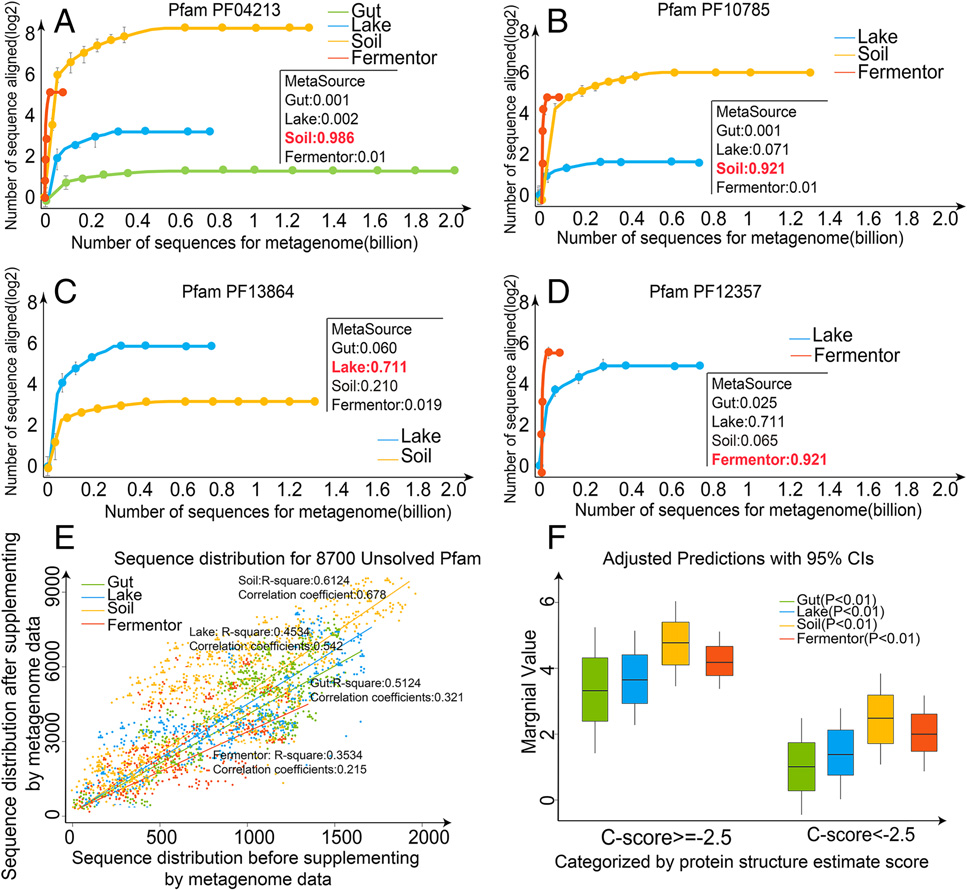
图 4. Pfam家族的边际效应评价。图片来源于PNAS
有效同源序列补充的MetaSource预测模型
研究者开发了MetaSource预测模型来识别一个或一组生物群系,可以更好地补充特定Pfam家族的同源序列集合。
首先,为了确定查询的Pfam家族的源生物群系是否为四个生物群系之一,研究者以单个生物群系标记的964个Pfam家族为训练集,7736个结构未知Pfam家族为测试集构建了二值分类模型。如图5A所示,在二元分类检验中,MetaSource在0.001个排列P值下的AUC为0.96。
其次,采用多分类随机森林算法来预测Pfam家族的四个生物群系中最可能的源生物群系。964个Pfam家族经过20次交叉验证迭代,结果显示MetaSource对Pfam家族具有很强的预测能力,在0.001排列P值下平均AUC为0.94。
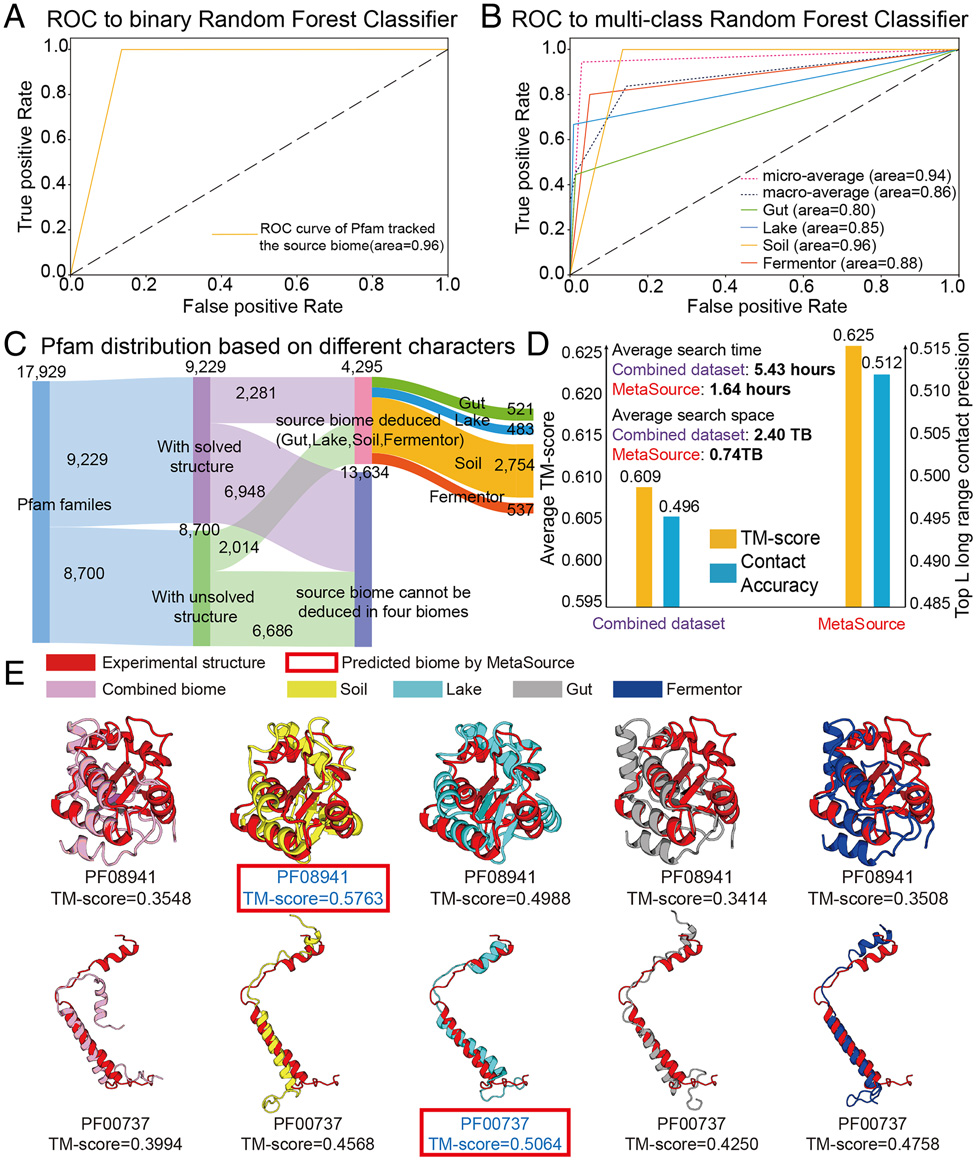
图 5. MetaSource对Pfam家族预测的源生物群系。图片来源于PNAS
为了进一步研究Metagenome数据库和MetaSource模型在3D结构建模中的实际用途,研究者将204个已知结构的Pfam家族纳入验证集。首先,C-I-TASSER利用基因组数据库中的MSA (DeepMSA的step 2结果)生成了TM-score = 0.583的模型,该模型仅使用Uniclust30基因组数据库,比C-I-TASSER模型高2.5%。
在DeepMSA的第3步添加宏基因组数据库后,C-I-TASSER模型的TM-score提高到0.609,比仅使用P值为3.8E-11的基因组数据库高出4.5%。这一结果,通过扩展MSA的覆盖范围和多样性,再次证明了宏基因组数据库在三维结构预测中的有效性。总的来说,通过将DeepMSA工具与宏基因组数据库相结合,C-I-TASSER模型的TM-score比简单的一步HHblitMSA收集方法提高了7%。
MetaSource能够预测获得最高Neff(或最高TM-score)的生物群落,在204个已知的Pfam家族的准确率为79.9%(或80.2%)(排列P值:0.001)。在图5D中,研究者进一步比较了C-I-TASSER模型。结果表明,虽然序列数据库的容量要小得多,使用Meta-Source的目标数据集的接触精度(0.512)和TM-score(0.625)却高于组合数据集(0.496和0.609)。
在图5E中,研究者给出了PF08941和PF00737两个已知结构的Pfam家族,MetaSource预测土壤和湖泊分别为最佳源生物群落。在这两种情况下,只有使用MetaSource预测生物群系的模型,才能创建一个TM-score高于0.5的模型。
总结
本研究利用2.4TB微生物组测序数据模型库,在更加宏观的层面对微生物组-序列-蛋白结构之间的关联性做了验证,并构建了机器学习模型来更高效地利用微生物组辅助预测蛋白结构及功能。
本研究仅以C-I-TASSER结构建模方法作为概念证明,更多的基因组数据集,包括其他生态指标,可在未来直接纳入这个模型。此外,随着该领域的快速发展,仅考虑接触图约束的C-I-TASSER,可能不再代表蛋白结构预测的技术水平。
该研究首次发现了微生物组的同源序列在不同生态位中的不均衡分布和富集特征,相关领域人员可以借此更深入地理解功能基因的适应性进化。靶向宏基因组选择方法能够对蛋白结构预测和功能注释的效率和有效性产生更加显著的影响。
参考文献
Cihan Ayaz, Lucas Tepper, Florian N. Brünig, Julian Kappler, Jan O. Daldrop, Roland R. Netz, Non-Markovian modeling of protein folding, Proceedings of the National Academy of Sciences Aug 2021, 118 (31) e2023856118; DOI: 10.1073/pnas.2023856118.