

使用氢-氘交换质谱(HDX-MS)结合计算建模,提供了一种新颖的方法来可视化降解诱导的三元复合物,体现了理性降解剂设计的思想,是第一个利用溶液数据的模型。
背景介绍
靶向蛋白降解(TPD)领域在过去的十年中呈指数增长,其目标是开发利用泛素-蛋白酶体系统标记蛋白质的破坏疗法。实现TPD的一种常见方法是使用一种异双功能分子,称为降解剂,将感兴趣的蛋白靶标招募到E3连接酶机制中促进泛素化,随后由蛋白酶降解。降解剂表现为连接酶激活剂,且不一定需要调节靶蛋白活性,这扩大了可调节蛋白的范围,有利于靶向一些难药靶点。
中间三元复合物(靶蛋白-降解剂-连接酶)的生成在降解过程中至关重要。理解三元复合物几何结构为选择性、催化效率、连接剂化学和合理的降解剂设计提供了有价值的见解。深入了解靶标相对于连接酶的定位,对合理的降解剂设计具有潜在的价值。
BRD4是一个有前景的肿瘤治疗靶点,且已有公司对BRD4的降解物进行了成药性研究。化合物CFT-1297是一种BRD4降解剂,具有强大的生物化学特性、极高效的体外降解能力和良好的药代动力学特性。
本研究中,作者利用HDX-MS识别降解诱导的蛋白-蛋白界面。HDX-MS是研究蛋白质构象动力学的一种强有力的技术。实验在溶液中进行以避免晶体接触。HDX-MS数据虽然信息丰富,但缺乏晶体结构的分辨率。因此,计算技术与HDX-MS衍生约束的结合对提高模型的实用性有潜在的协同作用。作者随后利用这些数据结合约束蛋白对接建立三元复合体的三维模型,进行进一步的分析。这是第一个利用溶液数据的模型,并提供了一种新颖的方法来可视化降解诱导的三元复合物以支持合理的降解剂设计。
CFT-1297促进BRD4-CRBN复合物形成
使BRD4在HEK 293T细胞中降解
首先,作者用实验证明了CFT-1297分别对BRD4和CRBN的结合,在一定浓度下可以促进三元复合物的形成,且相比于二元复合物,在CFT-1297存在时更倾向于形成三元复合物(Kternarytf = 4 nM vs Kbinaryd = 58 nM, 图1B,D)。实验数据表明CFT-1297具有较高的协同性(α=21)。另外,作者测试了CFT-1297在HRK293T细胞中降解全长BRD4的能力(图1E)。3h时,BRD4显著降解,DC50值为5 nM,蛋白水平降低至Emax为3%。这些数据突出了CFT-1297作为有力的候选化合物在三元复合物中连接BRD4与E3连接酶CRBN的能力。
另一个分析的降解剂为dBET6小分子,与CFT-1297具有相似的化学骨架。虽然同为BRD4降解剂,但与CFT-1297不同,dBET6在三元复合物中展现出较大的中立的蛋白-蛋白相互作用及较小的协同性(α=2)(图1D)。dBET6在3h时,于HEK293T中降解BRD4的DC50值为6 nM,Emax为3%(图1E),与CFT-1297相似。此外,之前报道的dBET6的负协同性与晶体结构所暗示的有利结合模式的观点相反。这些差异促使了对dBET6和CFT-1297的三元复合物形成的进一步分析。
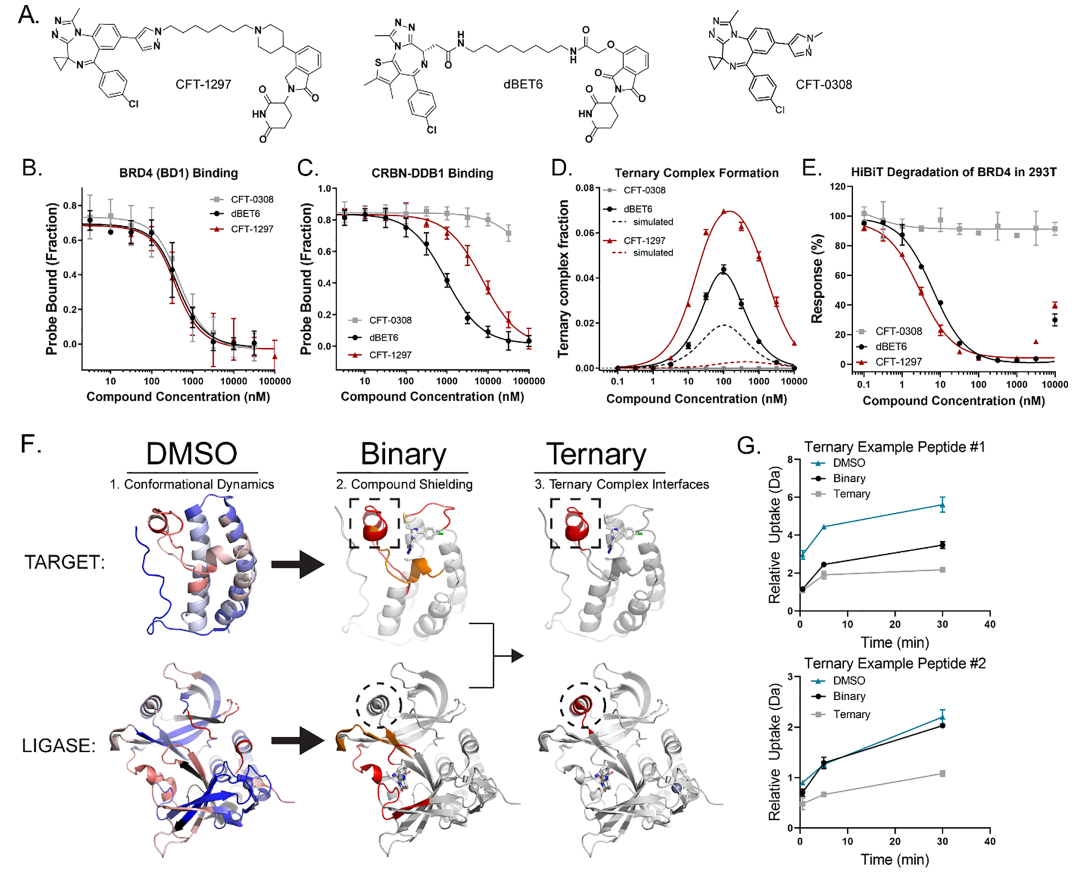
图1.. 化合物的特点及HDX方法流程。图片来源:ACS Chem. Bio.
CFT-1297在二元复合物中诱导BRD4(BD1)结构域溶剂屏蔽
使用HDX-MS来识别降解诱导蛋白-蛋白界面,其方法是检测复合物形成时屏蔽靶蛋白和连接酶的差异。在三元复合物形成之前,异双功能降解剂可以分别与每一种蛋白质接触。这种二元相互作用可导致在蛋白的结合位点发生屏蔽事件。
作者的HDX-MS实验遵循了一个严格的分析顺序:(1)连接酶和目标蛋白与二甲基亚砜(DMSO),(2)每个蛋白分别与感兴趣的降解剂,(3)包括连接酶、目标蛋白和异双功能降解分子的三元复合物(图1F,G)。使用这种方法可以评估蛋白质本身的动力学,了解由于降解物造成的屏蔽,然后确定哪些区域在这三种成分的存在下被屏蔽。
作者发现,在HDX-MS实验条件下,CFT-1297在BRD4的BD1结构域中诱导了溶剂屏蔽 (图2A,B)。在30分钟时,化合物结合引起的屏蔽与DMSO对照的分离最为显著。多肽的相对分数差异高达35%,相对摄取量为3.5Da差异。具有代表性的摄取图显示了具有显著保护作用的肽段(图2B)。
为了将这些肽映射到最相关的结构上,作者解析了BD1和CFT-0380的复合物晶体结构,CFT-0380是CFT-1297的靶向配体弹头。屏蔽肽都位于结合位点周围的ZA环(残基76−106),最大的屏蔽发生在αZ’螺旋上(图2C,残基88−93)。这个长环位于结合位点的下方,在没有化合物的情况下显示出显著的氘摄取(40-50%),这表明它在载脂蛋白状态下是非常动态的。溶剂屏蔽数据表明,CFT-1297能显著地稳定ZA环。
接着作者在HDX-MS实验中用CFT-1297对二元和三元复合物形成中的溶剂屏蔽效应进行了深入研究。结果表明,CFT-1297不会使CRBN-DDB1产生额外的溶剂屏蔽。
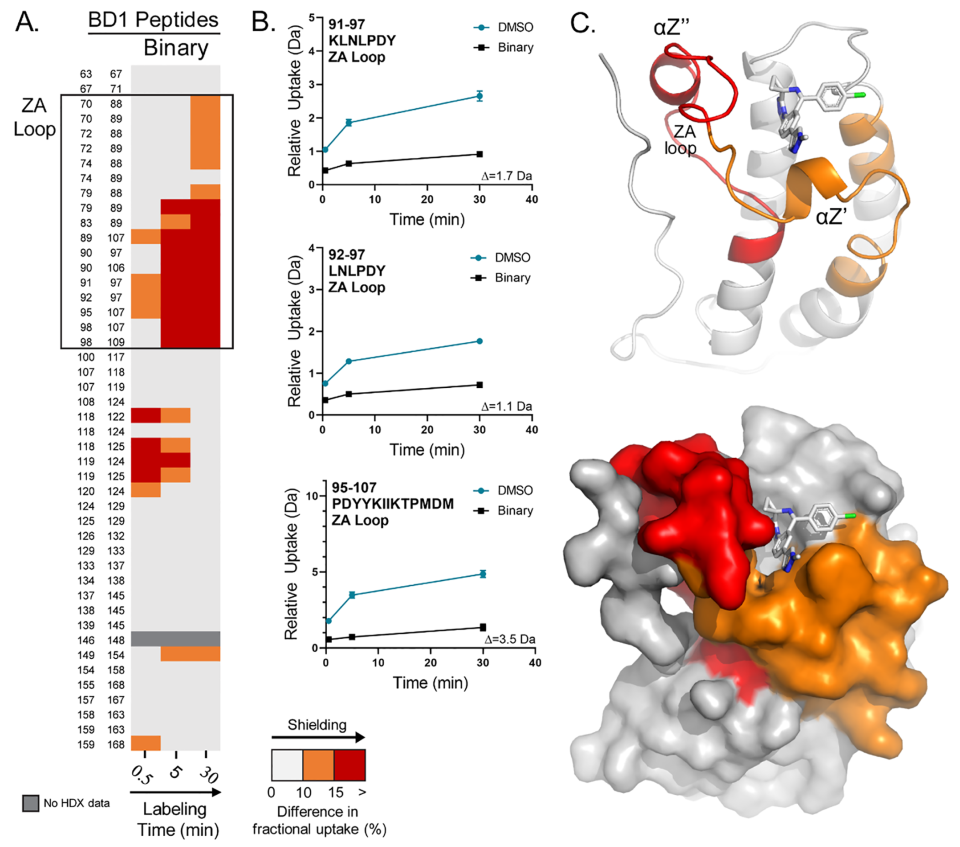
图2 HDX分析显示结合到CFT-1297的BRD4 (BD1)的ZA loop上有明显的屏蔽。图片来源:ACS Chem. Bio.
HDX-MS揭示了由CFT-1297促进的三元复合物的蛋白−蛋白界面
结合HDX-MS和AlphaLISA的二聚化分析结果,作者清楚地观察到了由降解物诱导的蛋白−蛋白界面促进的靶蛋白和连接酶蛋白上的额外屏蔽事件(图3)。这一结果为每个蛋白质上的区域提供了有价值的信息,增强了对三元复合物的几何形状和空间偏好的理解。使用dBET6进行的三元复合物实验没有产生额外的屏蔽事件。
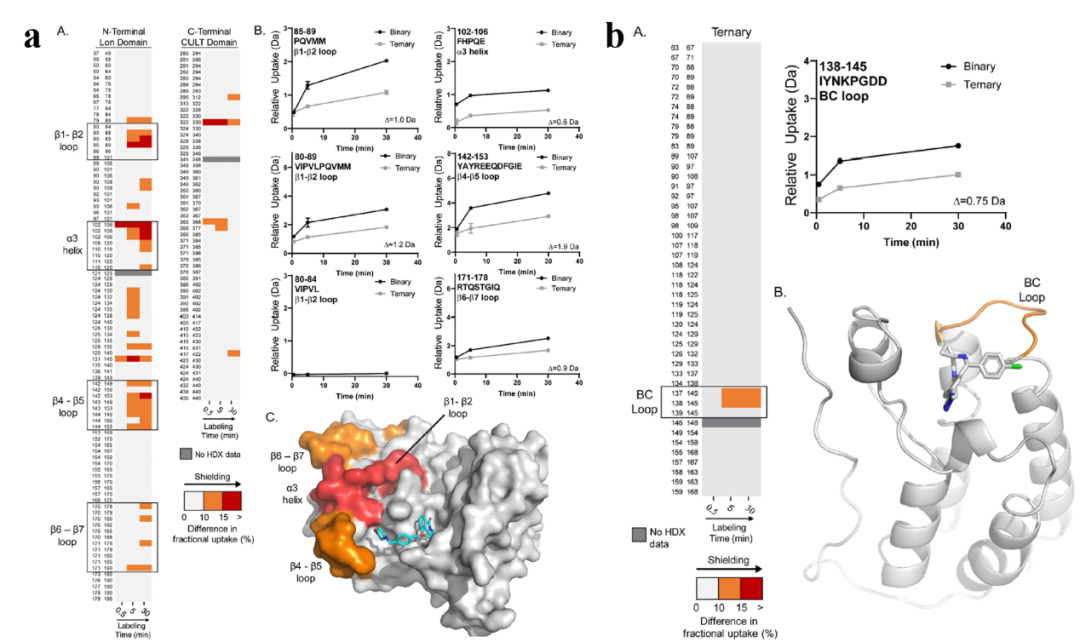
图3.(a)用CFT-1297进行的三元复合物实验, (b)BC loop环上可见三元复合物对BD1的屏蔽。 图片来源:ACS Chem. Bio.
CFT-1297的三元复合物对接和HDX评分结果
CFT-1297和dBET6的HDX-MS特征对比表明,降解物和E3连接酶产生不同的三元复合物构象取决于降解剂的性质。理解和可视化这些构象可以为未来的降解剂设计提供重要的见解。为此,作者结合HDX-MS数据,开发了一个计算三元复合物的对接工作流程。该方法添加了HDX约束和更详尽的构象采样。
以CFT-1297的数据为例(图4)进行了两个实验,分别采样了10,000和100,000个降解剂构象。每个构象都与BD1结合位点内的目标配体结构对齐。一个CRBN晶体结构,通过甘氨酸突变的环来软化柔性蛋白区域的冲突效应(基于CRBN结构集成分析),然后通过共享的CRBN戊二酰亚胺结合基序叠加到每个传递的构象上。任何导致冲突的降解构象(范德瓦尔斯半径为0.7)将在进一步的分析中被删除。
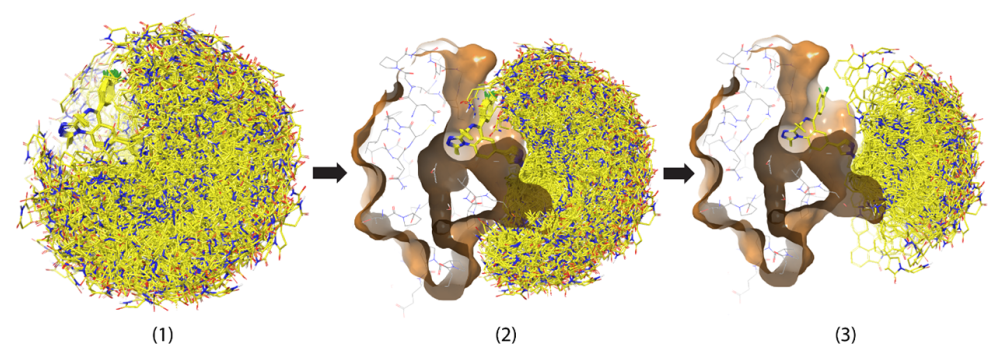
图4. 三元复合物(EDT)对接方法工作流程。图片来源:ACS Chem. Bio.
蛋白−蛋白界面侧链细化后,使用作者的HDX评分方法对得到的三元复合物进行排序,并使用ZRANK进行评分。HDX彼此间的得分和HDX与ZRANK之间的得分比对分别如图5A、B所示。根据BRD4 HDX评分选择了5种构象。这些三元复合物被分离出来并被覆盖(图5A,C),显示了一簇具有多个连接体取向的相关三元复合物。这五种三元复合物最有趣是因为它们的高靶标(BRD4) HDX评分,有利的ZRANK值,以及戊二酰亚胺以相同的方向聚集在一起,表明在一个类似的三元复合物周围振荡。
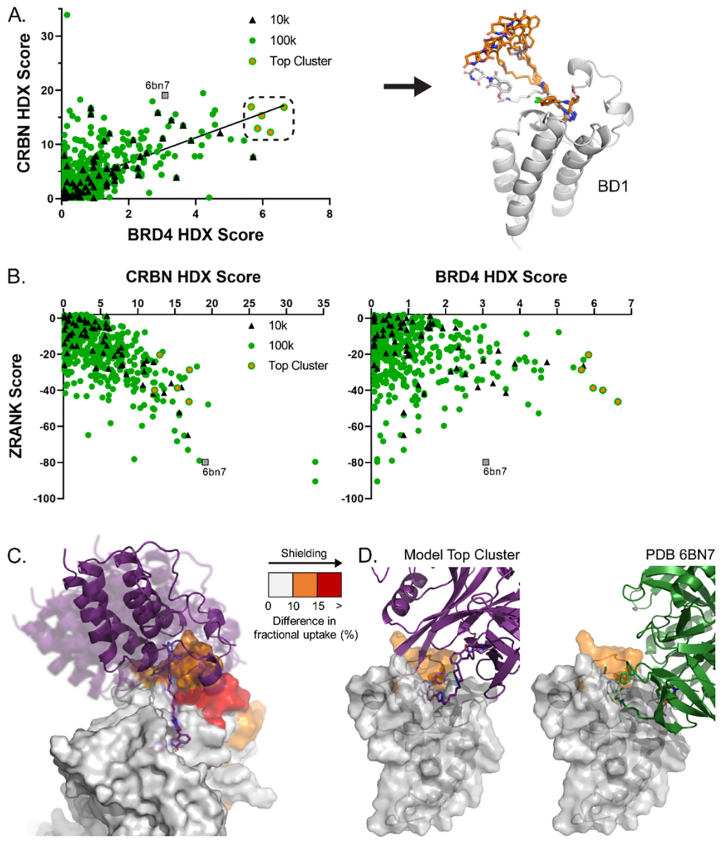
图5. HDX-MS数据与计算分析过滤结果相结合结果。图片来源:ACS Chem. Bio.
利用dBET23(PDB:6BN7)的三元复合物,将对接结果与晶体学数据进行了比较。选择这种结构是因为它与CFT-1297有相同的输出向量,更容易进行视觉比较。该结构的CRBN连接酶部分被作者将BRD4结构叠加到作者的对接模板上,并将对接结果作为HDX评分函数的输入(图5A,D)。在图5D中,作者强调了来自得分最高的集群的模型如何与在HDX实验中被屏蔽的BRD4(BD1)区域结合,同时6BN7以一种不能保护BD1域的构象扭曲。
结论
本研究表明,HDX-MS可以用于研究降解物诱导的三元复合物,当与约束对接结合使用时,提供了基于solution-state信息的三维模型。该方法被用于表征BRD4降解物CFT-1297和dBET6,它们表现出截然不同的氘吸收谱。三元复合物的结构和计算模型的发展是推进TPD领域的关键。HDX-MS数据很容易获得,并在溶液状态下提供有价值的信息。与计算分析相结合,可以通过识别新的模型和进一步的降解剂优化来加强三元复合物建模。
参考文献
Eron SJ, Huang H, Agafonov RV, Fitzgerald ME, Patel J, Michael RE, Lee TD, Hart AA, Shaulsky J, Nasveschuk CG, Phillips AJ, Fisher SL, Good A. Structural Characterization of Degrader-Induced Ternary Complexes Using Hydrogen-Deuterium Exchange Mass Spectrometry and Computational Modeling: Implications for Structure-Based Design. ACS Chem Biol. 2021 Nov 19;16(11):2228-2243. doi: 10.1021/acschembio.1c00376. Epub 2021 Sep 28. PMID: 34582690.