

一种基于注意力的卷积神经网络深度学习模型,该模型可利用初级序列生成的简单二级结构来提供高度准确的糖基转移酶折叠预测。
背景介绍
糖基转移酶(GTs)是一大类酶,其任务是合成复杂的碳水化合物,制造了细胞中的大部分生物质。CAZy数据库根据总体序列相似性,将50多万条GT序列划分为114个科。虽然家族内的序列具有可检测到的相似性,但家族间的序列却很少或没有相似性。GT序列的广泛多样性成为研究连接序列、结构、折叠和功能关系的主要瓶颈。
与其他大型蛋白家族一样,GTs在三维结构折叠中也表现出比初级序列更高的保守性。在所有114个家族中,只有3个主要的折叠(GT-A,-B和-C折叠)能被确定。目前,GT家族被划分为GT- A(34个),GT-B(32个)和GT-C(10个)。虽然,此前众多基于多序列比对的方法,为GT- A折叠结构和演化提供了见解,但这些方法无法处理其他结构数据或结构同源性有限的GT折叠的情况。
最近,深度学习方法特别是多层神经网络为大型复杂数据集中通过特征提取和模式识别进行的序列分类和折叠预测提供了新的机会。预测的准确性在很大程度上依赖于输入的多序列比对的质量且模型不能直接扩展到不同蛋白家族。因此,生成准确的多序列比对是一个挑战。此外,现有的深度学习模型的黑盒子性质,阻碍了对有助于结构或折叠预测的进化特征以及序列的直接生物学解释。
主要内容
来自美国乔治亚大学的Natarajan Kannan等研究者报告了一种基于注意力(CNN-注意力)的卷积神经网络模型,该模型将二级结构注释作为输入,用于预测GT-折叠类型。这些粗粒度输入特征是基于蛋白质二级和三级结构远比一级序列保守的这一前提。该模型没有使用氨基酸的物理化学性质,也不依赖于产生进化或基于对齐的信息,但是折叠预测的平均准确率达到了96%,家族分类的平均准确率为77%。研究者进一步利用开放集识别的最新进展,并使用一个特殊修改的重构误差损失项来确定GT之间的相似性,从而将该模型扩展到已知的GT折叠之外。该模型主要有三个方面的优点:(1)研究者提出了一种利用二级结构预测作为输入数据来探索蛋白质折叠的无比对方法;(2)关注模型的可解释性,能挖掘模型学习到的特征并进行有意义的生物推理;(3)训练后的模型可扩展到未知折叠的GT家族中,并预测出最可能采用新折叠类型的GT家族以指导新糖基转移发现的进一步研究。相关的研究成果以“Mapping the glycosyltransferase fold landscape using interpretable deep learning”为题发布在国际著名期刊Nature Communications上。
用于识别、分类和预测糖基转移酶折叠的深度学习框架
研究者从CAZy数据库中收集了50多万条GT序列,并根据序列相似性、长度等标准对其进行筛选,生成了44,620条GT序列用做训练数据,涵盖所有的折叠和家族。同时,研究者使用NetSurfP2.024识别的二级结构模式,并将其作为唯一输入,训练六层CNN模型进行多任务折叠和家族分类(图1)。
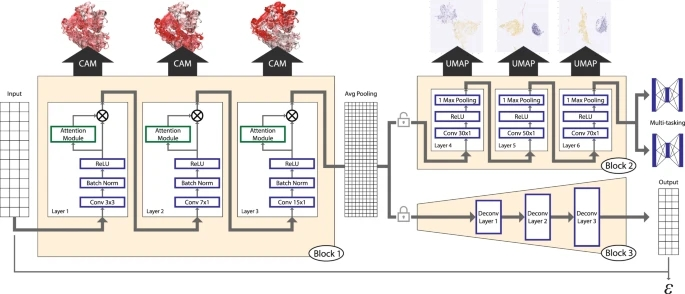
图 1. 所使用的深度学习模型的总体示意图。图片来源于Nat. Commun.
CNN模型的前三层(第1块, 图1),在类标签引导下学习保守的二级结构特征中不同层次的模式。这些特性被存储为特定于层的权重以及它们的空间分辨率,从而可以使用grade -CAM(CAM)的特定于类的激活映射等方法将它们投射回线性序列和3D结构中。CAM值可以用来识别模型所给定GT折叠的特征。最后三层(第2块, 图1)进一步优化关联特征权重值,然后将其送入全连接多任务分类器中,生成高精度的分类。研究者提取这些优化的特征嵌入,并使用统一流形近似和投影(UMAP)进行降维分析,以将分类做可视化处理。
为了对未知结构或折叠的GT族进行分类,研究者将一个自动编码器框架集成到现有的模型中,其中从Block 1中优化的权重被冻结,并用作编码器的通用特征提取器。然后将Block 3(图1)设计为具有CNN模型镜像结构的解码器,进行反卷积操作。
所有GT折叠的绘景揭示了主要折叠类型内的不同簇
UMAP算法可将Block 2的三层特征图可视化(图2a)。正如预期的那样,研究发现了所有主要GT折叠之间的分离,突出了模型的区分能力。
此外,研究者还发现了GT-A、-B和-C折叠类型的不同子结构。为了进一步分析这些子结构,研究者首先对三种折叠类型中的每一种进行单独的UMAP分析,并使用高斯混合模型(GMM)算法对产生的投影进行聚类,以识别主要GT折叠类型中的聚类。这获得了两个GT-A簇和三个GT-B和GT-C簇。
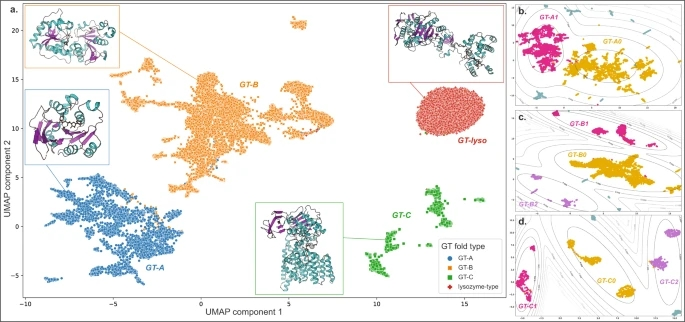
图2. UMAP投影显示了主要GT折叠类型的分离。图片来源于Nat. Commun.
两个不同的GT-A簇占了大部分的家族,34个家族中的17个组成了一个更大的GT-A0簇。10个家族被归为GT-A1簇,而其余7个家族没有被归为簇,分散在两个中心簇之外(图2b)。同样,GT-C序列也分散在三个主要的簇中(图2d),十个家族中只有两个(GT59和细菌GT85家族的Alg10糖基转移酶)没有被归为其中任何一个簇。
GT-A簇的CAM映射突出了共享结构特征的差异
为了解主要GT折叠及其各自簇的结构特征,研究者将CNN模型前三层的每一层得到的CAM值映射回它们各自的序列。研究者首先将CAM值映射回GT-A公共核心对齐(图3a, b)。
研究发现GT-A核中最保守的区域(如DXD motif、G-loop和特征Rossmann折叠的前两个beta片)对应着CAM值最高的区域,表明该模型是利用这些保守区域来区分GT-A型折叠与其他GT型折叠类型的。
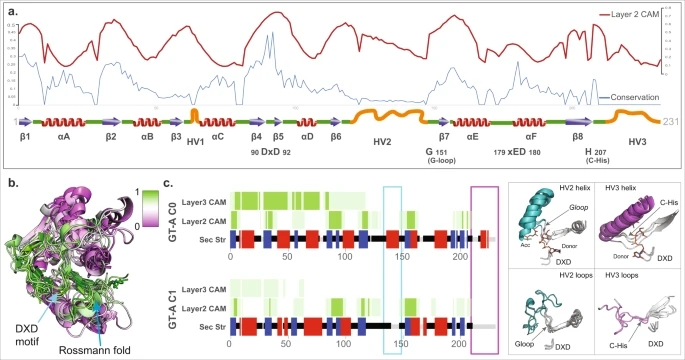
图3. CAM突出GT-A折叠核心。图片来源于Nat. Commun.
第2层生成的CAM图谱信息量最大,与GT-A折叠的核心特征匹配良好。第1层的CAM值对应于分散在整个域内的微小区域,可能表明了模型学习到的局部特征,而第3层的CAM值延伸到更长的连续区域(图3c),可能捕获了远程相关性。
GT-B和GT-C簇的多重保守核心

图4. 不同GT-B和GT-C折叠簇的CAM图突出了它们各自的保守核心。图片来源于Nat. Commun.
由于缺乏序列守恒,生成GT-B折叠宽序列比对尤其具有挑战性,为了理解从CNN模型中获得的模式,研究者为每个GT-B家族生成了家族水平的比对。而后研究者计算了每个家族的共识二级结构和平均2层CAM图(图4a)。这些家族都反映了GT-B折叠的典型的两个β/α/β Rossmann-折叠结构域。CNN-注意模型中最一致的模式是C端Rossmann折叠。CAM值图表明,与6个beta片相关的特征在区分GT-B族方面有重要地位,而且这个C端区域的保守性也从GT-B0扩展到GT-B1、GT-B2和其他未分组的GT-B族。
此外,将CAM值与代表性结构进行映射显示,在GT-B家族中,C端Rossmann-fold的取向和结构是很保守的,偶尔会在环区插入家族特异性的片段(图4b)。因此,该研究支持C端Rossmann域是GT-B折叠家族的共同结构特征。
在C端Rossmann折叠上游,N端Rossmann折叠区二级结构的CAM值也较高,这可能表明它对于区分有2个Rossmann折叠的GT-B折叠和只有一个Rossmann折叠域的GT-A折叠具有重要意义。然而,这些CAM值模式在不同的家族中并不一致。
相反,GT-B2簇内的所有家族都被发现在N端Rossmann折叠中保留了至少6个beta片和5个alpha螺旋,如CAM值所示(图4a,b中的洋红色框), GT-B2核心的扩展包括N和C端Rossmann折叠域。
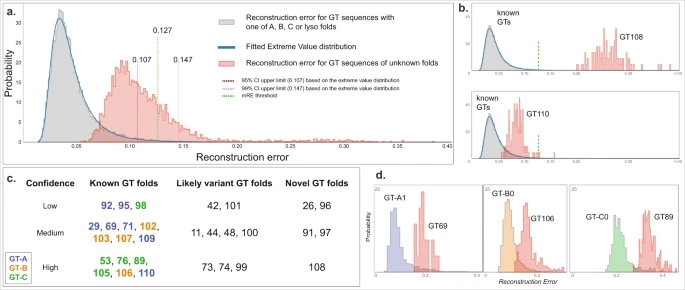
图5. GT-u族的折叠预测。图片来源于Nat. Commun.
利用卷积自动编码器模型识别具有新型GT折叠的族
图5a显示了已知序列(GT-A、-B、-C和-lyso为灰色)和未知折叠序列(红色)的RE分布。
为了从统计上评估哪些GT-u族具有显著高于已知折叠的正则表达式,研究者首先对训练数据拟合了一个极端值分布以计算95%和99%的置信区间(CI)。然后,将每个GT-u家族的中位RE值(mRE)与这些CI进行比较,以做出折叠预测。作者注意到未知的RE分布的峰值落在95% CI范围内(低于0.107,图5a),这表明大多数GT-u序列采用了一个已知的折叠。
研究者进一步为两个GT-A、三个GT-B、三个GT-C和一个GT-lyso聚类建立了9个自动编码器模型并计算了RE。如果GT-u家族的mRE小于0.127,但FAS评分无阳性,则被认定为变异折叠型。最后,将mRE大于0.127的科定义为新型折叠类型。
研究表明,有5个家族的mRE非常高(大于0.127),预测将采用新型GT折叠(图5c)。研究者预测了其中三个家族,真菌β-1,2-甘露糖基转移酶Bmt/Wry (GT91),植物肽基丝氨酸α-半乳糖基转移酶Sgt (GT96)和细菌α-2,6-唾液酸基转移酶(GT97),它们可能采用新的GT折叠。
研究者还确定了四个最有可能采用GT-B折叠的GT-u家族(图5c),包括:细菌α-1,3- l -鼠李糖基转移酶(GT102),细菌O-抗原多糖β-1,4-N-乙酰氨基葡萄糖转移酶(GT103),植物鼠李糖半乳糖醛酸I 4-α-鼠李糖基转移酶的GT106家族,以及KDO转移酶的GT107家族。
结论总结
本文用一个简单的训练数据集构建了一种用于预测GT折叠类型的卷积神经网络模型。该模型学习的特征可以成功地区分蛋白激酶折叠序列和非蛋白激酶折叠序列,准确率约为96%。与GTs相似,该模型也分离了主要的激酶基团,准确率为77%。
与大多数“黑盒”深度学习模型相反,该工作流的输出是一种高度可解释的深度学习模型,可以产生精确的折叠预测和定量输出并提供有意义的生物学见解,而不需要主要序列或结构对齐。因此,该方法为大型蛋白质家族的计算和进化分析增加了一个强大的工具。
参考文献
Taujale, R., Zhou, Z., Yeung, W. et al. Mapping the glycosyltransferase fold landscape using interpretable deep learning. Nat Commun 12, 5656 (2021). https://doi.org/10.1038/s41467-021-25975-9