

通过实验结合计算的方法,证明了四环素衍生物对FP-2的变构抑制作用,预测了可能的结合模式,为进一步优化四环素骨架及发现选择性更好效力更强的抑制剂奠定了基础。
背景介绍
恶性疟原虫半胱氨酸蛋白酶falcipain -2(FP-2)被认为是开发新抗疟药物的潜力靶点。多年来,许多FP-2高效抑制剂因其对人类组织蛋白酶脱靶的低选择性而均未进入临床。
FP-2属于C1半胱氨酸蛋白酶家族,这种酶显示出疟原虫半胱氨酸蛋白酶共有的两种独特的结构特征:“鼻状”N-端延伸(FP-2鼻)和突出到溶剂中的β-发夹 (FP-2臂)。缺失分析表明,FP-2鼻是重组酶正确折叠所必需的,FP-2臂是血红蛋白的远端识别位点,当臂缺失时会失去血红蛋白酶活性。目前为止,绝大多数已知的FP-2抑制剂都是正构或竞争性抑制剂。阿根廷圣马丁国立大学的Emir Salas-Sarduy团队之前的研究发现四环素系列的抗生素甲烯土霉素是FP-2的弱非竞争性抑制剂,表明变构可以作为一种抑制FP-2的替代机制。
变构位点通常在进化上不那么保守,因此它们提供了一种跨受体家族实现亚型选择性的途径。此外,它们允许调节蛋白质活性(而不是完全废除),这可能有利于恢复生理系统的良好功能。较正构药物而言有更多实质性的优势。
文中,作者评估了四环素家族其他成员对FP-2的抑制活性,即四环素、土霉素、金霉素、多西霉素和米诺环素(图1),并确定其抑制机制。确定该系列的活性不仅可以作为构建结构−活性关系的起点,还可以为阐明其结合位点提供线索,也可以为设计新的、更有效的变构抑制剂奠定基础。
化合物的发现和机制确证
首先,作者通过实验的方法确定了FP-2可以被几种四环素衍生物所抑制,并且具有亚摩尔级别的亲和力。这些化合物均表现出独特的非竞争性抑制剂模式,在亚摩尔范围内都表现出高度相似的Ki值,α值范围为0.6-0.9。(图1)
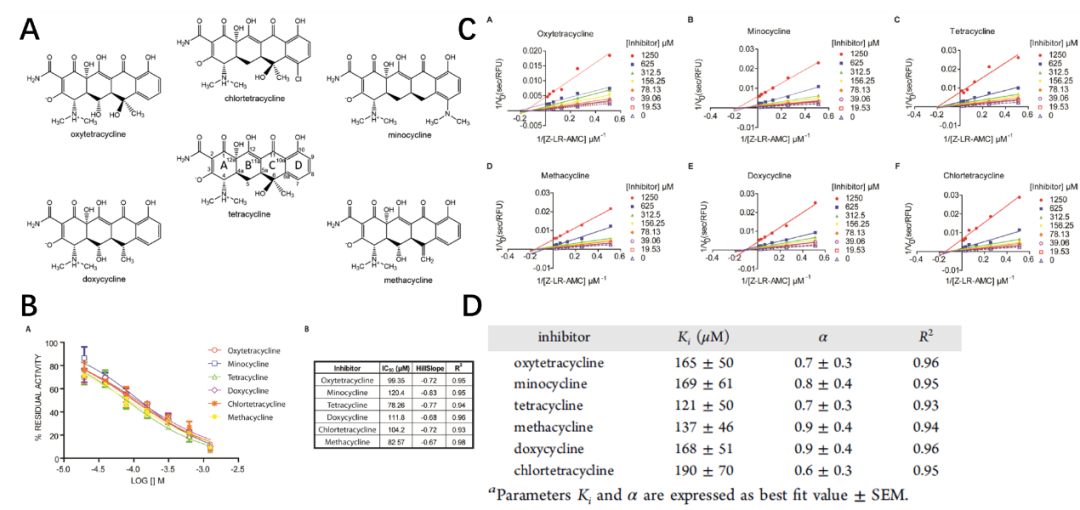
图1. 四环素衍生物的结构及其抑制FP-2的实验结果。图表来源:JCIM
四环素衍生物的非竞争性抑制确证
根据已有的非竞争性抑制剂EC48与FP-2的共晶结构(PDB:6SSZ),作者分析了四环素衍生物与FP-2非竞争性结合的机制。作者使用一个大的FRET底物进行了抑制实验。FRET底物长28个残基,最早用于开发FP-2的异质酶分析方法。FRET理论上可以完全阻断EC48样的结合模式,阻止四环素衍生物与FP-2活性位点的S侧的结合,并以高浓度这种经典竞争方式取代它们。
作者分析了所有化合物在三种不同浓度的FRET底物下的剂量−响应曲线,并计算了最高(5×KM)与最低(0.25×KM)底物浓度的实验IC50比值(REXP)(图2),用于对每种四环素衍生物的抑制机制进行分类。在所有情况下,REXP值显著低于竞争性抑制剂理论预期的值(RTHEO=4.8),更接近非竞争性抑制剂 (RTHEO=1)。有趣的是,三种抑制剂显示REXP<1,金霉素在该系列中REXP值最低,与该化合物的最低α值一致。这表明金霉素对酶−底物复合物的结合偏好,是一种非竞争性样行为。
结果表明,与底物长度无关,四环素衍生物是FP-2的非竞争性抑制剂。因此,混合抑制剂EC48观察到的与S’侧的结合被排除。因此,作者随后使用了计算的方法来预测所研究的四环素衍生物最有可能的结合位点。
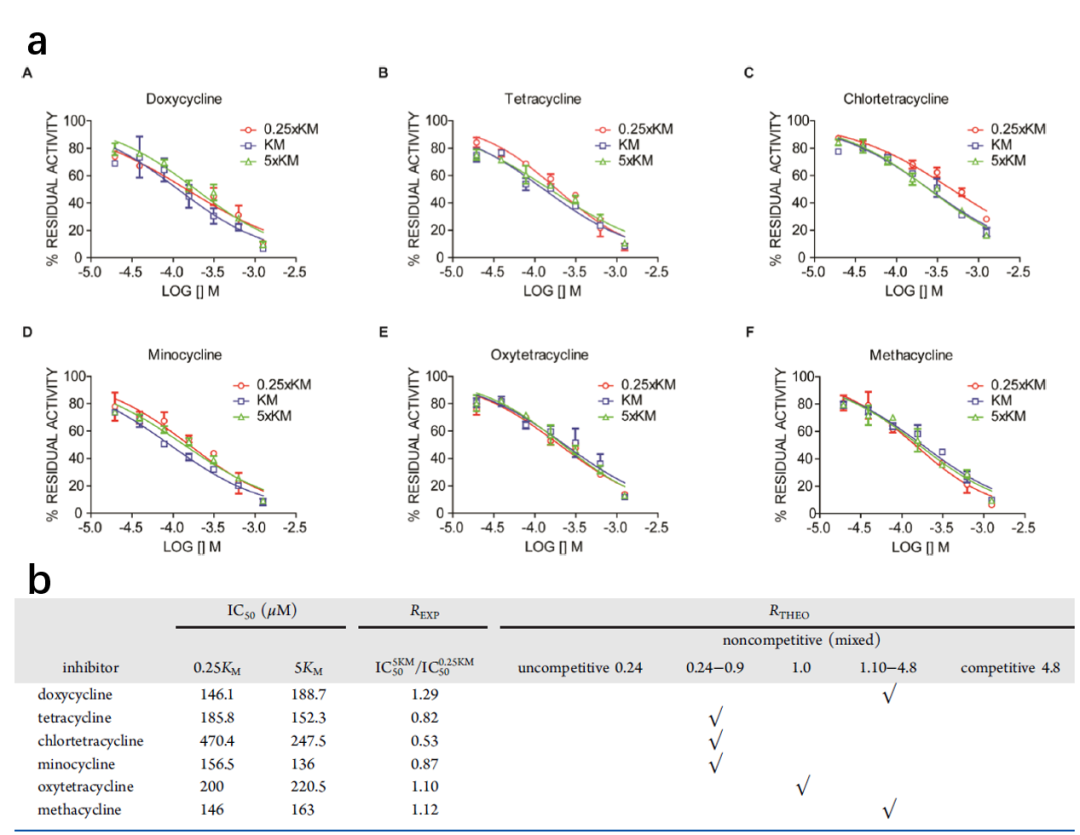
图2. 使用FRET做底物,四环素衍生物的非竞争性抑制确证结果。图表来源:JCIM
MD模拟和自由能计算预测四环素衍生物
与FP-2远端变构位点的结合模式
将配体放置在远离蛋白质表面的不同位置后,通过多个2μs MD模拟研究了甲烯土霉素与FP-2的结合模式。通过计算分析,确定了甲烯土霉素与FP-2的最低能量结合模式为A.11.0。
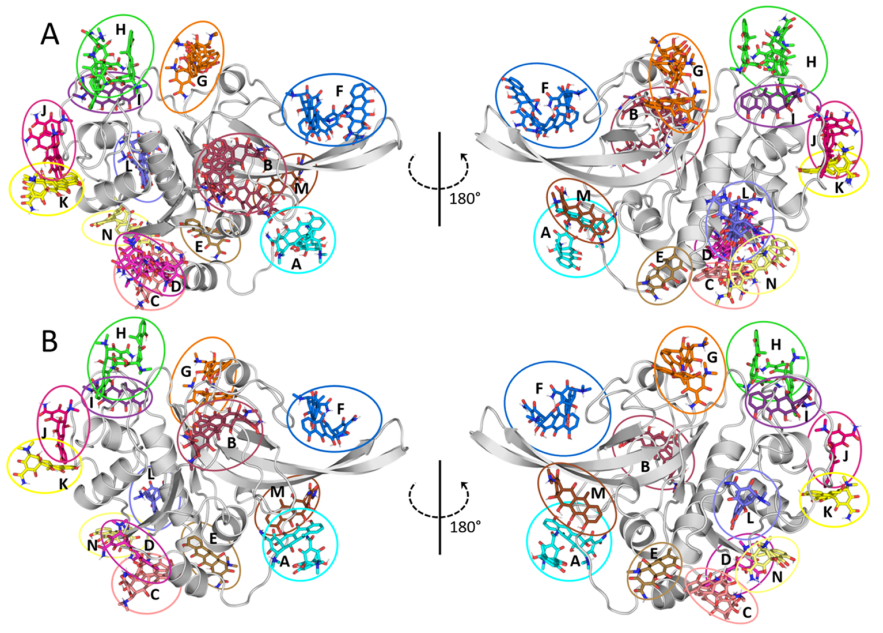
图3. MD模拟中甲烯土霉素与FP-2的主要结合模式。图片来源:JCIM
FP-2:甲烯土霉素复合物的预测结构表明,甲烯土霉素结合了一个远离活性位点凹槽的变构位点,与非竞争性抑制机制一致(图4A)。结合位点相当于先前表征的同源蛋白酶人组织蛋白酶K(hCatK)的变构位点,称为位点6。此外,在变构位点界面观察到的关键相互作用属于保守的四环素骨架,而可变区域主要暴露在溶剂中。这一特征是解释这里研究的所有四环素衍生物的类似抑制效力的基础。
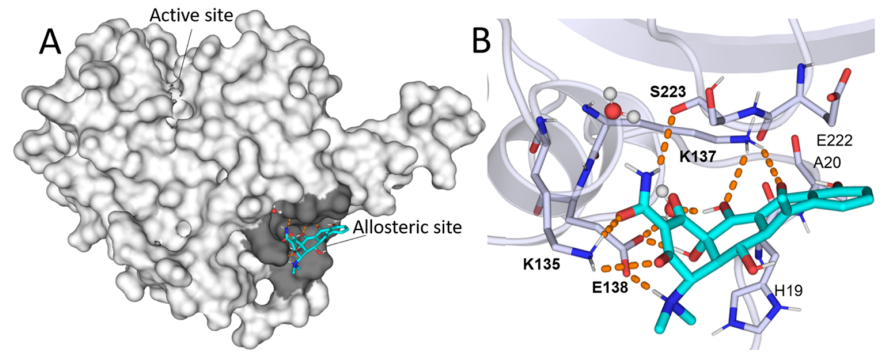
图4. 预测的结合模式。图片来源:JCIM
在识别最低能量的甲烯土霉素结合到位点6后,作者通过在分解转换期间增加λ窗口的数量,运行复制MD模拟,并将模拟时间从10ns延长到30ns,对该复合物的ΔG0bind值进行了精确的计算。最终得到FP-2:甲烯土霉素复合物的ΔG0bind值为−5.7 kcal/mol,比简化方法计算的大2.4 kcal/mol,与Ki估计的实验亲和力(−5.2 kcal/mol)非常一致。改进后的方案允许获得一个可靠的ΔG0bind,calc值,其误差相对于实验值< 1 kcal/mol,这提高了预测结构的有效性。
作者随后通过计算二甲胺四环素的ΔG0bind值,来评估四环素支架可变区域的化学修饰对FP-2亲和力的影响。选择二甲胺四环素是因为它与甲烯土霉素相比具有最显著的结构差异。使用构建的FP-2:二甲胺四环素结构模型进行的TI自由能计算表明,ΔG0bind,calc值为−5.0 kcal/mol,与实验值相匹配(−5.1 kcal/mol)。因此,所提出的结合模式至少与两种明显不同的四环素衍生物所显示的FP-2的类似实验亲和力相一致。实验测量结果证实了化合物在溶剂暴露的可变区的修饰几乎不会改变其对FP-2的亲和力。
计算分析甲烯土霉素对FP-2活性位点的修饰
作为变构抑制剂,当甲烯土霉素结合于位点6时可能会扰乱FP-2的构象平衡。作者结合群落和信号通路分析来调查这一现象。CA采用蛋白的网络表示,其中Cα原子对应于由表示成对关联的边连接起来的节点。这个网络聚集在高度相关的残基组中,这些残基的集体运动与蛋白的其余部分的耦合更松散,形成所谓群落。另一方面,通路分析通过计算连接两个区域的信号通路如何在蛋白质结构中传播,揭示了变构位点与活性位点的耦合。通常,这些技术依赖于从所研究系统的MD轨迹计算出的相关矩阵。
使用0.5截止值和接触图过滤,过滤原始相关矩阵后进行CA。网络被划分为最小数量的群落,在0.01的容忍范围内最大化模块化。作者发现,在存在甲烯土霉素的情况下,FP-2被分成8个群落,比游离状态多一个(图5A)。结合甲烯土霉素后,FP-2群落显著重组,包括那些形成活性位点的群落。
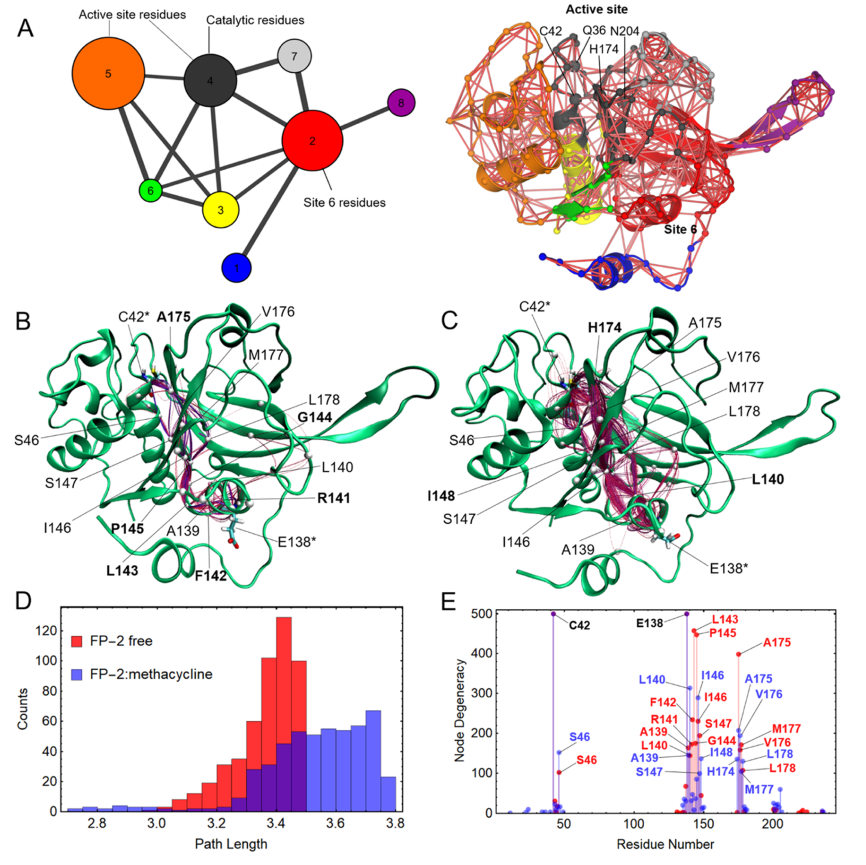
图5. 计算分析甲烯土霉素对FP-2变构抑制作用的分子基础。图片来源:JCIM
作者通过计算E138到C42之间的信号通路,分析了在甲烯土霉素存在和缺失两种情况下活性位点和位点6之间的联系。基于E138与甲氧环素的多重氢键结合,选择其作为源残基(source residue)(图5B),它很可能在配体与变构位点的结合中发挥关键作用。另一方面,C42是主要的催化残基,负责对底物的易剪肽键的亲核攻击。此外,C42和E138分别位于相反的区域(L和R),这意味着计算的路径将显示变构信号如何通过FP-2的域间空间传播到活性位点。这两个特征使作者选择C42作为合适的sink residue。
分析结果表明,甲烯土霉素在结合时干扰了与活性位点耦合的变构位点,不仅干扰了介导这种联系的残基,而且还干扰了游离和复合物状态时的FP-2的路径长度分布。另一个分析这种变化的方法是通过确定信号路径上的节点简并度,即在两个系统中,特定残基在整个计算路径集中出现的次数。从图5E可以看出,R141、F142、L143、G144、P145和A175在自由FP-2中参与的信号传播明显多于FP-2:甲烯土霉素配合物。相反,L140、I148和H174在后一种体系的路径中被过度表示(图5C、E中加粗标记的残基)。这些结果进一步说明了甲烯土霉素对活性位点和变构位点之间联系的影响。
讨论
本文首先通过实验证明了具有四环素骨架的化合物以非竞争性的方式抑制FP-2,Ki值在121~190μM之间。再通过计算的方法,研究了配体结合时的结构修饰作用。结合配体向蛋白质表面自由扩散的长MD模拟和精确的结合自由能计算,构建了一种强有力的计算方法,可以预测这些化合物的结合模式,结果与一些实验结果一致。此外,生成的结构是一个有价值的起点,可以优化四环素骨架实现更有效的抑制以及对该变构位点使用虚拟筛选方法来发现新的FP-2抑制剂。
参考文献
Hernández González JE, Alberca LN, Masforrol González Y, Reyes Acosta O, Talevi A, Salas-Sarduy E. Tetracycline Derivatives Inhibit Plasmodial Cysteine Protease Falcipain-2 through Binding to a Distal Allosteric Site. J Chem Inf Model. 2021 Dec 28. doi: 10.1021/acs.jcim.1c01189. Epub ahead of print. PMID: 34962803.