

薛定谔公司和UCSF的研究者通过2个靶点的回顾性实验及1个靶点的盲测,展现了主动学习技术降低对接成本的强大能力,仅略损失hits召回率,使超大规模虚筛平民化。
背景
随着按需合成的化合物库的出现,用于虚拟筛选的可购买化合物的数量近年来呈爆炸式增长,已超过了 10 亿种化合物。例如,ZINC 库在 2016 年 4 月包含大约 1 亿个化合物,2019 年 7 月为 5 亿个,2020 年 1 月为 10 亿个,以此推算,未来5年可购买化合物将超过百亿。用传统的基于对接的虚拟筛选方式来筛选这类超大虚拟库的成本水涨船高。最近的两个相关研究无论是使用OpenEye的Orion云计算平台还是VirtualFlow进行超大规模筛选都需要耗费大量的计算资源。这类对接虚筛在文献中十分少见也侧面反映了此方式所需成本高昂。主动学习已经在药物发现领域有许多成功的案例,包括对接及自由能计算。但至今仍缺少基于真实的超大规模虚筛及测活数据来对比传统对接与主动学习辅助对接的命中率及多样性的研究,而这正是今天这篇文章所关注的。
主动学习的整体流程
作者使用了3个筛选体系,虚筛库的大小分别如下
1.AmpC:99,459,562个分子;
2.DRD4:138,312,677个分子;
3.MT1:150,927,915个分子。
值得一提的是,这三个靶点分别倾向于结合阴离子、阳离子和中心分子。
对接软件则使用了DOCK3.7 和 Glide SP,主动学习流程通过Schrödinger Suite 中提供的 AutoQSAR/DeepChem (AQ/DC) 包开发,机器学习模型包括GCNN和ECFP-RF。AQ/DC可以自动使用5折CV进行模型超参数优化,并最终给出15个模型进行集成,集成模型使用所有模型预测平均值作为最终预测值,标准差作为预测不确定性的指标。用户可以通过设定搜索时间控制AQ/DC中模型超参数优化覆盖的参数空间大小及耗费的时间。Glide使用LigPrep进行配体准备,其中Epik的pH参数设置为7.0 ± 1.0,立体异构最大数目设置为16,其他均为默认值。DOCK3.7使用ZINC15中的配体准备流程来优化。
如图1所示,作者首先随机抽取库中部分分子进行对接并将结果用于训练AQ/DC模型,接下来是一轮或多轮主动学习。主动学习指通过对整个虚拟库使用AQ/DC模型进行打分,然后使用一定的策略选出部分分子进行对接,将该结果与此前对接结果合并重新训练AQ/DC模型。该过程可重复多轮,最终得到的AQ/DC模型再对整个虚拟库打分作为最终的打分。由于整个数据集其实此前已经全部对接过,作者使用对接命中化合物的召回率来评价主动学习模型,对接命中化合物的标准为“对接打分排名前1万的化合物”。此外,所有的评价实验都有进行三次重复。
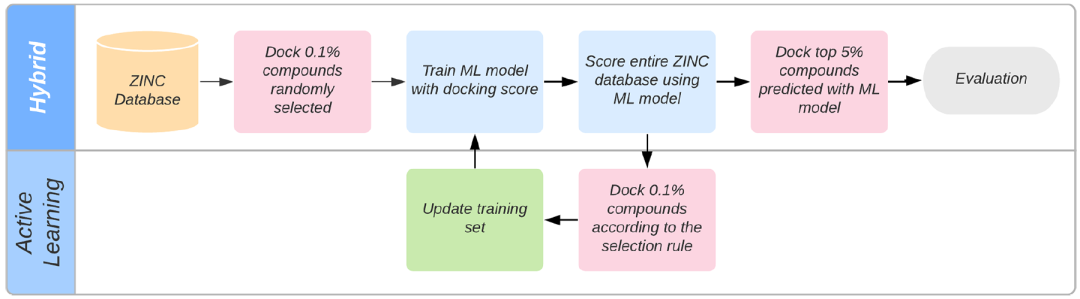
图 1. 主动学习的整体流程。来源:JCTC
用于虚拟筛选的文库在规模和化学多样性方面可能会有很大差异。作者评估了约 100 M 子集的 ZINC15 市售化合物和 Sigma-Aldrich Market-Select 库(约 8 M 化合物)。与普通的商业库(Sigma)相比,ZINC身为组合库的特性使它很适合主动学习技术,因为化合物类似物更多,在化学空间中分布更为稠密,可以对ML模型进行更有力的训练。
随机抽取虚拟库中部分分子,进行对接选取打分头部的分子,然后计算整个虚拟库与此头部分子的相似性并用该指标作为最终打分从高到低进行筛选,此为零模型(Null Model)。构建零模型的目的在于评估主动学习技术的排序能力是否仅仅是因为学习了结构间的相似性,还是来自于学习到的对接打分的知识。零模型使用Schrödinger Suite 中提供的 GPU 加速 FPsimGPU进行计算,该软件对计算过程进行了一定的近似。
AQ/DC 筛选召回虚拟hits和实验确证hits的能力
如图2展示了模型打分排序前x%分子中对应的hits覆盖率(召回率),图中模型均使用虚拟库0.1%大小的随机子集进行训练,可以看出AQ/DC模型对hits的召回能力很强。相对于 AmpC,基于 Glide SP 的模型表现比DOCK差,原因可能是这次筛选中DOCK的对接参数针对AmpC进行了优化,而Glide没有。
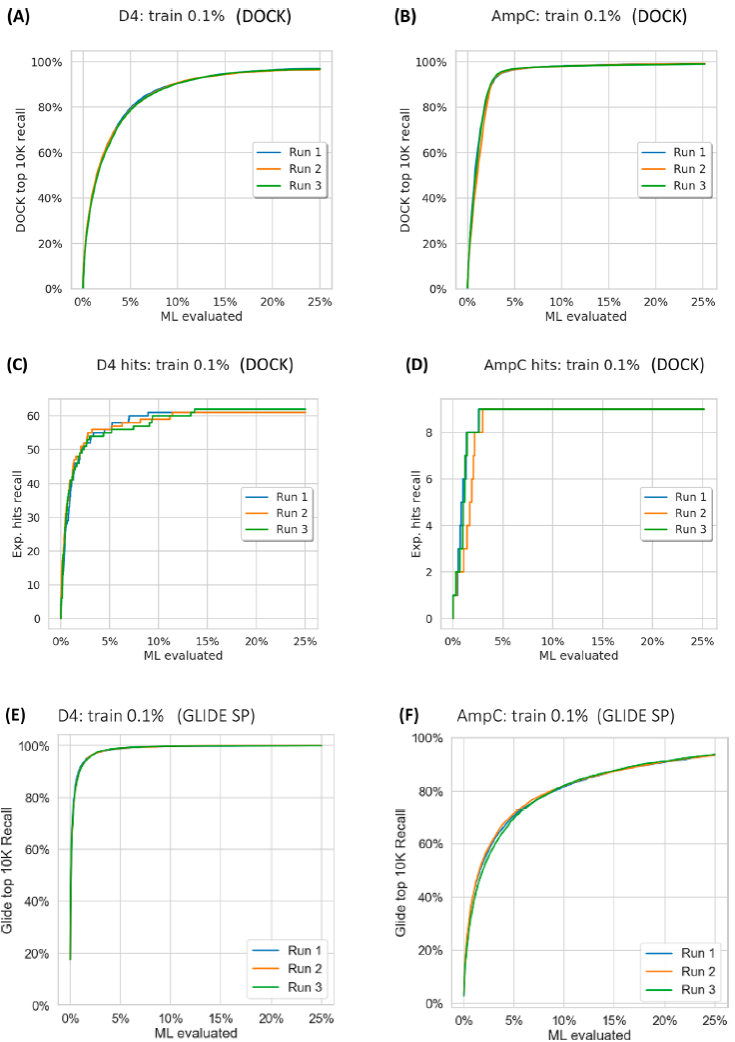
图 2. AQ/DC 模型针对前 10K 虚拟命中和实验命中的召回率。来源:JCTC
在训练时间不变的情况下,为了提高模型的性能,作者考虑以下两个影响因素:
(1)对接化合物训练集的大小:
作者测试了6组参数:0.01% 、0.02%、0.05%、0.1%、0.2% 和 0.5%,随着训练集大小的增加,虚拟命中化合物的召回得到改善。
(2)主动学习方案的迭代过程中采样方式对模型效果的影响:
分别对4种策略进行了测试a.ML 打分前 0.1%;b. ML打分不确定性的前 0.1%; c.ML 得分前 10% 中随机抽取得到0.1%;d. ML 得分前 5% 的化合物中不确定性的前 0.1%。其中b方案反而降低了召回率。令人惊讶的是,d方案在效果与对接成本中取得了最好的平衡。
主动学习方法可以节省近 30 万次对接计算,这些结论与是否使用 DOCK 或 Glide SP 无关。无论如何,拥有更大的数据集(0.5%)和使用主动学习来优化模型(主动学习 0.1% + 0.1%)都优于较小训练集的模型(图3)。在筛选的头部部分,性能的提升尤为明显。
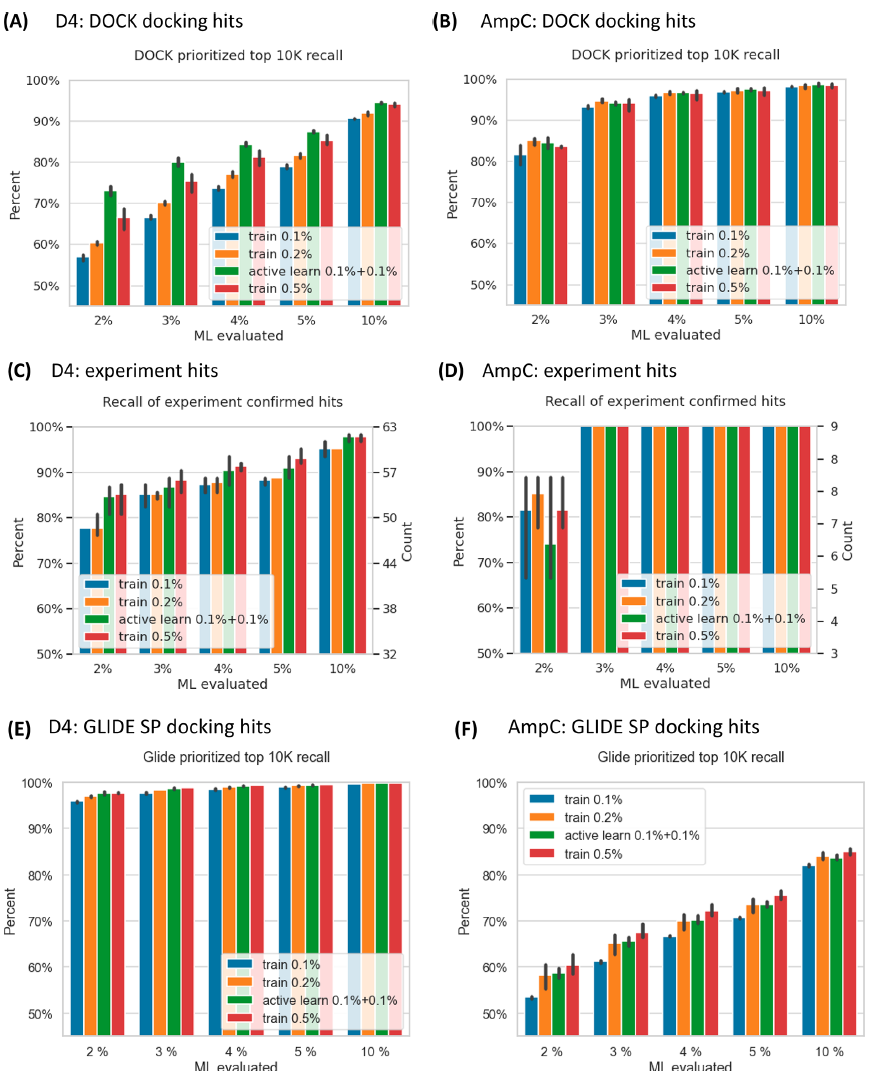
图 3. 三种采样方式对AD/DC 模型召回的影响。来源:JCTC
AQ/DC打分头部分子与对接命中分子的
化学多样性对比
作者使用 ECFP4 指纹对分子进行了聚类分析,仍然是主动学习的方式可以以低成本召回更多的同类命中分子(图4A)。作者还评测了 Schrodinger 的 Shape GPU 程序计算的 3D 形状相似度,结论类似。另一种评估化学多样性的方法是计算独特的 Bemis-Murcko 骨架的数目。发现必须通过重新对接AQ/DC打分的前2%-5%的分子后根据对接打分选出头部1万才能较好的召回对接命中分子的BM骨架(图4B)。这一结果突出了基于物理的方法在筛选中发挥的重要作用。在纯 ML 方法依赖于插值的情况下,基于物理的方法有更强的泛化能力。
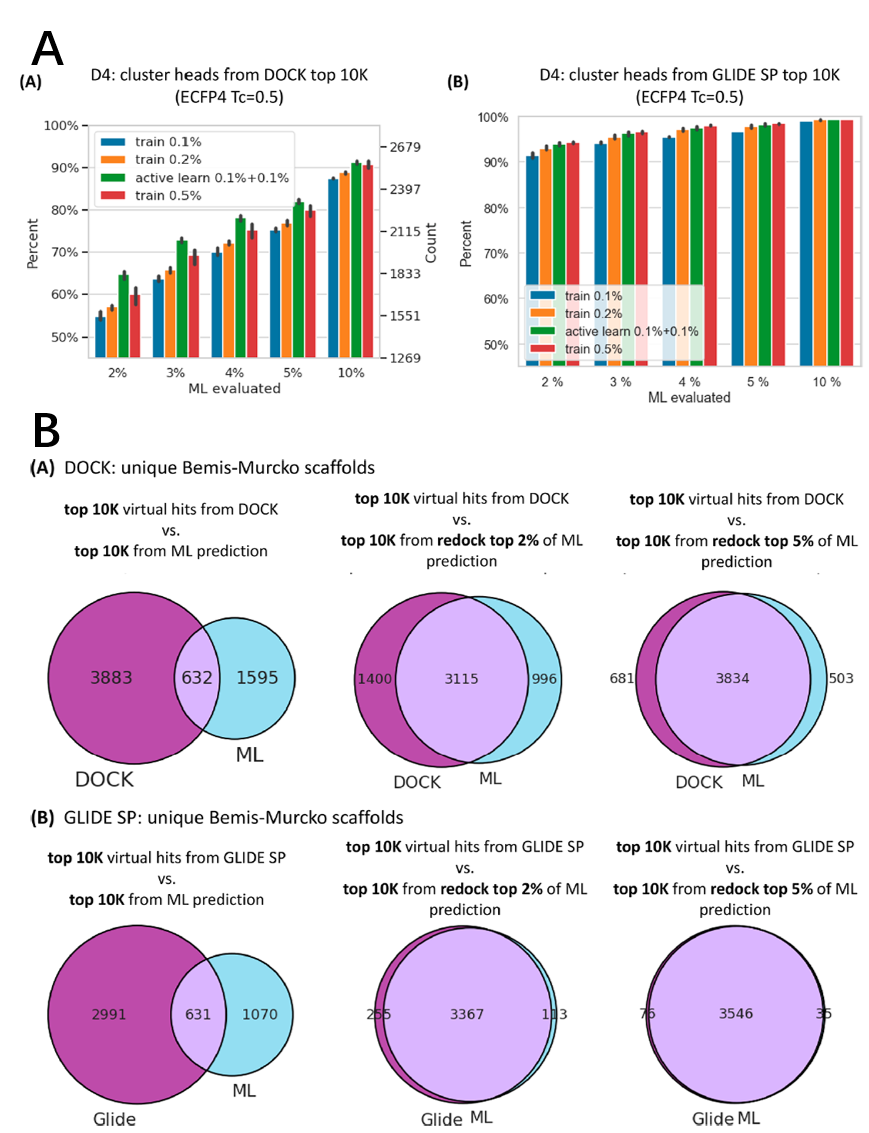
图 4. AQ/DC 模型与对接命中分子的多样性比较。来源:JCTC
至于主动学习迭代轮次对模型效果的影响,作者发现迭代轮次增加可以一定程度上提升模型的召回率,但这也意味着对接计算成本的增加,且在3轮迭代以后,效果提升非常有限。
作者随机选择了 0.2% 的虚筛库做对接,选择对接分数排名前 100、500 和 1000 的化合物作为探针分子去构建零模型,如图5所示。很明显,与零模型相比,AQ/DC模型不仅仅是学到了结构的相似性。
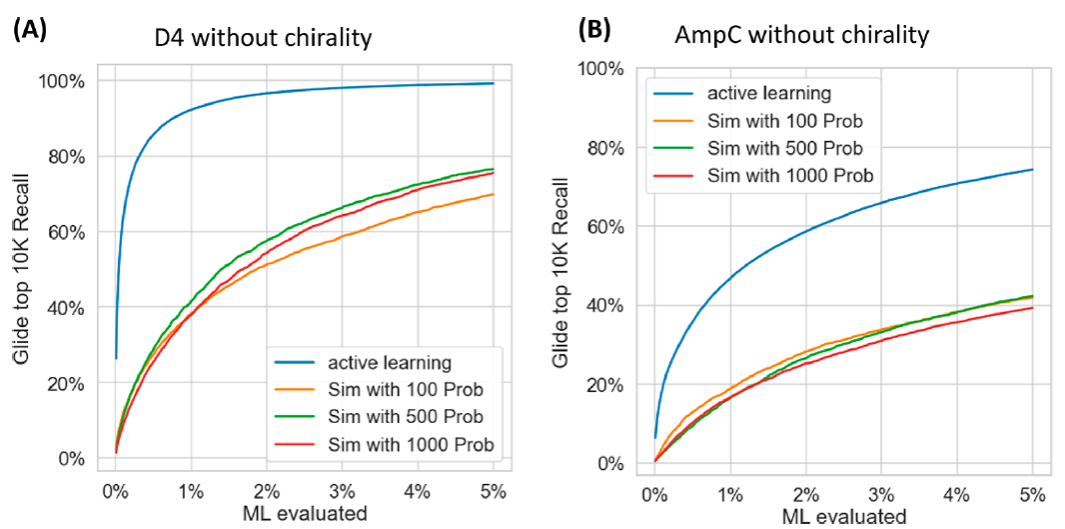
图 5. 主动学习模型与零模型效果对比。来源:JCTC
作者还在Sigma–Aldrich Market Select库上进行了类似的实验,但发现AQ/DC模型对对接命中化合物的召回能力明显差于ZINC上的表现。作者认为这是两种库的化合物的化学空间分布差异造成的。ZINC是一种组合库,因此在化学空间的局部会有更密集的分子占据,而普通商业库往往是通过收集,因此化学空间分布会更加离散。
为了验证这一理论,作者从与 Glide SP 的DRD4 的对接中选择了两个库中得分最高的 20 个化合物。我们通过提取与初始化合物具有特定化学相似性的一组最近邻,对这些虚拟命中化合物中的每一个进行了“命中化合物扩展”。相似度大于 0.5 的近邻分子按数值被分为三组:介于 0.5 和 0.6 之间,介于 0.6 和 0.7 之间以及> 0.7。由于两个库容量差异巨大,作者对数目进行了归一化。整体上介于0.5-0.6间的似像非像的近邻分子,ZINC比Sigma明显多了许多。太像和太不像的分子都难以提供给ML模型最多的可学习的信息,而正是介于0.5-0.6间的相似又不那么相似的近邻分子可以更加有力的训练模型,因此模型在ZINC库上表现更好。
作者在MT1体系上盲测主动学习流程的效果,结论与前面两个靶标的结果类似。
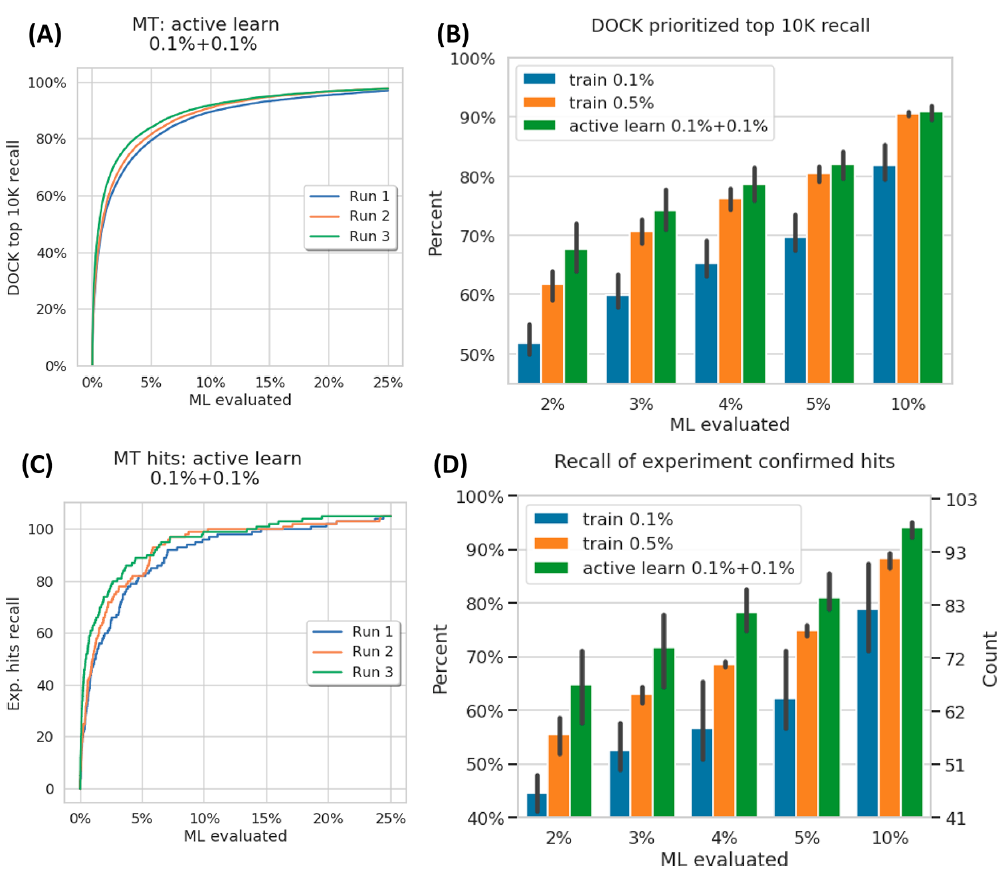
图 6. MT1 体系上 AQ/DC 模型的 效果。来源:JCTC
计算成本的比较
作者统计了DRD4体系使用不同虚筛方案的计算成本,如图7所示,除了以CPU时为单位的表示以外,作者还贴心的换算成了100个CPU+1个GPU的普通机器下的一个实际计算耗时(Wall Time)。可以看出,主动学习对计算时间的节省是巨大的。
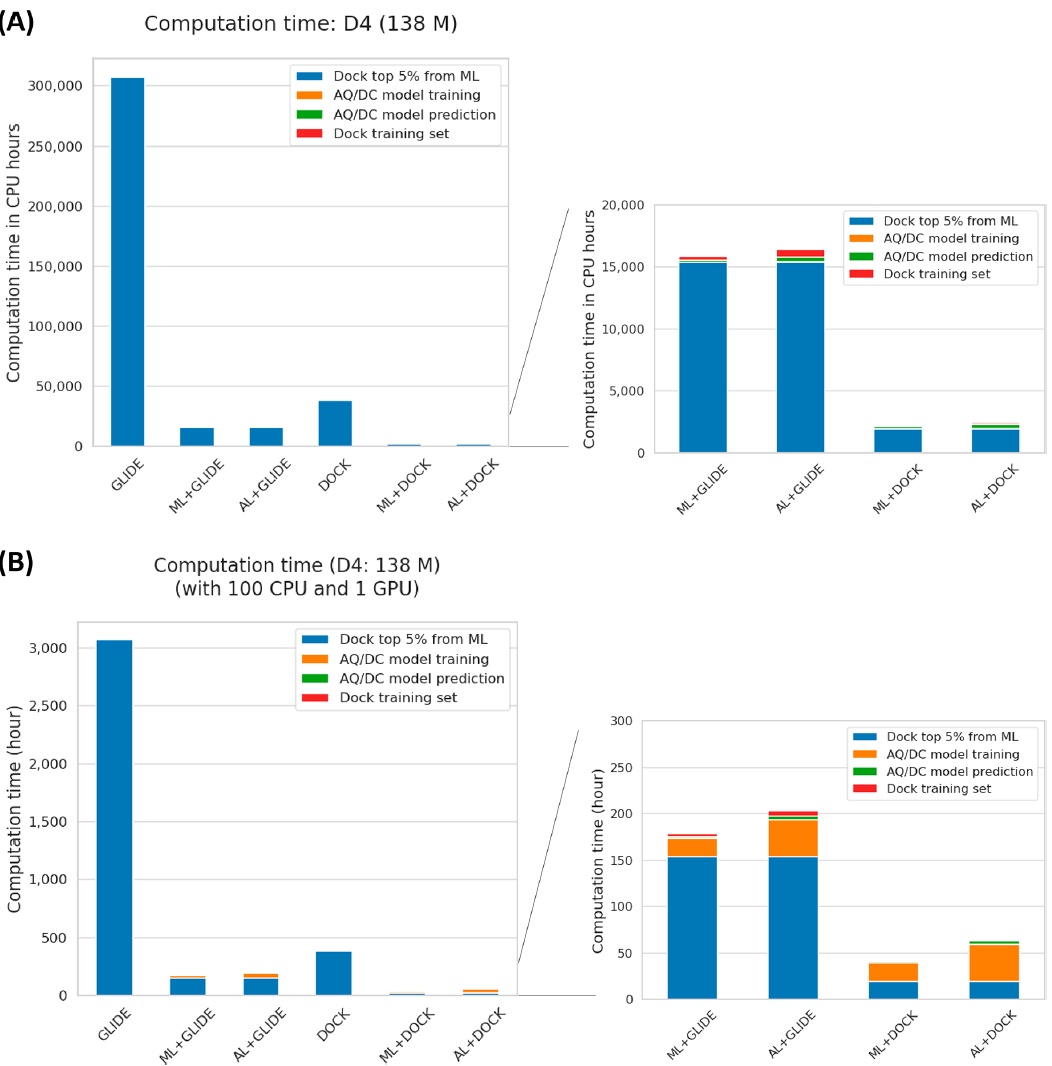
图7. DRD4 体系的 1.38 亿分子虚拟筛选计算成本。来源:JCTC
对接不仅输出一个打分,还输出结合构象,而主动学习模型无法输出结合构象。根据作者以上的结果),对AQ/DC打分头部分子进行对接(即重对接),不仅可以挽救ML打分和对接打分相关性没有那么好的缺点,还可以给出真正的对接打分和结合构象,供筛选后续的人工挑选使用。
结论
超大型虚拟筛选已经被证明能给出令人惊喜的结果,但由于巨大的计算成本导致目前仍没有太多研究使用此类方法。本文介绍了一种基于主动学习的对接虚筛方案,显著提高了虚筛的通量。该方法通过一种新颖的采样方式,平衡了虚筛中两个关键问题:1)找到打分最高的化合物,2)保证头部化合物的结构多样性。作者在DRD4和AmpC两个靶点上进行了回归性研究,并在MT1靶点上做了盲测,在头部分子中覆盖了传统对接80%的命中化合物,在头部5%的分子中覆盖了传统对接命中化合物90%的骨架,计算成本降低了14倍,使超大规模虚筛计算成本平民化。
参考文献
Ying Yang, Kun Yao, Matthew P. Repasky, Karl Leswing, Robert Abel, Brian K. Shoichet, and Steven V. Jerome Journal of Chemical Theory and Computation 2021 17 (11), 7106-7119