

世界上第一个基于网络的用于存储、管理和共享描述生物大分子间复杂相互作用的片段分子轨道计算数据集的公共数据库——FMO数据库。
背景介绍
了解蛋白质和候选药物之间的分子相互作用,对于设计有效的蛋白靶向药物是必不可少的。传统的蛋白质配体相互作用包括:氢键、静电相互作用,π-相互作用和疏水相互作用,这些相互作用通常可用分子力学力场和经验规则预定义的公式来描述。然而,自20世纪90年代以来,人们发现了具有量子力学(QM)作用特征的CH/π、硫原子和卤素原子相互作用。但由于目前确定蛋白质-配体相互作用的公式,很大程度上依赖于分子力场,尚未充分考虑QM计算数据,因此,可能存在一些潜在的重要相互作用尚未被发现或定义不清的情况。为了成功检测所有相互作用并准确预测相互作用能值,必须进行从头计算QM。
当进行生物大分子,如蛋白质配体配合物的从头计算QM时,通常需要巨大的计算成本。为了解决这一问题,研究人员开发了片段分子轨道(fragment molecular orbital, FMO)方法,该方法采用带片段的多体展式技术,降低了计算成本,使研究人员能够高效地进行从头算QM计算。FMO方法的另一个优点是,可以计算片段-片段相互作用能值,即片段间相互作用能(IFIE)和对相互作用能分解分析(PIEDA)值,以及静电(ES)、色散相互作用(DI)、交换排斥(EX)和高阶混合项电荷转移(CT+mix)能量对这些值的相对贡献,这些数值对于详细分析蛋白-配体和蛋白-蛋白相互作用是有用的。
目前,虽然许多独立的研究小组都进行了FMO计算和分析,但主要是在药物发现和结构生物学领域,而且计算出来的FMO数据通常在研究结束后不会面向公众公布。近年来,计算机性能的提高使得进行大量的FMO计算成为可能,预计在不久的将来会积累大量的FMO数据。
主要内容
来自日本东北大学和星薬科大学的Kaori Fukuzawa以及日本理化研究所的Daisuke Takaya & Chiduru Watanabe & Teruki Honma等人开发了FMO数据库(FMODB),这是世界上第一个用于存储、管理和共享描述生物大分子之间复杂相互作用的FMO计算数据集的公共数据库。数据库中的每一项都包含了有关背景数据,如数据是如何编译的、每个分子系统的总能量和片段间相互作用能(IFIE)以及对相互作用能分解分析(PIEDA)值。目前,该数据库包含了13600多套FMO计算数据集,并在前端实现了全面的搜索功能,同时,介绍了靶向蛋白的筛选、实验结构的预处理、数据库的构建以及数据库前端的详细内容。随后,研究者通过比较FMO方法和分子力学方法得到的氢键、离子对和XH/π相互作用的IFIE值分布,证明了FMODB的应用有效性。通过比较,数据的统计分析为三种类型的相互作用提供了标准参考值,这将有助于确定给定系统中的每一种相互作用相对于FMODB数据中包含的相互作用是强是弱。最后,研究者演示了使用数据库来检查卤素原子对人组织蛋白酶L及其抑制剂之间的结合亲和力的贡献。研究发现,由PIEDA导出的静电项与含卤素的组织蛋白酶L抑制剂的结合亲和性密切相关,表明了定量分析卤素相互作用的QM计算的重要性。相关的研究成果以“FMODB: The World’s First Database of Quantum Mechanical Calculations for Biomacromolecules Based on the Fragment Molecular Orbital Method”为题发布在国际著名期刊Journal of Chemical Information and Modeling上。
数据库公开访问地址
https://drugdesign.riken.jp/FMODB/
网站首页:
FMODB的构造
在本研究中,FMO计算的结构包括了X射线晶体学、核磁共振、低温电子显微镜、M模拟快照以及建模结构获得的实验结构。FMO计算前的预处理,可以通过专用软件手动或自动进行。在这里,由FMODD或BINDS的参与研究人员进行手工预处理,并根据每个研究人员打算如何使用FMO计算数据集选择特定的预处理方法(例如,结合亲和预测或分子识别分析)。作为手动预处理的替代方案,FMODD的开发工作组启动了一种自动预处理方法,该方法被称为Auto-FMO协议(图1)。
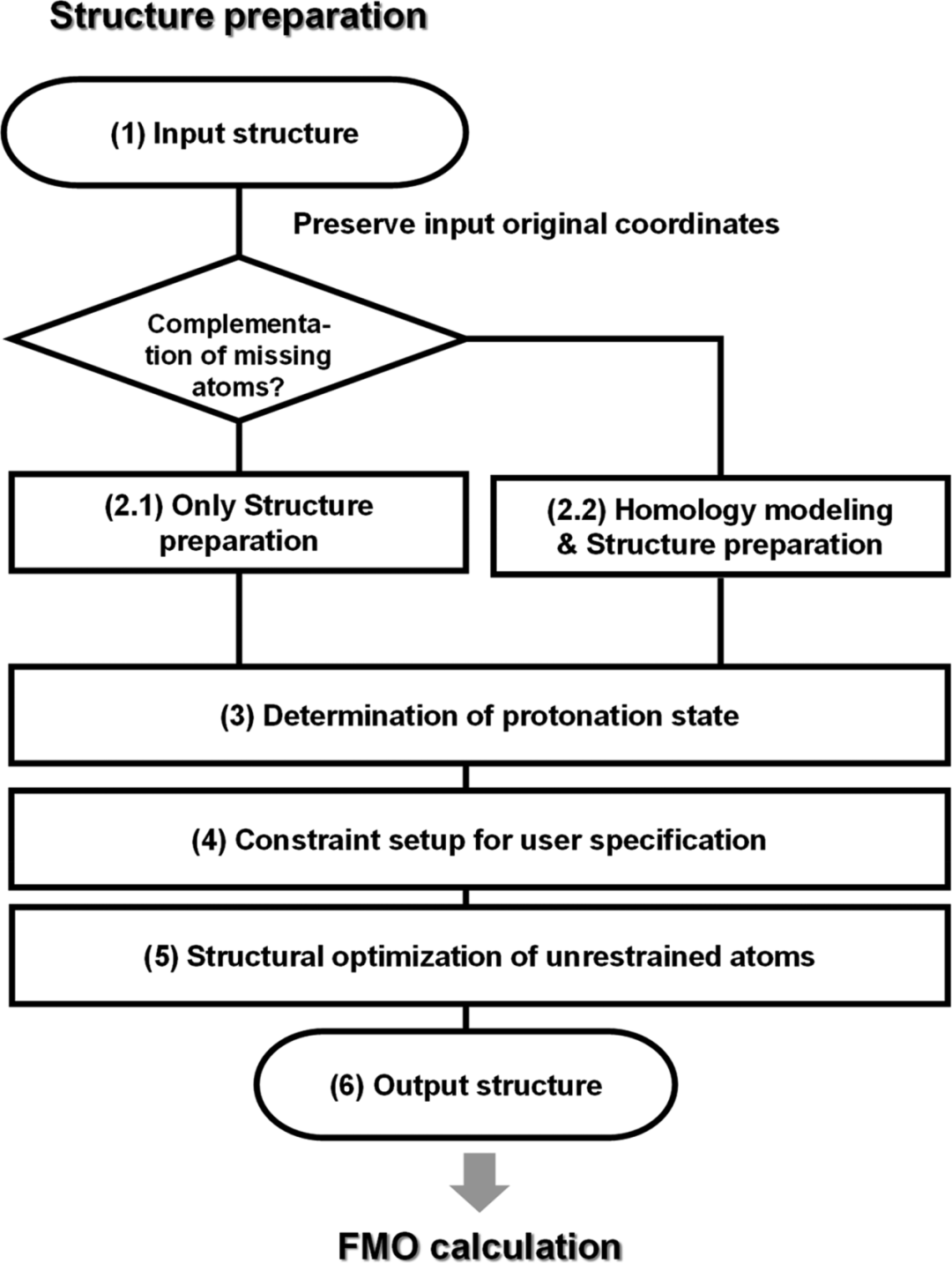
图 1. 自动FMO协议对片段分子轨道(FMO)计算结构进行预处理的工作流程。图片来源于JCIM
研究者构建了一个基于web的前端,允许用户可以轻松地查看每个条目的分子结构、注释以及IFIE和PIEDA数据。为了证明FMODB的有效性,研究者进行了统计分析,定义了IFIE和PIEDA的几个分子间和分子内相互作用的标准参考值。
氢键、离子对和π-轨道相互作用的检测结果如下:
首先,利用MOE中的prolig_Calculate函数,通过几何定义的相互作用指纹(IFP)识别片段对间和分子内的相互作用,该指纹根据原子类型、原子间距离和取向角检测不同的相互作用。
其次,通过几何IFP分析,定义原子对相互作用的距离如下:图2显示了在ERα和17β-雌二醇复合物中检测到的相互作用示例(PDB ID: 1ERE;FMODB ID: 5 p4np)。
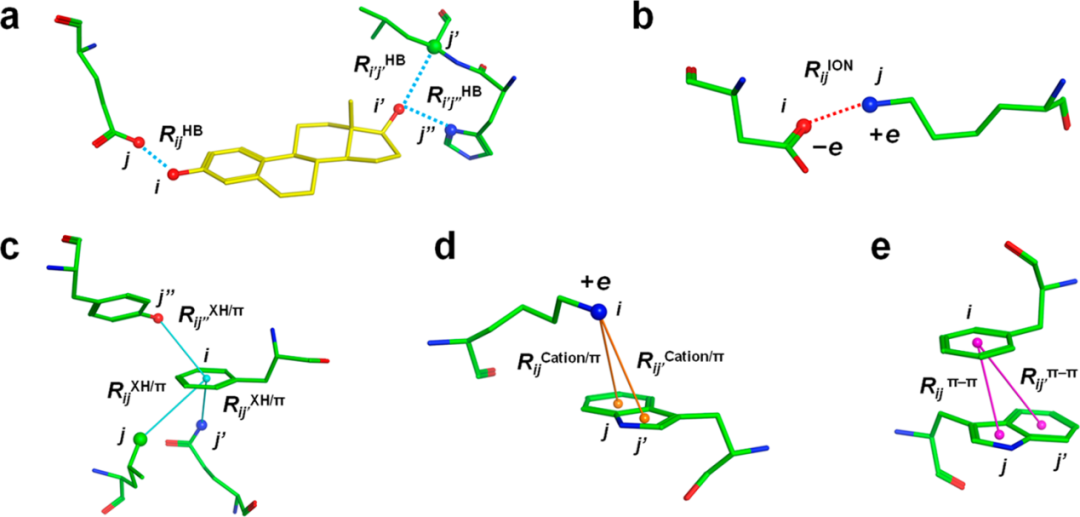
图 2. 在雌激素受体α (ERα)和17β-雌二醇(PDB ID: 1ERE;基于几何相互作用指纹分析的FMODB ID:5P4NP)。图片来源于JCIM
最后,为了检验每一种相互作用的能量分布,研究者通过IFP检测到的原子与FMO计算中构建片段对的原子之间的对应关系,将几何IFP分配给基于FMO-和MM-的片段对相互作用能。
截至2020年9月9日,共对13667组蛋白质靶点进行了FMO计算,包括1468个独特的PDB条目;13239次FMO计算在Auto-FMO协议下进行(表1)。
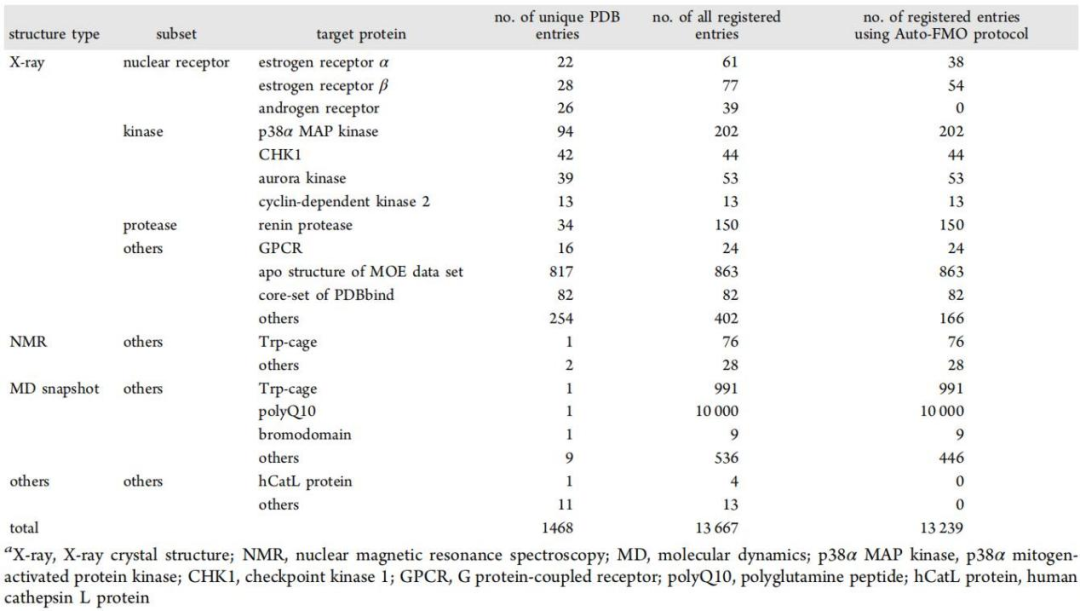
表 1. FMO数据库中的数据集列表。表格来源于JCIM
分子间和分子内相互作用的统计分析
通过如图3所示的过程选择片段对的数据集进行评估。研究者从1486个X射线晶体结构中选取了912932个片段对,它们的距离在vdW半径的2.0倍以内(不采用二聚体ES近似),并通过Auto-FMO协议进行计算。最终获得了612477对含有氢键相互作用的片段对,15801组离子对相互作用,102720组 XH /π相互作用,3406组阳离子/π相互作用,394组π -π相互作用(图2)。
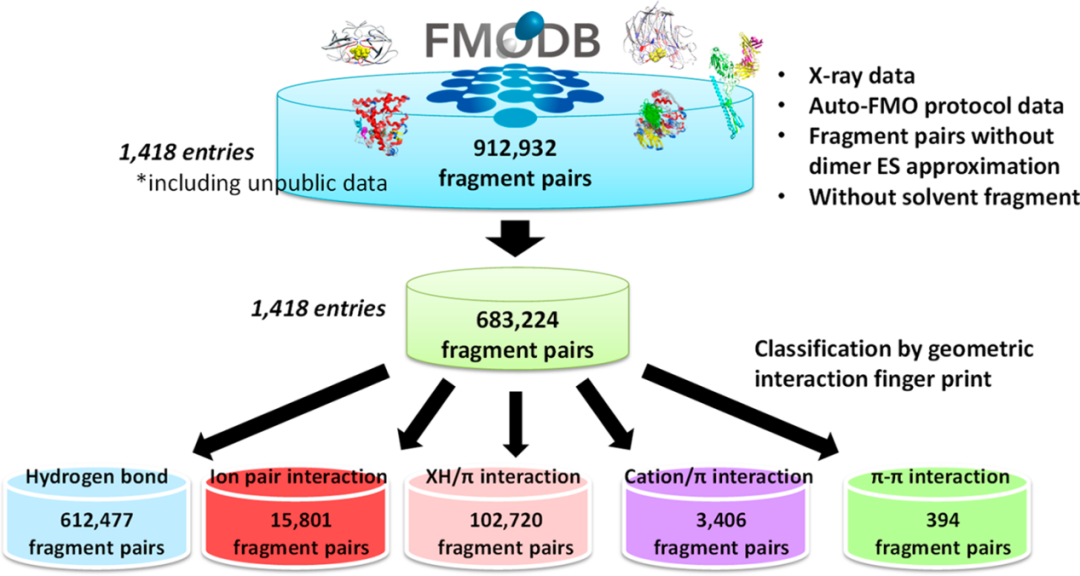
图3. 选择具有指定几何相互作用指纹的片段对,以及对分子间和分子内相互作用的能量分解和片段间相互作用能的统计分析。图来源于JCIM
hCatL与一系列卤化抑制剂的相互作用分析
图4显示了hCatL蛋白与氢化或卤化配体(X = H, F, Cl和Br)之间的相互作用示意图。将hCatL蛋白分解为单个氨基酸残基。配体在Cys25处与蛋白质共价结合,从蛋白质中分离出来,分成三个片段。配体中的卤素原子(X)通过卤素键或氢键与Gly61主链C短键长的O原子相互作用,作用距离为3.31 Å。单个氨基酸残基在主链上的Cα和C短键长的O之间被剪切,而不是在sp2杂化的酰胺键上(图4,左上)。
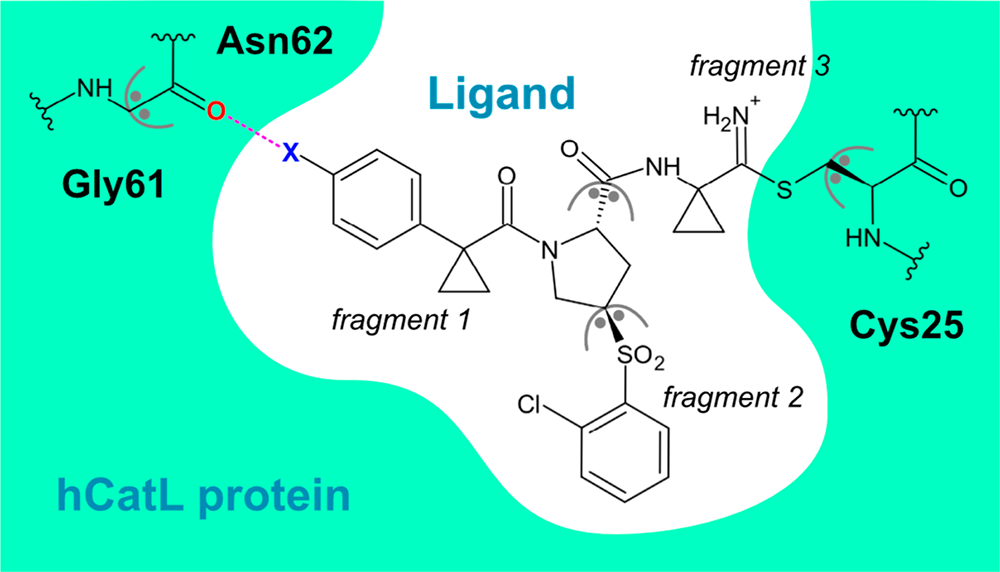
图4. 人组织蛋白酶L蛋白(hCatL)的Gly61(主链的O原子被认为属于Asn62片段)与氢化(X = H)或卤化(X = F、Cl或Br)配体之间的相互作用示意图。图来源于JCIM
图5a总结了hCatL的Asn62片段与各种卤化配体(X = F, Cl或Br)或氢化配体(X = H)相互作用的FMO-IFIE数据。由于CH-O氢键的形成,氢化产物具有吸引力的相互作用能(IFIE:−2.87 kcal/mol)。另一方面,在原子对距离为3.50 Å的情况下,由于O和F原子负电荷之间的静电斥力,氟化化合物具有+1.19 kcal/mol的排斥能。尽管有负极性的卤原子和氟原子,氯代和溴代配体依然表现出稳定的相互作用能(IFIE:−2.12和−3.16 kcal/mol)。图5a中的ES组分清楚地表征了这些相互作用的稳定性,表明存在σ-空穴。
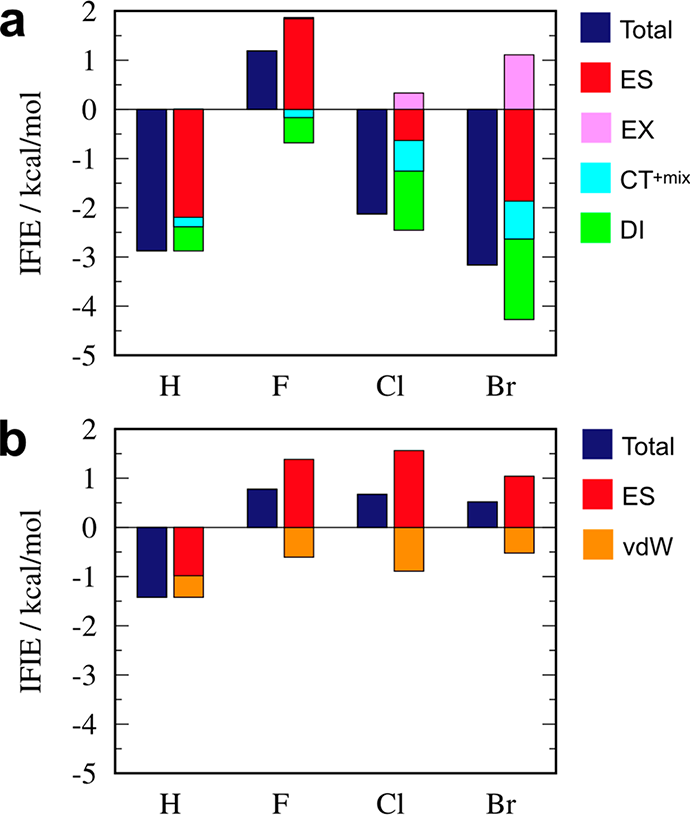
图5. 人类组织蛋白酶L蛋白Asn62片段与氢化或卤化配体相互作用的片段间相互作用能(IFIE)及其分解(PIEDA)值,由(a)片段分子轨道或(b)分子力学计算确定。图来源于JCIM
MM-IFIE值为静电和范德华相互作用的总和,使用了MOE中的Amber10:EHT力场(图5b)。在FMO数据中观察到的σ-空穴的稳定性没有被MM计算捕获,所有卤代化合物都显示出明显的静电斥力。这一发现证明了FMO方法优于MM方法的一个优点。
氢化物(X = H)通过静电作用与Gly61的C=O产生相当大的相互作用,作用距离为3.68 Å;该配体的IC50值为0.29 μM,其活性远低于计算结果。
总结
本文报道了世界上第一个基于FMO方法的生物大分子量子力学计算数据库FMODB的开发。FMODB于2019年2月20日启动,包括一个用于搜索和浏览数据库的web前端,最初发布的数据包含了2000多个蛋白质结构的总能量、IFIE和PIEDA数据。利用FMODB中目前记录的数据,研究者比较了FMO方法和MM方法计算的氢键、离子对和XH/π相互作用的能量分布。
未来,FMODB可以帮助结构生物学家和药物化学家等实验研究者定量地理解分子识别,并通过简单的QM数据来改进药物设计。研究者们将继续更新PDB结构的FMODB数据。除了蛋白配体配合物,研究者还计划涵盖质子泵抑制剂和核酸的计算数据。
参考文献
Takaya, Daisuke, et al. “FMODB: The World’s First Database of Quantum Mechanical Calculations for Biomacromolecules Based on the Fragment Molecular Orbital Method.” J. Chem. Inf. Model. 2021, 61, 2, 777–794. https://doi.org/10.1021/acs.jcim.0c01062