

CS-Annotate的核心是一种多任务深度学习模型的运用,可以同时从化学位移指纹中对溶剂暴露、碱基堆积和配对状态以及单个RNA残基构象进行分类。
背景介绍
NMR化学位移对分子中的局部电子环境很敏感,因此它们可以作为结构指纹。从化学位移中直接提取生物分子的结构特性一直是结构生物学的一个长期目标,但从化学位移数据中直接提取RNA结构特性的类似方法受到的关注较少。
本文作者训练了一个单一、多任务分类器,将RNA残基中不可交换的1H和13C化学位移作为输入,输出其溶剂暴露、相邻堆积、不相邻堆积或碱基配对的概率,这些是六种糖褶状态之一或是syn或anti构象。通过名为CS-Annotate的web服务器来公开多任务分类器,该服务器使用化学位移形成分类评分来注释RNA中单个残留物的结构属性。文章简要描述了如何训练和测试分类器,并演示了模型在RNA系统中的应用。
CS-Annotate在线访问地址
https://smaltr.org/
演示视频地址
https://youtu.be/ZfpxLe46SvE
网站首页:
图1是对11个任务进行训练的渐进神经网络(PNN)模型的演示,每个任务都有两个隐藏层。用于训练多任务分类器的数据集由104个RNA的化学位移和结构数据组成。化学位移及原子结构信息分别从BMRB和PDB得到。使用 MC-Annotate 确定每个 RNA除溶剂暴露外的所有结构特性。用POPS 计算残基溶剂可及表面积 (SASAs)。如果残基的 SASA 大于整个训练集的 SASA 值的平均值加上标准差 (SD) 的一半,则将残基定义为溶剂暴露。然后将与单个残基和相邻残基相关的化学位移数据与其注释特性相结合,形成化学位移结构数据集。训练集有 3068 个样本,测试集有 129 个样本。
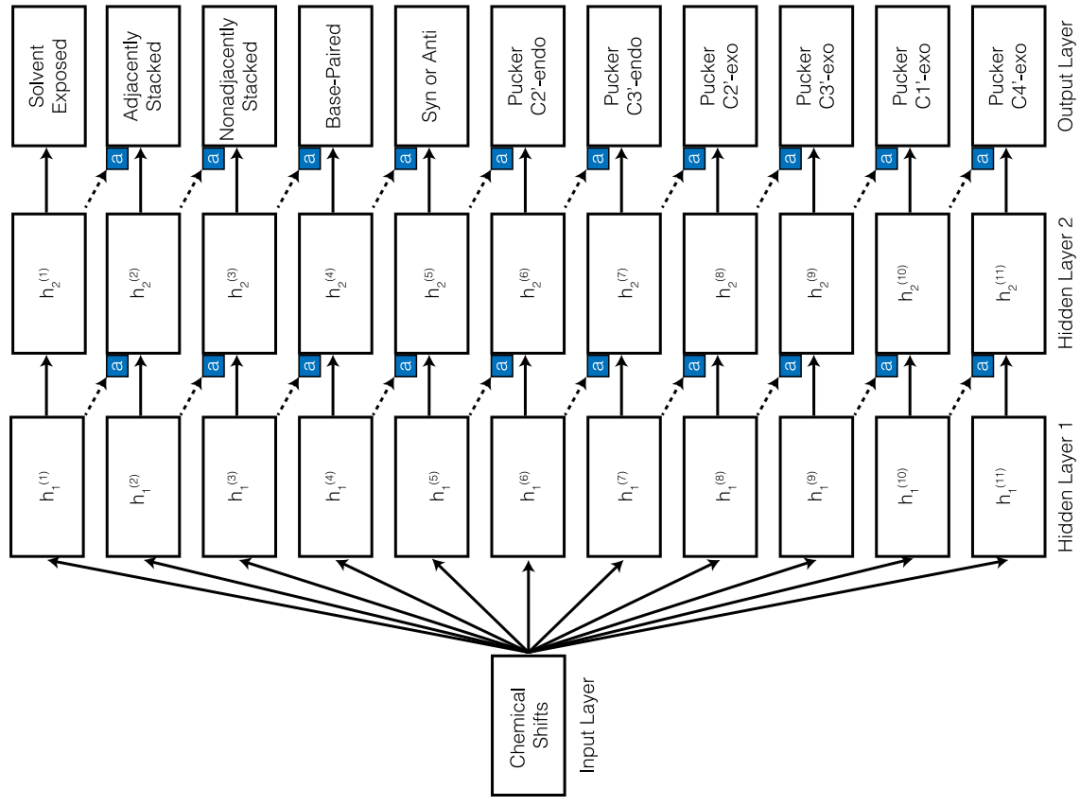
图1 渐进神经网络(PNN)模型演示。图片来源:JCIM
模型结果
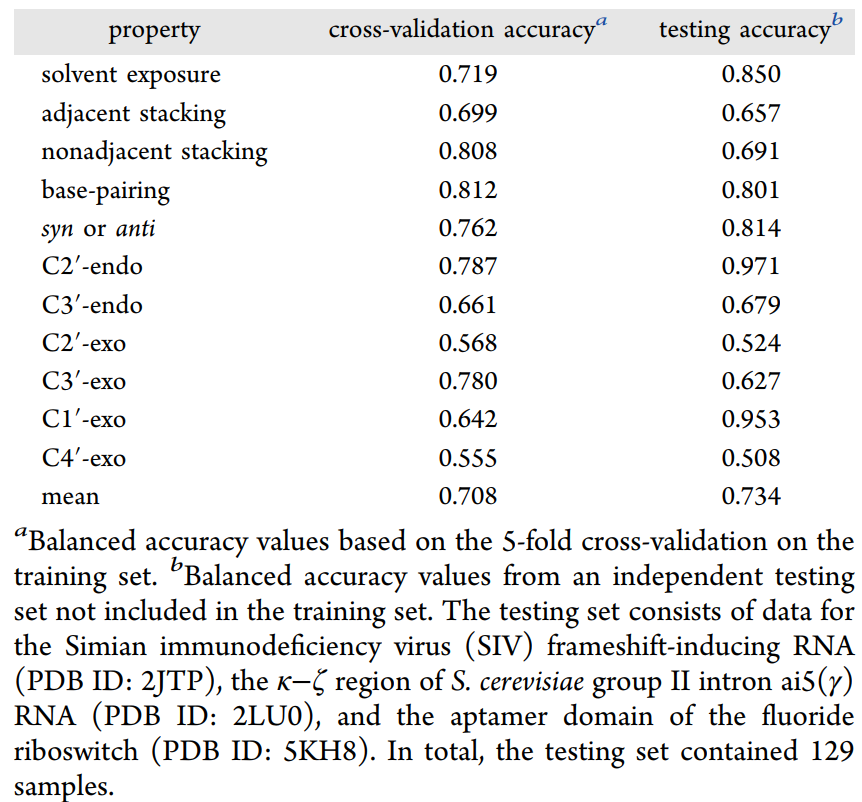
为了评估PNN是否能从化学位移特征中同时注释RNA中单个残基的11个结构属性,作者对训练集数据进行了五折交叉验证(表1)。
11个任务的平衡精度(balance accuracy)从0.555到0.812不等,均值为0.708,强于多个基线模型。使用整个训练集对PNN进行再训练,并在三个测试RNA的残基的测试集中验证其性能。分类器的性能在训练集上的表现符合交叉验证结果:平衡精度介于0.508和0.971之间,均值为0.734。糖折叠模式的预测精度比其他属性的低。
表1 渐进多任务分类器的平衡精度。表格来源:JCIM
在氟化核糖开关上的应用
图2a所示的是作者的多任务分类器在应用于氟化物核糖体开关的适配体结构域时得到的注释图。与NMR结构相比,在10个溶剂暴露概率最高的残基中有7个确实是高度暴露(其残基SASA>200 Å2) (图2b)。除了G4之外,所有被预测为邻接堆积的残基都得到了正确的分类(图2c)。然而,一些残基(例如,U12、C13、A21、C22、U35、A37、C44、U45和U47)被误归类为不邻接堆积,表明该分类器对该任务的敏感性低于特异性(表1)。出乎意料的是,该分类器还能注释几个非邻接残基。例如,G4、A5、G10、A17、G23、A36、G39都表现出较高的非相邻堆积分类得分,也确实参与了非相邻堆积相互作用(图2d)。
对于单个残基的碱基配对状态,U6、A46、U47被误分为碱基配对,U12、C41、A42、C43、C44被误分为未配对(图2e)。总的来说,该多任务分类器找回了氟化核糖开关中大多数残基的碱基对状态。氟核糖开关中大部分残基的糖苷扭转(χ)处于anti构象,syn构象的G30、high-anti构象的A19、U28和A37除外。有趣的是,这些是分类器预测的最有可能是syn的四个残基(图2f)。在糖褶方面,除A17、U18、A19、U28、C29和A37六个残基外,氟核糖开关中大部分残基为3’-endo糖褶构象。这6个残基,以及A20、A21和C22,被多任务分类器预测为最不可能(p<0.10)处于3’-endo构象的残基(图2a)。根据相对糖褶分类分数,A17和U18被正确识别为更有可能为2’-endo(图2g),尽管它们的绝对分类分数较低(p∼0.31;图2a)。
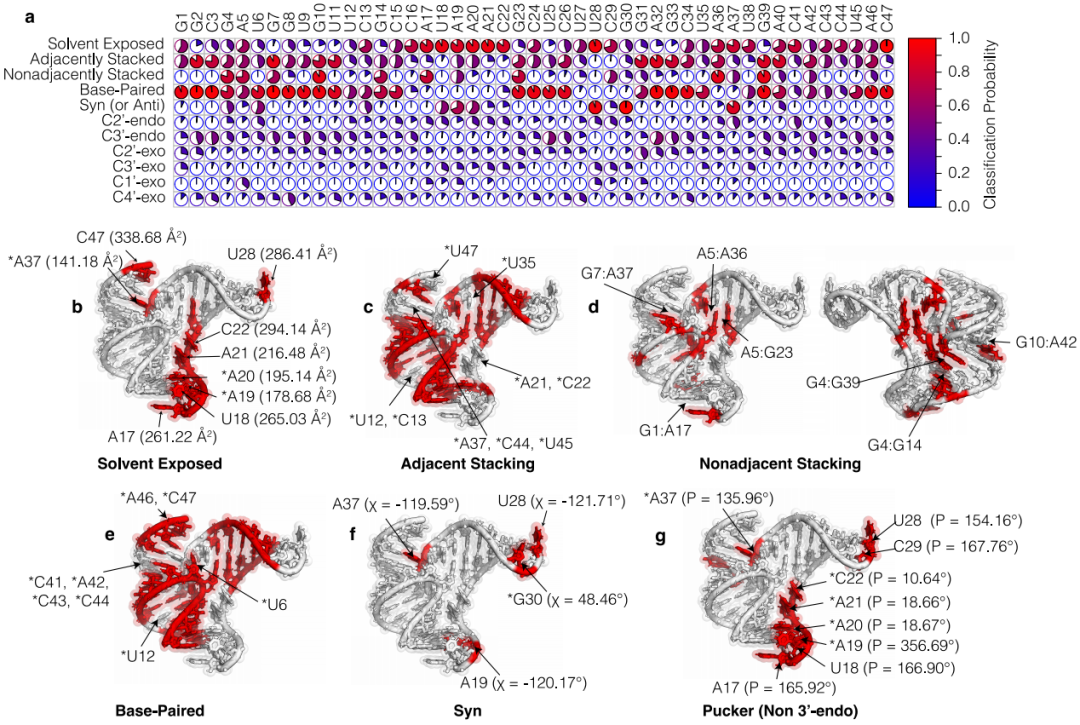
图2氟化核糖开关的适配体结构域化学位移生成的注释。图片来源:JCIM
CS-Annotate的限制
正如在上述氟核糖开关中的应用,作者训练的多任务分类器可以基于一组指定的化学位移来注释RNA中的多个结构特性。因此,他们将模型部署到一个称为CS-Annotate的工具中。CS-Annotate将RNA分配的化学位移以格式化的文本文件输入到模型中,然后将输入的化学位移传递给分类器,然后返回一个图形注释图(图2a)和一个包含残基预测的文本文件。
CS-Annotate的一个限制是,因为训练分类器使用化学位移i−3、i −2、i−1、i、i+1、i+2和i+3来估计残基的性质,它的预测只有在无分配间隙的情况下才有意义,即对RNA中每个残基中一些不可交换的1H和1C原子有指定的化学位移。
为了与基于结构的注释工具如MC-Annotate所使用的糖褶分类方案一致,CS-Annotate输出了许多糖褶状态预测。然而,在某些情况下,用户可能会对更粗粒度的预测很感兴趣。为了实现这一点,用户可以对CS-Annotate的预测进行粗粒化,例如,将残基分类为C3-endo。
最后,作者注意到PNN分类器是与顺序相关的。其对目标顺序的选择[即,哪个任务是第一个,哪个任务是第二个]是任意的。鉴于多任务分类器的11个目标的组合有3900万种。因此,他们并没有探索这个巨大的“超参数”空间。
尽管有这些警告,作者设想CS-Annotate可以作为一种工具,在研究的主要焦点不是确定3D结构的情况下(例如,在进行机制研究时),或者在无法获得通常用于通过NMR解析RNA结构的数据的情况下(例如,当研究RNA的瞬态时),从NMR化学转移实例生成可靠的结构假设。
结论
作者训练了一个多任务分类器,它从一组分配的化学位移中分类RNA中的11个残基结构属性,并将该分类器置于CS-Annotate工具中。使用CS-Annotate,用户将能预测溶剂暴露、相邻/非相邻的堆积相互作用、碱基对状态、糖苷取向和糖褶状态。CS-Annotate将是RNA详细结构分析的有用工具,并与其他预测建模工具一起,用于检测RNA中的化学转移结构关系。
参考文献
Zhang K, Abdallah K, Ajmera P, Finos K, Looka A, Mekhael J, Frank AT. CS-Annotate: A Tool for Using NMR Chemical Shifts to Annotate RNA Structure. J Chem Inf Model. 2021 Apr 26;61(4):1545-1549. doi: 10.1021/acs.jcim.1c00006. Epub 2021 Apr 2. PMID: 33797909.