

一种可用于研究药物锂对中枢蛋白GSK-3β相互作用网络影响的新方法,同时计算了GSK-3β在锂离子或钠离子存在下与其分子伴侣的亲和力。
背景介绍
自20世纪80年代蛋白相互作用(PPIs)界面结构被揭示以来,PPIs就一直受到科学家的关注。PPIs形成一个相互作用的网络,其中蛋白形成节点,PPIs形成边。这种蛋白相互作用网络(PIN)可以调节细胞对胞内外扰动的响应,并维持生命体系的稳定。然而突变引起的结构变化或蛋白水平的改变,可能会扰乱PIN从而导致各种疾病的发展。迄今为止,小分子已被证明可以靶向特定的蛋白-蛋白界面和特定的蛋白质结合贴片 (PBP),并且已经表明靶向单个 PPI 可以影响细胞信号传导并可以传播 PIN。
在PIN中,枢纽蛋白有大量的相互作用伴侣。这些蛋白质可能含有大量的PBPs。每个蛋白内部都存在一个残基间相互作用网络,即蛋白质接触网络(protein contact network, PCN)。一个小分子与一个蛋白质的相互作用,可能会影响整个PCN,并影响该蛋白质的多个PBP。
GSK-3(糖原合成酶激酶-3)蛋白是一种高度保守的丝氨酸/苏氨酸激酶,由不同的激酶调节,影响100多种底物,并参与许多细胞信号通路;此外,GSK-3β亚型被认为是PIN中的一个枢纽。
锂作为抗惊厥和抗躁狂药物的历史,可以追溯到19世纪70年代。锂在20世纪70年代早期被FDA批准用于治疗双相情感障碍(躁狂抑郁症)。这种离子直接或间接地抑制GSK-3的活性。锂可以增加GSK-3β的抑制性磷酸化,它还可能通过与镁离子竞争来直接抑制GSK-3β的催化活性。
主要内容
本文中,来自伊朗高级基础科学研究所的Maryam Rouhani等人利用一种新的方法,研究了锂药对GSK-3β作为枢纽蛋白相互作用网络的影响,并计算了GSK-3β在锂或钠离子存在下与其分子伴侣的亲和力。为此,研究者利用自适应回火分子动力学模拟,在锂离子或钠离子存在的情况下,得到了GSK-3β蛋白结构。研究者确定了GSK-3β对其分子伴侣的PBP,最后利用基于单体的方法,计算了各结合贴片对相关分子伴侣的亲和力。此外,通过比较在LiCl和NaCl存在的MD模拟中GSK-3β的结构动力学,研究者提出了锂抑制GSK-3β的新机制。相关的研究成果以“Effect of Lithium Drug on Binding Affinities of Glycogen Synthase Kinase-3β to Its Network Partners:A New Computational Approach”为题发布在国际著名期刊Journal of Chemical Information and Modeling上。
自适应回火分子动力学(AT-MD)模拟
研究者利用20 ns常规MD模拟对GSK-3β结构空间进行了采样,观察到结构在−1.6×105 ~−1.4×105 kcal/mol势阱中波动。为丰富GSK-3β的结构集合,研究者建立了多个不同的起始结构和不同的模拟时间片段的AT-MD模拟(图1)。同时,研究者通过结合不同的AT-MD模拟片段,在LiCl或NaCl存在下建立了GSK-3β结构的集合。
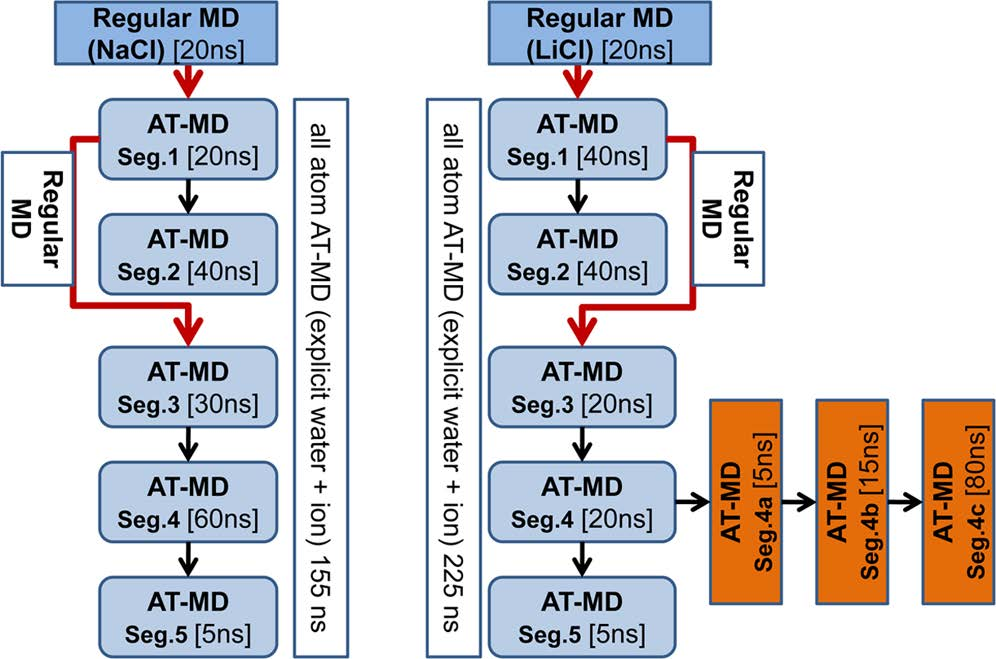
图 1. 计划生成丰富和不同的结构组合。图片来源于JCIM
通过在二维密度图上绘制GSK-3β在锂离子和钠离子存在下蛋白质旋转半径和%疏水可及表面积(ASA)变化的势能,研究者发现锂离子引起了结构整体稳定性的巨大变化(图2)。在锂的存在下,有一个稳定的结构集合(图2下图中的深蓝色),与开始的结构相比有点膨胀(Rg = 23 Å)。稳定体系的山谷(图2下图中的蓝色)周围,环绕着明显的不稳定结构峰(图2下图中的黄色),具有较高的可及疏水表面。这种清晰的整体在钠的存在下显得很模糊。下图所示的二维密度图显示了Li+对蛋白质稳定性的影响。
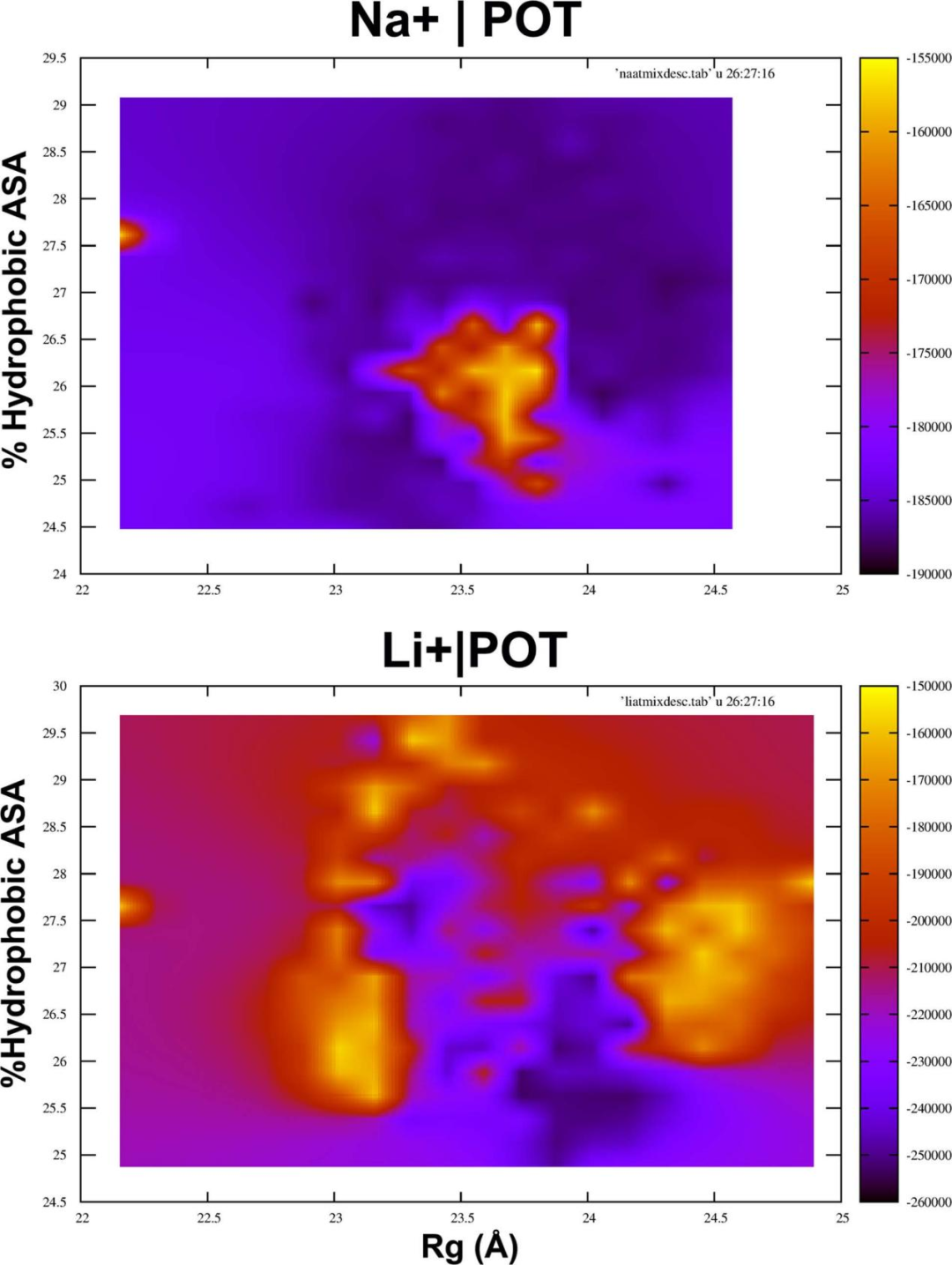
图 2. 三维密度与稳定状态特性。图片来源于JCIM
基本动力学分析(EDA)与离子分布
为了捕捉锂离子对GSK-3β可能的运动影响,研究者向EDA提供了用AT -MD生成的结构集合;EDA结果显示,在钠离子存在下,GSK-3β在C端有较高的运动水平(图3)。从方形波动图中可以看出,蛋白质与锂离子的相互作用阻碍了运动。GSK-3β动态变化的结果之一是活性位点开放大小的变化。
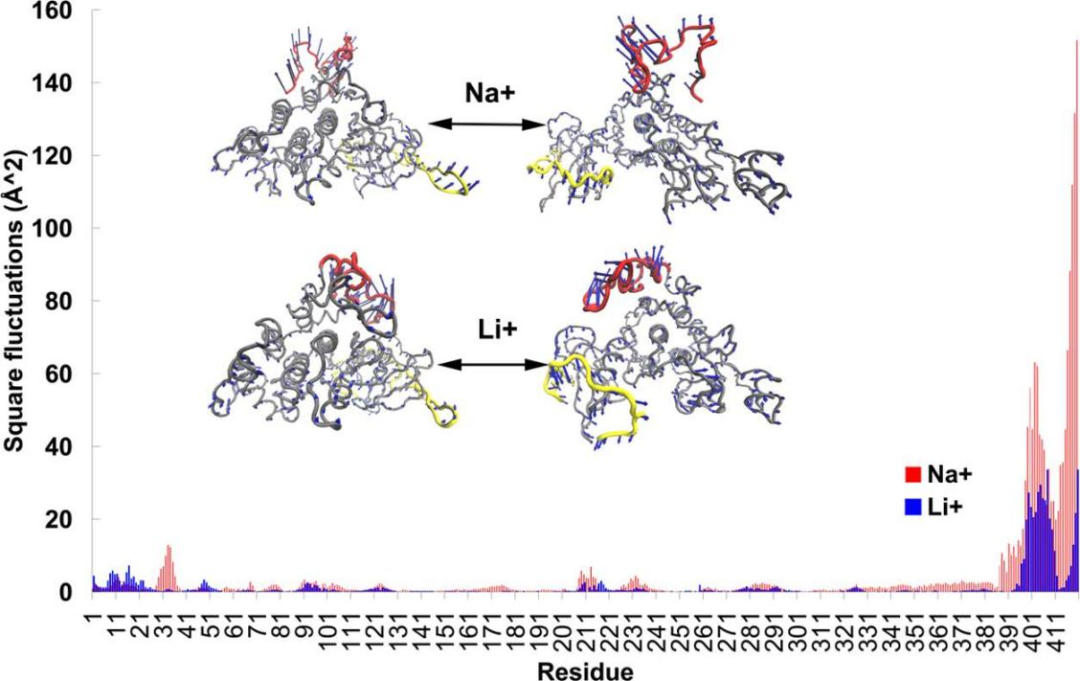
图 3. 三维(3D)密度代表稳定状态特性。图片来源于JCIM
对于活性部位的代表性残基,研究者测量了常规MD模拟、AT-MD模拟和EDA生成轨迹中三对残基之间的距离变化(图4)。结果表明在大多数情况下,锂通过推动GSK-3β的两个域相互靠近来关闭活性位点。
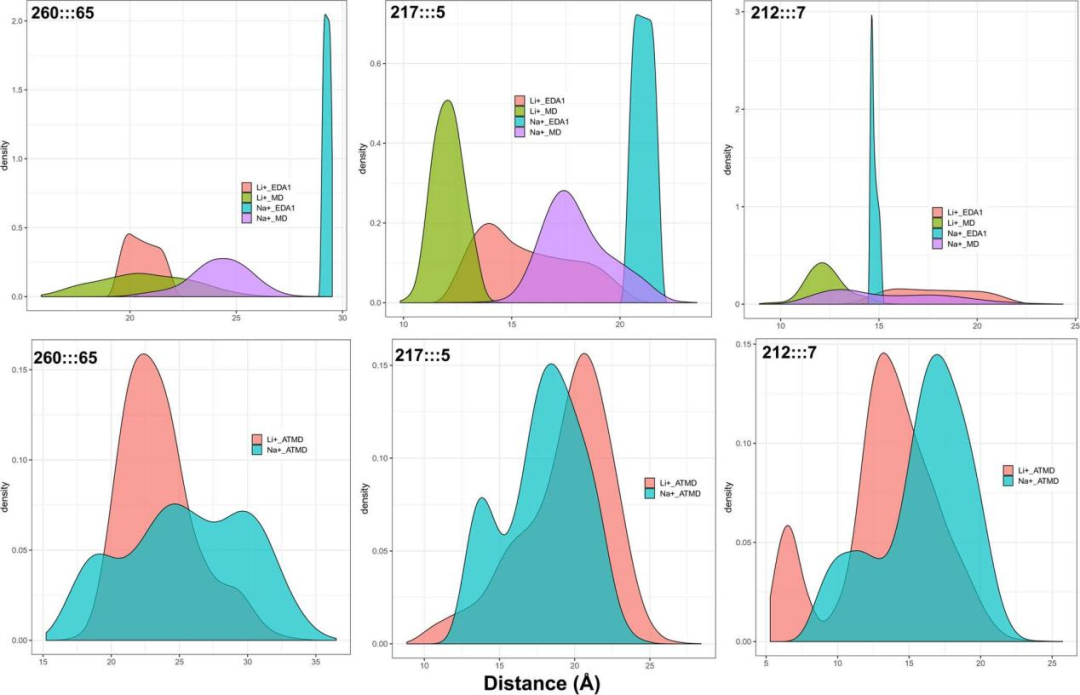
图 4. GSK-3β活性位点开放的动力学结果。图片来源于JCIM
在AT-MD模拟中,GSK-3β三维结构周围的离子分布是不同的。通过计算GSK-3β结构周围锂离子、钠离子和氯离子的平均密度,研究者发现当对应的阳离子是钠离子时,带负电荷的离子在GSK-3β活性位点沟槽中的密度更高(图5)。在锂存在的情况下,它可能扎根于GSK-3β的封闭活性位点。
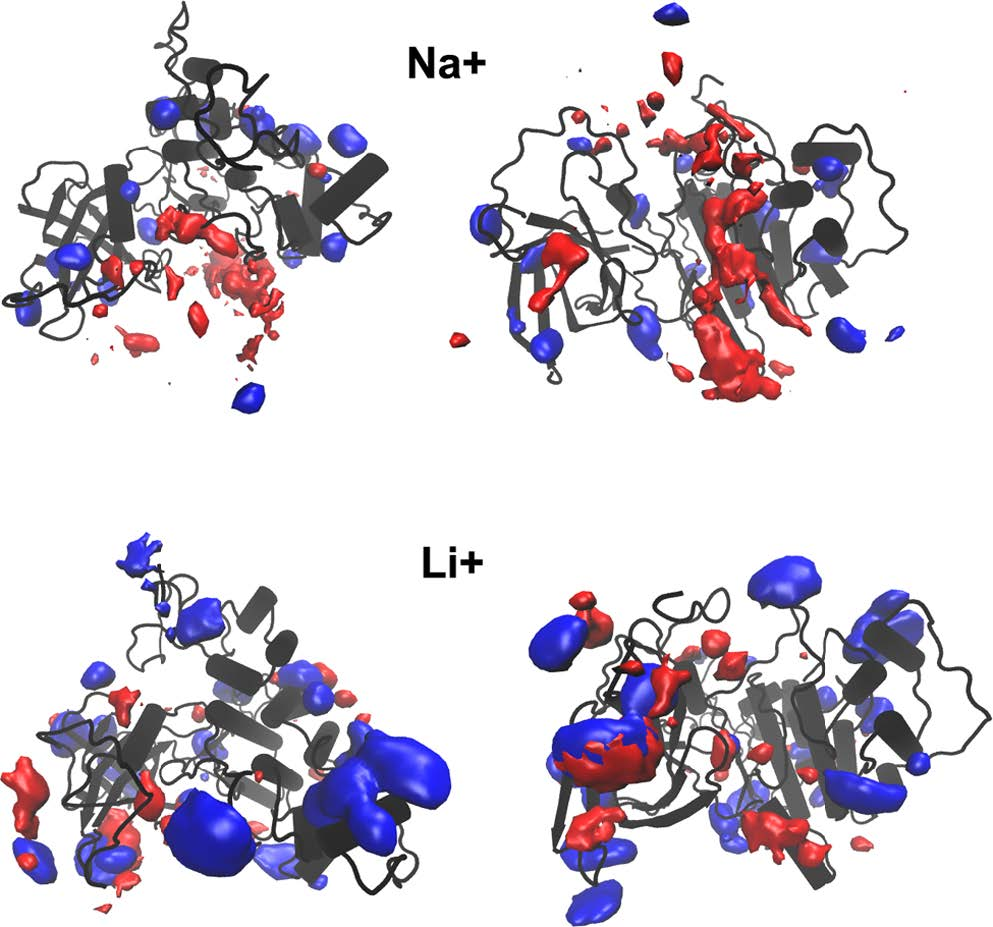
图 5. AT-MD系统中GSK-3β结构周围离子的位置密度计算,并将其呈现为等面图。图片来源于JCIM
相比之下,在GSK-3β的开口周围观察到较高的锂密度。通过在AT-MD模拟中平均GSK-3β蛋白在LiCl或NaCl存在时的静电势,研究者观察到,锂存在的情况下,一半的GSK-3β活性位点周围有扩展的正静电势表面(EPS)(图6)。而在钠离子存在下,GSK-3β的C端具有明显的负静电势。所观察到的阳性EPS,可能会引导磷酸化底物进入活性位点。
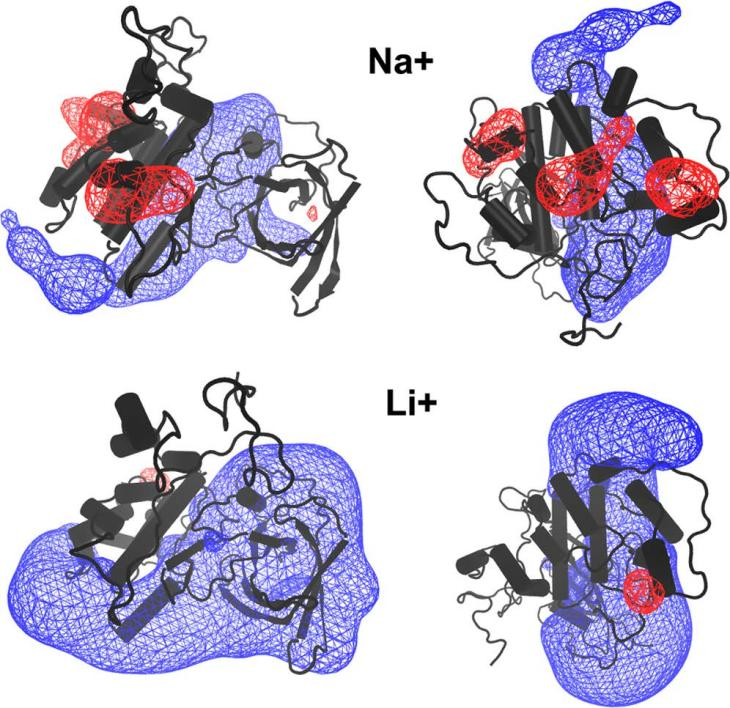
图 6. GSK-3β的静电势。图片来源于JCIM
锂离子对GSK-3β及其伙伴亲和性的影响
通过MD模拟,研究者比较了两种离子对PBPs的亲和力,并计算了常规MD、自适应回火MD (AT-MD)和基本动力学分析(EDA)模拟的z-score值。在片段1、片段2、片段3、片段4、片段4a、片段4b、片段4c和片段5中进行了AT-MD仿真(图1)。表1总结了在有Li+或Na+的情况下,沿轨迹有更高、相等和更低亲和力的GSK-3β结合贴片的百分比。
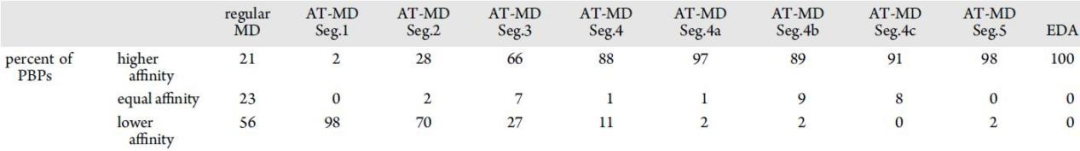
表 1. 在有锂或钠的情况下,高、相等或低亲和力PBPs的百分比。MD从常规MD开始,在自适应回火(AT-MD)的连续片段中进行,并由基本动力学分析(EDA)完成。表格来源于JCIM
集合1(包含全部聚合的AT-MD轨迹)的双样本t检验结果表明,当周围环境中钠离子被锂离子取代时,在209个GSK-3β结合贴片中,163个贴片(78%)的亲和力增加,36个贴片(17.2%)的亲和力降低,10个贴片(4.8%)对其伴侣的亲和力没有改变。从GSK-3β/轴抑制蛋白1 (Axin-1)、GSK-3β/原癌基因FRAT1 (FRAT-1)和GSK-3β同型二聚体复合物的PDB结构中分离得到的3个结合贴片,在锂存在时的亲和力高于钠存在时的亲和力。
PANTHER数据库用于定义GSK-3β环境中锂离子存在时对通路的影响。在Interactome INSIDER预测的GSK-3β的209个伴侣分子中,189个被PANTHER(通过进化关系分析蛋白质)数据库识别。这些蛋白质参与了75种不同的途径。在预测的GSK-3β伴侣分子中,19个蛋白参与Wnt信号通路,17个蛋白参与血管生成(图7)。在Wnt和血管生成途径中,74%和65%的这些蛋白在锂存在时,比在钠存在时更接近GSK-3β。促性腺激素释放激素受体通路(GRHR)和胆囊收缩素受体信号通路图(CCKR),各包含15个GSK-3β伴侣,其中67%和73%在LiCl存在时,也显示出比NaCl存在时更高的亲和力(图7)。
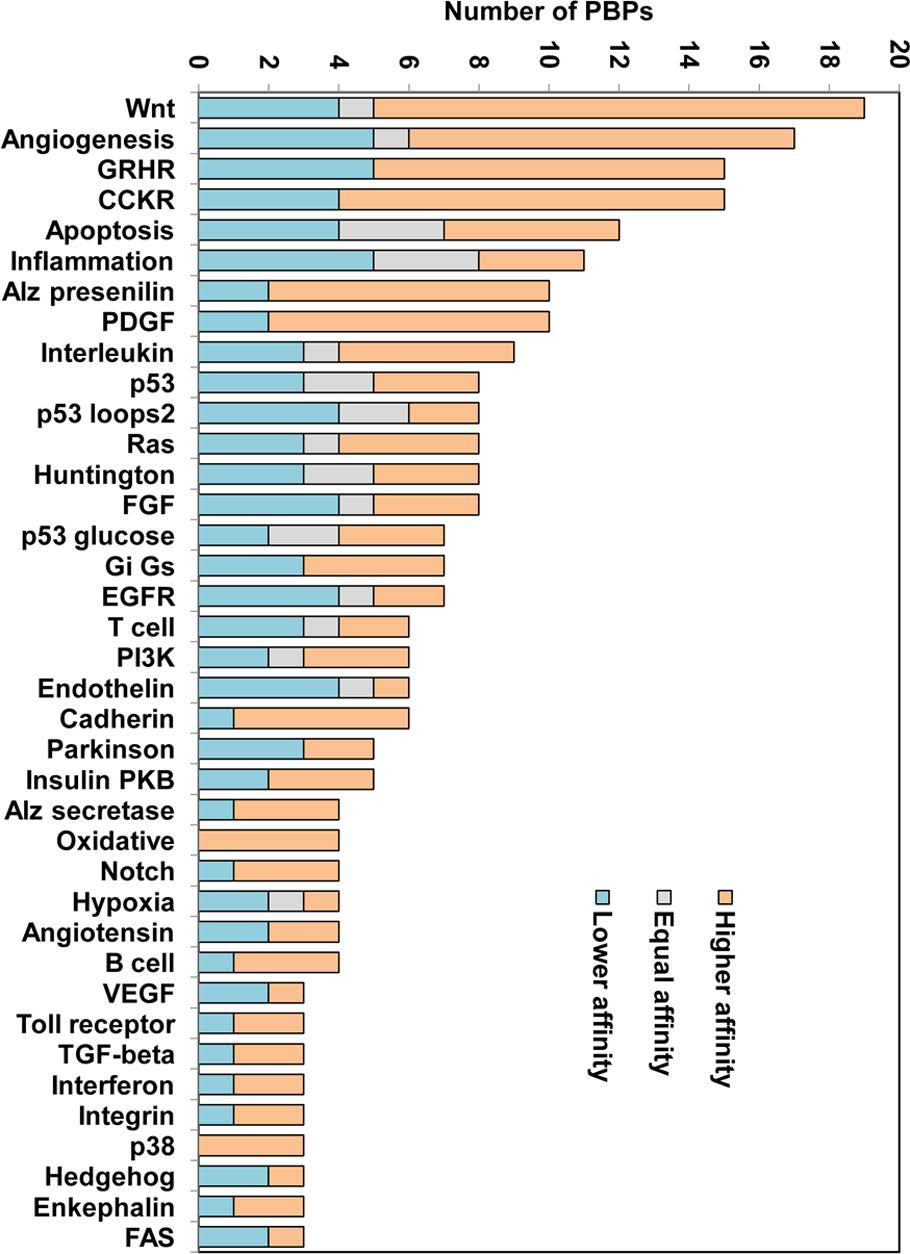
图 7. GSK-3β蛋白结合贴片(PBPs)的数量与参与多种途径的伴侣相互作用。图片来源于JCIM
结论总结
本文研究了LiCl作为药物,对GSK-3β蛋白在PIN中作为枢纽的各种蛋白结合贴片亲和性的影响。结果表明,一个小的离子,可以同时影响大量的PBPs,可增加或减少该蛋白与不同伴侣的结合亲和力。锂能改变GSK-3β的结构性质。它诱导酶活性部位的关闭。目前的研究提出了锂离子如何影响GSK-3β对其蛋白底物的作用以及锂离子如何影响GSK-3β PIN的新机制。本文首次提供了一种方法,即仅利用靶蛋白的结构来研究药物对其靶蛋白相互作用网络的影响,而不需要蛋白质复合物中其相互作用伴侣的结构。
参考文献
Rouhani, Maryam, and Hamid Hadi-Alijanvand. “Effect of Lithium Drug on Binding Affinities of Glycogen Synthase Kinase-3 β to Its Network Partners: A New Computational Approach.” J. Chem. Inf. Model. 2021, 61, 10, 5280–5292. https://doi.org/10.1021/acs.jcim.1c00952