

高通量实验与定向进化启发的深度学习方法碰撞,展示了机器学习使核靶向非生物微蛋白的从头设计将反义寡聚体输送到细胞核的过程。模型还可破译和可视化序列-活性预测。
背景介绍
已有文献使用二元分类器来优化活性细胞穿透肽 (CPP)活性,美国麻省理工学院的Bradley L. Pentelute团队则采用了新的方法,他们通过使用大型标准化数据集与深度学习来设计新的功能性微蛋白并预测活性。这种基于深度学习的设计策略,包括了序列的生成器、预测序列活性的预测器和提高序列活性的优化器三个部分。作者构建了一个包含 600 个独特反义-微蛋白偶联物的文库(图 1a),根据绿色荧光蛋白 (EGFP) 荧光读数来测试序列活性(图 1b、c)。残基被编码为指纹,用于训练预测神经网络(图 1d)。生成器神经网络可以生成新序列并用作优化的种子,在预测器-优化器中优化这些新序列,从而输出数百个高预测活性的新序列(图 1e)。
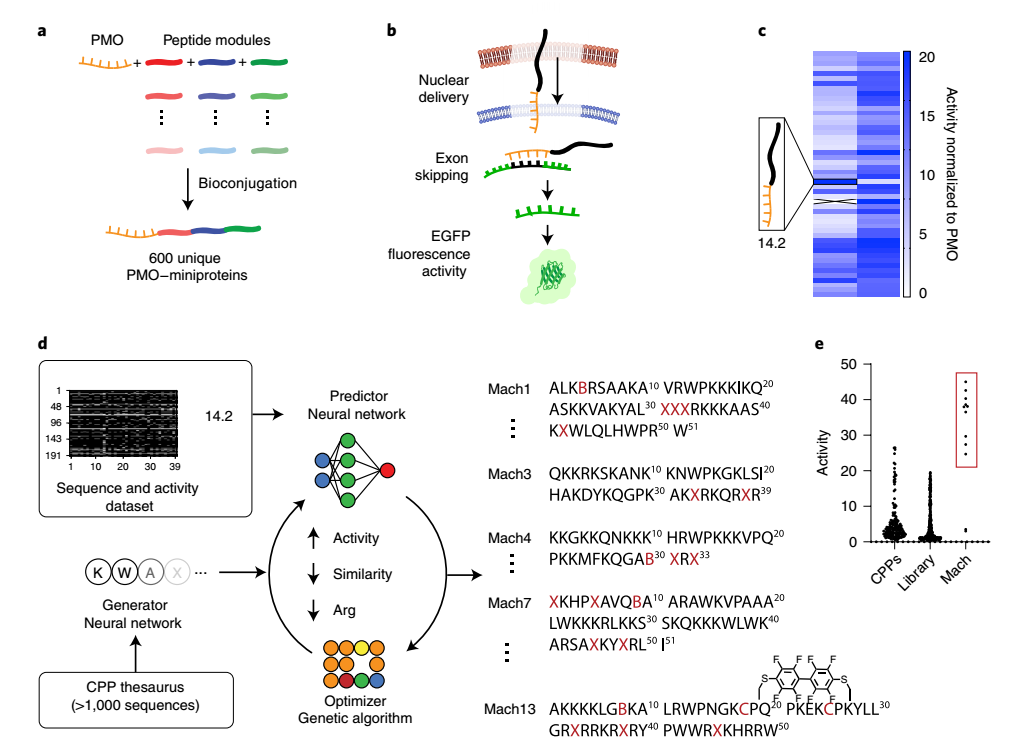
图1. 基于定向进化的机器学习模型预测用于大分子传递的高活性非生物微蛋白。图片来源:Nat.Chem.
主要内容
数据集的构建
合成策略采用四个模块:一个用于 PMO,三个用于包含不同结构和功能的不同肽序列库。作者在具有化学选择性和不可逆性的生物偶联反应中合成构建体,产生足够纯度的产物,用于体外直接测试。
建立深度学习模型
作者关注每个残基的分子结构,使用了基于沿序列堆叠每个残基的传统化学信息学指纹的拓扑表示。肽序列可以表示为残基指纹的矩阵,用零填充直到每个序列矩阵的长度相同。单个残基指纹是基于整个单体分子图的位向量(图 2a),向量中的每一位对应于一个子结构,这种方式简化了残基的表征,并度量了残基之间的相似性。最后将指纹编译成行矩阵以编码肽序列的酰胺骨架。
随机选择20% 的数据集作为验证集来检验预测器对于卷积神经网络的效果。验证集上的均方根误差是训练数据标准偏差的 0.4。只要预测值落在训练值的范围内(归一化活性为 0.32-19.5),预测相对误差为 11%(图 2c)。在回归和分类任务中,使用指纹和单热编码对其他模型架构进行了测试。结果表明,大多数模型都受到训练数据范围的限制,只有 CNN-FP模型能够外推并生成预测的活性,然而外推预测的偏差显著增加。由于异常值的存在,基于拓扑表示的模型在验证数据集上的效果并未优于单编码模型的预测效果,并且在 Mach 数据集上的表现更差。使用 one-hot 编码的 CNN 模型尽管其总体平均误差最低,但几乎没有外推能力。当研究异常值对模型性能的影响时,作者发现模型集成的 CNN one-hot 模型对验证数据集更优,而集成的 CNN-FP 模型则对 Mach 数据集更优。
作者开发了一个基于循环神经网络的生成器,它根据 CPP 生成“类似 CPP”的启动序列。使用可用序列的数据集作为输入训练模型,最后使用模型生成种子序列。结果发现生成器方法生成的序列能更好地满足预设的三个标准(高预测活性、低相似性和低 Arg 含量)。
优化器基于定向进化完成循环。生成器生成的序列被随机突变并使用目标函数评估序列的活性,从而进一步改进序列。在保持水溶性的同时最小化长度、Arg 含量以及与库的相似性,估计序列的净电荷。在对每个序列进行 1000 次迭代后,该模型提供了数百个具有广泛预测活性值的独特序列。除了高活性序列,作者还预测了非活性序列作为阴性对照。通过在相反方向引导优化器的进化,但保持其他约束相同,模型能够生成在氨基酸组成上与活性预测相似的非活性序列 (Mach11)。合成后,Mach11结合物显示出较低的实验活性,这证明了模型在预测独特序列活性方面的稳健性(图2c)。
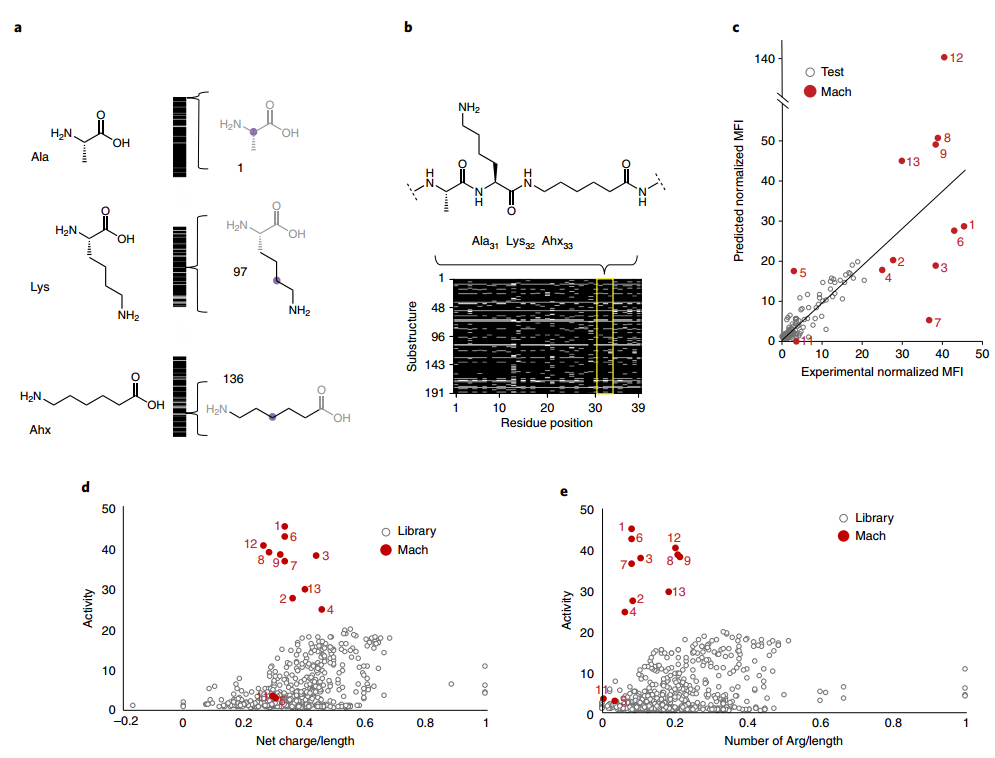
图2. 基于机器学习的生成器-预测器-优化器循环预测核靶向非生物微蛋白。图片来源:Nat.Chem.
预测模型的可视化和解释性
作者通过可视化关键的残基子结构来解释预测器CNN,开发了一种工具来解释序列与活性的相关性。该过程为序列中的每个化学子结构生成正和负激活值。具有较高激活的位表示对最终活性预测影响最大的特征。例如,对于预测的 Mach3 序列,两个 C 端氨基己酸 (Ahx) 残基的活性最高(图 3a),其次是Arg。使用这种可视化方法可以更好地了解训练模型如何设计序列。接着作者选择了五个不同长度的随机序列,将它们放入预测器-优化器循环中,以最大化其他设计约束条件下的活性,并可视化最佳预测的激活情况,可以看到 C 端残基更高的活化(图 3c),这很可能是由于 PMO 连接到了 N 端。另外研究人员还观察到带电残基和疏水残基的一般组成在不同的序列长度上保持不变(图 3d),无论序列长度如何,特定的残基指纹都被激活,例如 Lys、Ser 和 Asp 的侧链(图 3e,f)。
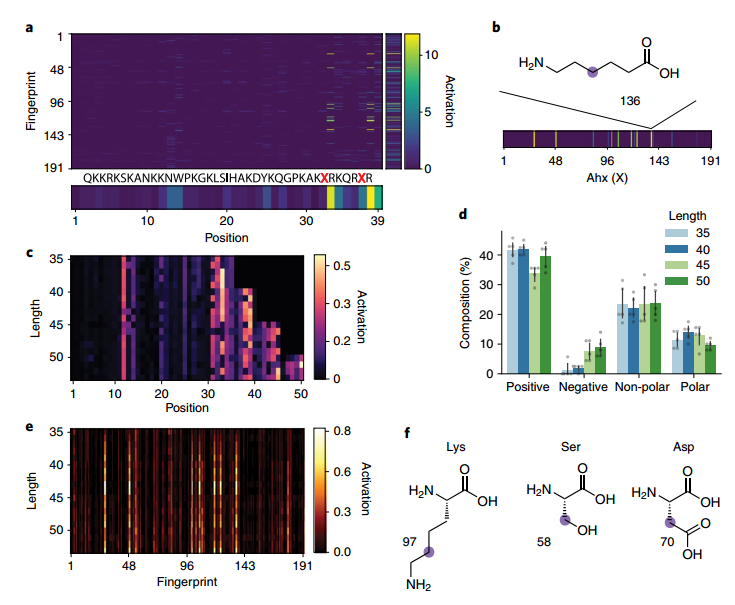
图3. 预测器 CNN 的解释揭示了激活的子结构。图片来源:Nat.Chem.
Mach微蛋白靶向PMO 转运
作者从模型预测的数百个微蛋白中合成并表征了 12 个候选物。选择 Mach1、2 和 6 是因为它们在 50 聚体序列中具有高预测活性。Mach3 被选为中等长度的肽(39 个残基),Mach4 被选为只有两个 Arg 残基的较短序列(33 个残基),选择 Mach5 是因为预测它具有中等活性和最低净电荷 (10.5)。Mach7 最初被设计为阴性对照——其中 Mach1 的序列被重新排列,直到模型预测最低活性。Mach8 和 9 是从更长的小蛋白(大约 80 个残基)列表中选择的,而 Mach12 和 13 是从包含 Cys 连接的大环的序列中选择的。最后,Mach11 是从活性在负方向上优化的序列列表中选出的,以表明该算法可以预测具有相似长度、电荷和氨基酸组成的肽,但没有 PMO 传递活性。
除了 Mach5 之外,几乎所有预计活性超过 20 倍的序列确实都超过了性能最高的模块化文库结构。由于模型在训练数据范围外外推,PMO-Mach 构造的预测和实验活性显示出比测试数据集更大的百分比误差(图 2c)。
经验证的预测的理化性质与 PMO 活性几乎没有相关性。作者将 Mach 构建体的活性与训练库中与各种理化特性相关的活性进行了比较(图 2d、e)。尽管文库构建体清楚地显示活性随 Arg 含量相对于长度的增加以及净电荷相对于长度的增加,但 Mach 构建体的活性与这些相同特性之间没有明显的相关性。
几种 PMO-Mach 构建体比以前表征的 PMO-CPP 具有更大的效力且无毒。作者进行了剂量反应实验以表征 EGFP 测定和乳酸脱氢酶 (LDH) 释放试验中的毒性。PMO-Mach2、3、4 和 7 的 EC50(半最大有效剂量)值均接近 1μM,并且在测试浓度下是无毒的(图 4a-c )。
Mash微蛋白转运其他生物大分子
肽核酸(PNA)是一类结构高度灵活的合成反义寡核苷酸, 测试的四种 Mach 微蛋白中的每一种都能够显着增强 PNA 递送(图 4d)。除了反义寡核苷酸,马赫肽还可以传递带电蛋白质,例如白喉毒素 A (DTA)。作者发现 Mach-DTA 构建体能够比单独的蛋白质更有效地递送到细胞胞质溶胶中,并且递送需要共价键(图 4e)。与马赫微蛋白的结合也改善了 EGFP 的传递, HeLa 细胞的共聚焦显微照片在与 Mach-EGFP 孵育后在细胞质中显示出弥散的绿色荧光,并在细胞核中显示出强烈的荧光(图 4f)。
最后,作者证明了 PMO-Mach 构建体在动物中安全正确地合成了蛋白质。含有与细胞测定中使用的相同的 EGFP IVS2-654 基因的转基因小鼠被单次静脉注射不同剂量的 PMO-Mach3 或 PMO-Mach4,并在 7 天后进行评估。两种构建体在股四头肌、膈肌和心脏中都表现出 EGFP 表达的剂量依赖性增加(图 4g-i)。
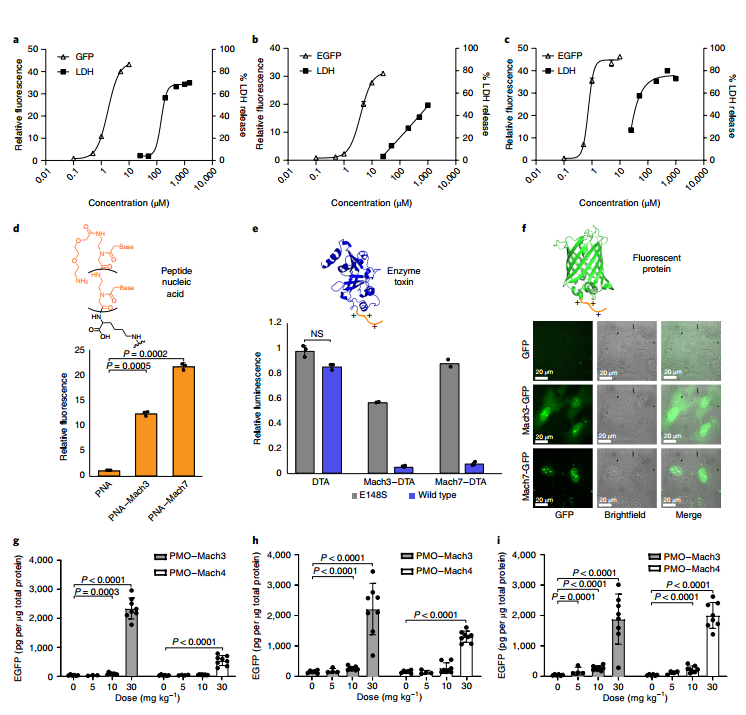
图4. Mach 微蛋白在体外和体内都具有高度活性,可将其他生物大分子传递到细胞质中。图片来源:Nat.Chem.
总结
本文构建的模型的核心优势在于:(1) 标准化的定量活性数据,(2) 模型在训练集之外进行外推的能力,以及 (3) 解释模型决策过程的视觉归因工具。该模型展示了如何将深度学习应用于功能性非生物微蛋白的从头设计。Mach 微蛋白是迄今为止开发的最有效的 PMO 递送构建体。机器学习框架可能会重新被用于发现具有其他所需活性的序列优化肽,只需要标准化的高质量输入数据集。这一策略将在未来帮助科学家快速设计对化学、生物和材料科学产生影响的去功能肽。
参考文献
Carly K. Schissel1, Somesh Mohapatra , Justin M. Wolfe, Colin M. Fadzen, Kamela Bellovoda,
Chia-Ling Wu, Jenna A. Wood, Annika B. Malmberg, Andrei Loas, Rafael Gómez-Bombarelli and Bradley L. Pentelute. Deep learning to design nuclear-targeting abiotic miniproteins. Nature Chemistry, 2021, 13, 992-1000.