

NIH开发了第一个可加速发现βGUS(β-葡萄糖醛酸酶)抑制剂的虚拟高通量药物筛选平台,可开发潜在的辅助治疗药物的靶点,进而减少与一线抗癌药物相关的SAEs(严重副作用)的频率。
背景介绍
对癌症而言,药物干预可能是治疗补救的第一线。由于先天和适应性免疫细胞的高复制率,癌症治疗过程中细胞凋亡出现的表型可能包括恶心和呕吐、食欲不振、脱发,以及发展为免疫损害状态。由于患者试图避免这些严重副作用,因此抗癌治疗的不依从性很高。这个问题说明需要关注新的治疗策略,即可以抑制这些脱靶效应,从而通过减少剂量限制毒性来提高抗癌药物的治疗指数的方案。
肠道内抗癌药物,通过尿苷5 -二磷酸葡萄糖醛酸转移酶(UGT)介导的肝脏代谢生成无毒药物-葡糖醛酸苷。反过来,共生肠道菌群中的β-葡糖醛酸酶(GUS)作用于药物葡糖醛酸苷,切割其糖部分进行营养补给,在脆弱的肠细胞存在下释放重新激活的药物分子。因此,在脆弱的肠细胞存在的情况下,微生物依赖的小分子抗癌药物的活化,可能是胃肠道(GI)严重不良事件(SAEs)的原因。
之前的研究中,研究者发现一种抑制βGUS的前药可能会提高一线抗癌药物的安全性和耐受性。这项研究的结果表明,提供能够阻断βGUS的治疗辅助制剂可能会减轻与小分子抗癌治疗相关的患者SAEs的负担。由于这种调节可能产生类似于或潜在地增强抗癌治疗的SAEs,因此,一种仅影响βGUS的精确抑制同时保持人类微生物组有益活性的策略,对该领域的药物开发至关重要。
虚拟高通量药物筛选(vHTS)是一种高效的靶向药物发现方法,它可以模拟药物与特定靶点的结合模式,通过预测结合能来推断药物疗效。当靶标和结合位点明确时,可以降低大量的材料和人工成本。
主要内容
美国国家卫生研究院的Anup P. Challa & Min Shen等人报道了通过基于结构的虚拟高通量筛选(vHTS)发现新的βGUS抑制剂的工作。研究者基于构建的βGUS同源模型,对美国国立卫生研究院(National Institutes of Health)国家转化科学推进中心化学库中的近40万种化合物进行了vHTS。从虚筛结果中通过多因素优先排序程序筛选出了291个化合物,获得了69个在体外βGUS生化检测中表现出阳性抑制活性的化合物。相关的研究成果以“Virtual Screening for the Discovery of Microbiome β-Glucuronidase Inhibitors to Alleviate Cancer Drug Toxicity”为题发布在国际著名期刊JCIM上。
βGUS同源模型的结构分析
研究者将vHTS的工作重点放在“H11G11-BG”上,这是一个具有代表性的βGUS同源物,涉及瑞戈非尼-葡醛酸的加工过程,它是酪氨酸激酶抑制剂瑞戈非尼的葡醛酸代谢物,用于治疗转移性结直肠癌、肝细胞癌和胃肠道间质肿瘤。图1为用H11G11-BG生成的MOE和I-TASSER同源模型,并显示了这些模型在结构上的异同。
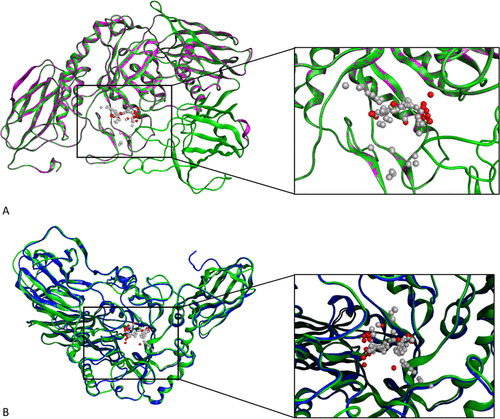
图1. 利用MOE和I-TASSER构建的βGUS的同源模型。图片来自JCIM
已知βGUS抑制剂的对接分析
研究者观察到MOE对接分数最低的化合物是磺化葡萄糖分子;但是考虑到该化合物与βGUS底物非常相似,并且在βGUS活性位点周围具有较小的表面积,研究者决定在选择模板结合模式时,探测表面积更大、结合能差略高的化合物。将该配体重新对接到研究者的全息靶结构上,并使用MOE的Protonate3D功能来获得所有具有可旋转键的靶点残基的最小空间阻碍构象,从而捕获了最终的结合姿势(图2)作为vHTS的模板。
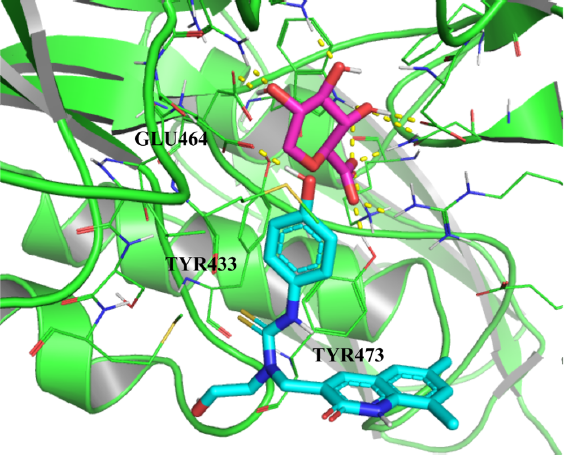
图2. ßGUS与1-((6,8-二甲基-2-氧-1,2-二氢喹啉-3-基)甲基)-1-(2-羟乙基)-3-(4-羟基苯基)硫脲结合的纯模板。图片来自JCIM
从对接分析中,研究者确定的关键残基包括:
1. Glu464(允许H键和亲核攻击,取决于靶标配体集合的环境pH值。)
2. Tyr433(考虑到苯环在该残基和模板抑制剂中的相对位置,如果Tyr R基团旋转使其苯环平行于候选苯环,则通过π -π堆积实现π -π稳定是可能的。)
3. Tyr437(允许通过与残基的自由羟基的H键稳定靶配体组装。)
4. Lys563(允许通过H键与带电的R基团稳定靶配体组装。)
5. Arg564(允许靶配体通过H键与带电荷的R基团稳定。)
6. Phe448(似乎在βGUS载脂蛋白构型中对抑制剂的结合提供了空间位阻,但与底物的结合促进了其逆行运动,因此这种构型的改变使配体能够访问位于靶点活性位点附近的抑制剂结合位点。)
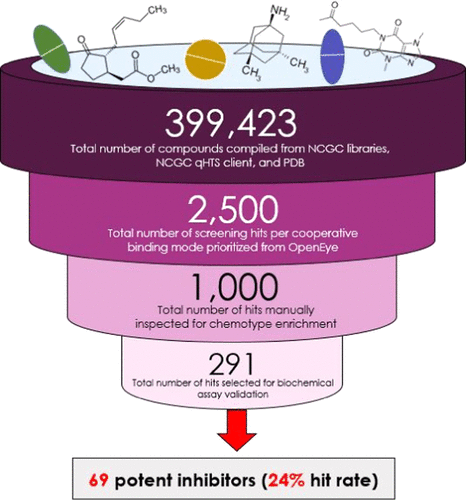
图3.发现ßGUS抑制剂的工作流程。图片来自JCIM
通过vHTS发现新型抑制剂及其结合模式
研究者在去除冗余位点和具有潜在不良功能的混杂化合物、细胞毒性化合物和PAINS motif后,选择了1000个经过筛选的化合物进行了多参数评估。对这1000个化合物进行了结构聚类,并对所有聚类和单分子的结合模型进行了目视检查。最后,根据预测的结合模型、排序分数、代表性结构簇和待测化合物的库存可用性,筛选出291个化合物。
作者为这291个化合物编译了一个结构文件库,将它们导入到MOE中重新对接研究者的同源模型,以确保它们保持在创建vHTS网格盒时指定的关键相互作用。在此过程中,研究者注意到优先命中位点与他们在βGUS活性位点中发现的关键稳定和亲核残基相互作用一致,一些Top化合物包含多个六元环。
在分析生化vHTS命中验证试验的结果中,研究者在291个vHTS命中中确定了69个被确认为有效(即CC≠4和CC < 0)的分子,命中率为24%。在这些强效化合物中,研究者观察到很少有单分子靶点。此外,发现了13个活性强的化合物,其活性被初步定义为未来探针开发的高质量活性物质(即IC50 < 10 μM,有效性< -50%)。对接流程如图3所示。
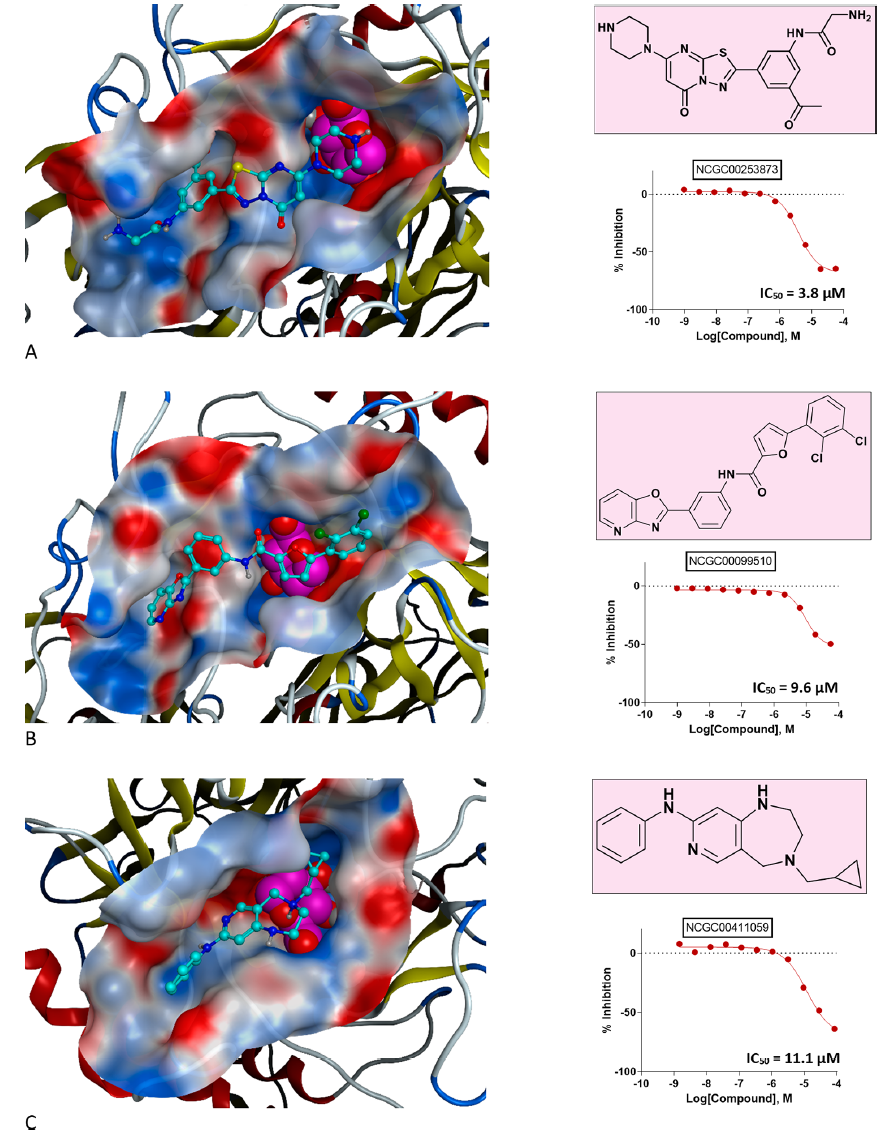
图4. vHTS得到的代表性化合物,包括RUC-2i(面板A)和5-(2,3-二氯苯基)-N-(3-(恶唑啉[4,5- B]吡啶-2-基)苯基)呋喃-2-甲酰胺(面板B)。图片来自JCIM
在图4中,研究者展示了top3命中化合物的结构、结合模式和剂量反应行为。通过对接实验,研究者注意到在选择抑制剂模板时,这些化合物之间的结合模式符合研究者的核心标准,这是对该方法有效性的概念的初步证明,也是对该结果有效性的进一步“完整性检查”。
NCGC00253873是研究结果中最有效的化学系列的代表性化合物,当研究者将它与I-TASSER同源模型重新对接时,重现了“约束”残基之间的关键相互作用。结果表明,生理相关βGUS菌株的IC50 = 3.8 μM,有效率= – 71%,CC = – 1.2,且具有清晰的逻辑量效曲线。
第二高的NCGC00099510对βGUS的IC50 = 9.6 μM,具有逻辑量效行为,CC =−1.2,有效性=−50%。第三高的NCGC00411059也表现出逻辑量效行为,IC50 = 11.1 μM,有效性= -72%,CC = -2.2。
在虚拟筛选加上生化βGUS试验这个工作流程中观察到的复合疗效是强大的。与仅通过体外平台测试NCATS化学库相比,大大缩小筛查的规模,这表明这些结果可能比标准的HTS工作流程更明确。此外,通过对化合物活性的新观察表明:vHTS具有从高通量药物发现平台挽救“假阴性”从而提高其敏感性方面的能力。
总结
本文展示了一个整体的、多层次的尝试,研究者试图通过vHTS的强大方法和下游生化分析体外验证来系统地发现βGUS抑制剂。通过对约40万个化合物的分析结果表明,这种方法是成功的,因为与标准的实验性HTS药物筛选计划相比,成功率提高了23倍。该工作流程未来可用于药物再利用,充分挖掘这类药物在治疗凝血障碍方面的潜在应用。
参考文献
Challa, Anup P., et al. “Virtual Screening for the Discovery of Microbiome β-Glucuronidase Inhibitors to Alleviate Cancer Drug Toxicity.” s: J. Chem. Inf. Model. 2022, 62, 1783−1793 DOI: 10.1021/acs.jcim.1c01414