基于从机理上理解复杂的生物相互作用系统的PyBindingCurve技术开发的CLAffinity,可利用药物筛选过程中常见的蛋白、配体和抑制剂浓度来模拟竞争实验。
背景介绍
敏感的、并行的和小型化的竞争实验,通常用于早期药物发现的初级筛选阶段,目的是从中等大小的小分子库中识别出被命中的化合物。筛选库中包含的小分子有可能通过竞争占据靶蛋白上相同的结合位点来抑制蛋白-配体相互作用。这些化合物,要么在靶蛋白之前添加到标记的配体溶液中,要么在一定孵育时间后添加到预先形成的复合物中以确保达到平衡。将仪器信号的变化转换为靶蛋白复合物的配体部分的测量,可以监测由与自由标记配体竞争同一结合位点的抑制剂引起的复合物解离。以这种方式建立竞争实验,使用标记的配体作为探针,通过该探针可以推断筛选库中潜在抑制剂的行为。在规划筛选试验时的假设是,通过一个容易与之竞争的弱结合配体可以获得最大的敏感性。
竞争系统的实际情况常使得这种假设不正确。靶蛋白和标记配体配合物的形成以1:1的化学计量,取决于蛋白质的浓度、相互作用的基本解离常数和标记配体的浓度。增加蛋白浓度,保持标记配体的浓度不变,形成双曲结合曲线,复合物浓度渐近达到最大值。如果决定在给定的亲和力下增加配体结合比例(络合配体与自由配体),为了在添加抑制剂时产生更大的读数变化,可以考虑在保持蛋白浓度不变的情况下增加配体浓度。除了冒着检测器饱和的风险或进入检测器的非线性响应范围外,这种方法还增加了自由未结合配体的数量,比结合配体的数量更多。另外,在保持标记配体常数的同时增加蛋白质浓度也有其自身的缺点;不与配体结合的自由蛋白质的丰度也增加了,而配体是信号的来源,也是复合物形成的功能。与游离蛋白结合的抑制剂,不会导致可检测的读数变化,导致检测灵敏度的降低。这些初步筛选试验假定结合系统完全平衡。
Huang等人反驳了以上假设,并建议使用亲和力最高的配体进行实验,以确定竞争性抑制剂的亲和性。但研究者注意到,这一建议不适用于基于竞争实验的初级筛选,因为即使是低亲和力相互作用也能实现明确的yes/no结合测定。
主要内容
英国爱丁堡大学的Steven Shave & Manfred Auer等人证明,在基于竞争的初级筛选中,使用非常高亲和力的配体会降低检测灵敏度,并最终丢弃潜在的有价值的活性化合物。研究者基于PyBindingCurve软件技术,开发了CLAffinity工具,它可利用药物筛选过程中常见的蛋白、配体和抑制剂浓度,来模拟竞争实验。CLAffinity揭示了基于检测参数的最佳标记配体亲和力范围,而不是优化检测灵敏度的一般规则。
开源代码
https://github.com/stevenshave/competition-label-affinity
也可用pip Python包管理器进行安装
配体亲和力对KD值范围内抑制剂检测的影响
制药公司通常根据标准浓度为10 μM和靶标配体结合度为0.7的标准来筛选化合物。研究者模拟了不同靶蛋白亲和力的抑制剂引入时配体结合的分数(仪器读数)。如图1所示,x轴表示标记的配体KD值,表示为pKD(−log10(KD)),并从低亲和力过渡到高亲和力(x轴)。随着标记配体亲和力的增加,达到0.7个配体结合所需的靶蛋白减少。当配体亲和力进一步增加,由于难以置换标记的配体,检测响应的动态范围被降低。初级高通量筛选,通常旨在识别具有良好检测敏感性的低微摩尔至中纳米摩尔KD值抑制剂,该信号用于产生明确的 yes/no决定,通过定义距离阴性对照均值三个标准差的检测阈值。这突出了用于进一步确证二次测定和KD值测定的化合物。因此,初级筛选的目标应该是使用能够产生最大响应的配体,通过大幅降低一系列抑制剂KD值的配体结合比例来实现。
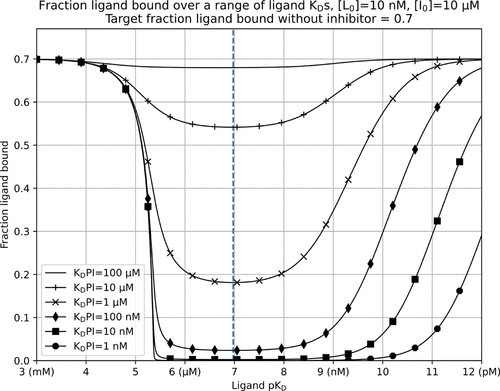
图1. 在竞争实验中,配体亲和力影响了KD值范围内抑制剂的检测。图片来自JCIM
对于图1所描述的系统,在一个范围内抑制剂KD值的信号变化最大的配体pKD为6.975 (105 nM KD)。
为了说明在初次筛选中选择具有最佳KD值的配体的影响,研究者考虑两种情况,首先使用一种与靶蛋白的KD值为100 nM的中等至高亲和力的配体,另一种与靶蛋白的KD值为1 nM的非常高亲和力的配体。在两个实验中,将标记配体的浓度保持在10 nM,使用标准库化合物筛选浓度为10 μM。通过实验读数检测和推断添加的抑制剂的靶蛋白KD亲和力值为1 μM。实验得到了以下结果:(i)使用KD亲和力值为100 nM的标记配体,在没有抑制剂的情况下,需要240 nM的蛋白才能得到0.7个配体。添加抑制剂后,配合物中配体分数为0.181,读数降低了74.1%。(ii)使用KD亲和力值为1 nM的标记配体,在没有抑制剂的情况下,需要9.3 nM的蛋白才能得到0.7个配体。添加抑制剂后,配体结合度为0.348,读数降低了50.3%。
配体结合靶分数越低,说明不同靶亲和力的配体所引起的信号降低差异越大。为了证明这一点,研究者考虑了另外两个实验,与上述实验相同,但使用的是靶分数配体,在没有抑制剂为0.1而不是0.7的情况下结合。在抑制剂存在的情况下,100 nM KD值的中-高亲和力配体和1 nM KD值的高亲和力配体分别能产生0.011和0.051个分数的配体结合反应。这相当于每次试验读出的动态范围的89.2%和48.5%。当错误地应用Huang的建议(在基于竞争的初步筛选中使用可能的最高亲和力标记配体)时,进一步增加了将抑制剂误判为非结合的可能性。
竞争实验的另一种可视化
图2是图1中数据的另一种表示,在x轴上交换配体pKD以获得抑制剂pKD。这需要对一系列抑制剂亲和值的信号还原一目了然,对于实际的分析读数可视化和理解预期反应更直观。在图2中,可以看到,对于弱结合100 μM KD值的配体(上实线,没有标记),在所有抑制剂KD值上,几乎不会破坏0.7个分数配体结合的小信号缩减。显然,这在使用标准浓度的竞争分析中是没有用的。当使用KD值为10 μM的配体(带+的线)时,开始看到对靶标的响应增加。研究发现,在抑制KD值范围内,信号显著降低。从100 nM KD的值来看,增加配体亲和力只会使响应曲线右移,降低低亲和力抑制剂的读数变化。100 nM配体KD值极限表示了图1中响应谷的右侧出现的特征上升。
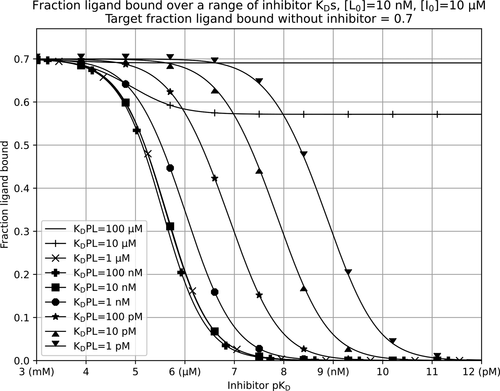
图2. 竞争实验的另一种可视化,显示了在配体KD值范围内,抑制剂pKD与配体结合的分数。图片来自JCIM
通过模拟,研究者已经证明了次优配体亲和力在基于竞争的初级筛选中可能产生的影响,即检测灵敏度降低和命中次数减少。在这里,不同亲和力的抑制剂之间的最大分离是必需的。图1所示的几乎是二元响应,使用100 nM KD值的配体(pKD为7)时,很难区分1、10和100 nM抑制剂亲和力,因为在所有情况下,分数配体结合几乎为零。
此外,图1可以用来直观地理解Huang关于KD测定的二次测定的建议。低亲和力配体抑制剂KDs的分离效果较差。配体的亲和性越高,不同KDs的抑制剂的可鉴别性就越高,在图的右上倾斜一侧,抑制剂之间存在较大的分离,表明引入抑制剂后,配体结合部分的变化较小。
总结
本文概述的模拟技术的应用有助于创建更高效的初级筛选及检测难药靶标的低亲和力抑制剂。为提高敏感性而采取的任何措施,都有助于扩大靶标抑制剂的化学多样性,从而可能进一步转化为新的线索。CLAffinity工具的应用,将防止Huang的规则不恰当应用,Huang规则的目的是用于二次确证检测,而不是初筛。研究者证明,选择高亲和力的配体,可以导致在不必要的高检测限下排斥有信号的化合物。这种对竞争系统的透彻理解,允许对分析进行调整或适配,使其进入高灵敏度的初级筛选阶段。
参考文献
Steven Shave, Nhan T. Pham, and Manfred Auer. CLAffinity: A Software Tool for Identification of Optimum Ligand Affinity for Competition-Based Primary Screens J. Chem. Inf. Model. Article ASAP DOI: 10.1021/acs.jcim.2c00285