

通过高通量虚拟筛选发现了MCULE-5948770040,一种作用于SARS-CoV-2主要蛋白酶的新型非共价小分子抑制剂。进一步的湿实验、X射线晶体学以及分子动力学模拟和机器学习阐明了配体的结合机制。
背景介绍
为了寻找新型冠状病毒大流行(COVID-19)的治疗方案,科学家们利用现有开发的针对SARS和中东呼吸综合征的治疗方案,发现了超过15种在病毒的复制中发挥着重要作用的病毒蛋白。在这些蛋白中,Mpro是一个很有吸引力的药物靶点,因为它在病毒复制中起着关键作用,且在人类基因组中没有任何密切相关的同源物。但识别SARS-CoV-2 Mpro的非共价抑制剂一直是一个挑战,因为其主要结合位点的内在柔性。
在本文中,美国国家虚拟生物技术实验室(NVBL)的Austin Clyde等人描述了利用HTVS平台发现非共价抑制剂的过程,该平台使用了超级计算机资源、集成对接策略、高通量实验筛选、X射线晶体学和分子动力学模拟(MD)。作为众包方法的补充,以及潜在O(十亿)化合物的HTVS,作者用了一个由MCULE库中650万库存化合物组成的库。使用Mpro的晶体结构进行了集成对接,对两个流行的对接程序(Autodock-Vina和OpenEye FRED)的Top评分化合物进行了实验表征。最终发现了一个Ki为2.9μM的抑制Mpro的分子,并解析了其1.8Å的室温X射线晶体结构。另外使用MD模拟来表征其与Mpro活性位点的结合机制,同时改变了酶的整体构象动力学。这份工作流程为快速发现针对SARS-CoV-2的可行先导分子提供了一个可扩展的框架。
On-Demand小分子库HTVS筛选Mpro抑制剂
作者首先从Mcule的可订购的On-Demand化合物库中对SARS-CoV-2的主要蛋白酶Mpro进行对接筛选。鉴于Mpro四个保守的结合位点(S1’、S1、S2和S4) 的内在柔性,作者使用五种不同的晶体结构,采用集成对接方法,使用了OpenEye Scientific FRED工具包中的对接方案和评分功能。
在5个结构、两个复合库和两个方案上,总共计算了超过6300万个对接。图1a总结了整个工作流程。流程部署在Argonne和Oak Ridge Leadership计算设施(ALCF/OLCF)的HPC资源上,使用了Theta和Summit超级计算机及德克萨斯高级计算中心(TACC;Frontera)和圣地亚哥超级计算中心(SDSC;Comet)。生成的对接库(包括准备和对接脚本)和对接分数可以作为一个可下载的数据集使用。
根据对接分数、肉眼检查和化合物的可得性对得到的化合物进行排序,并对选定的化合物进行实验验证。结果发现,每个晶体结构之间的对接得分分布略有不同(图1b,c),因此作者检查了总体分布的前0.1%。受体各自对接之间的相关系数最高为0.85(7BQY和6LU7),最低相关系数为0.001(6W63和7JU7)。
事实上,6W63的对接结果相对于其他4个受体是一个离群值,其相关系数最高仅为0.003。考虑到受体之间对接结果的差异,作者通过取可用系列中的最小值来创建一个一致性分数。OpenEye FRED有大范围的分数。受体之间的微小空间差异会导致很大的数值差异。如图1c所示,与使用单个样本相比,一致性得分的相关性存在显著差异。
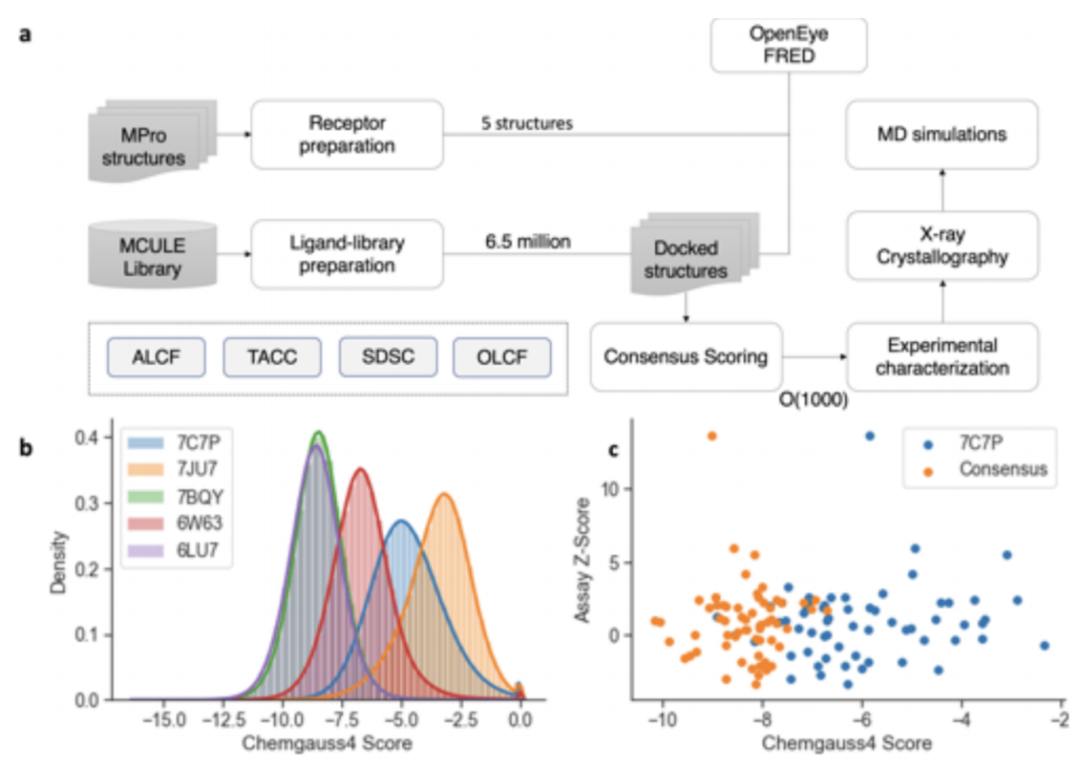
图1.(a)HTVS工作流程图;(b)Chemgauss4分数分布;(c)一致性评分。图片来源:JCIM
MCULE-5948770040作为
SARS-CoV-2Mpro抑制剂的实验验证
基于上述一致性评分程序,使用前20个不同的Mpro晶体结构,从Mcule数据库中选择了116个化合物进行实验筛选。在这116个化合物中,有5个无法排序,15个属于PAINS而被排除,最终有72个被交付。随后,作者对这些化合物进行初步的SARS-CoV-2 Mpro活性抑制筛选,筛选后与酶预孵育,并确定FRET肽裂解的初始速度。如图2,至少有7种化合物的抑制率为25%,其中MCULE-5948770040在20μM时的残留活性最低(12%)。
作者随后在40μM底物下测定了MCULE-5948770040体外Mpro抑制的浓度依赖性,IC50为4.2μM。在40μM未标记肽底物上也进行了基于正交定量高通量MS的终点分析,得到相似的IC50为2.6μM。另外,在20−500μM的底物和0−25μM的抑制剂下测量的初始速率与Ki为2.9μM时的竞争性抑制机制相一致。
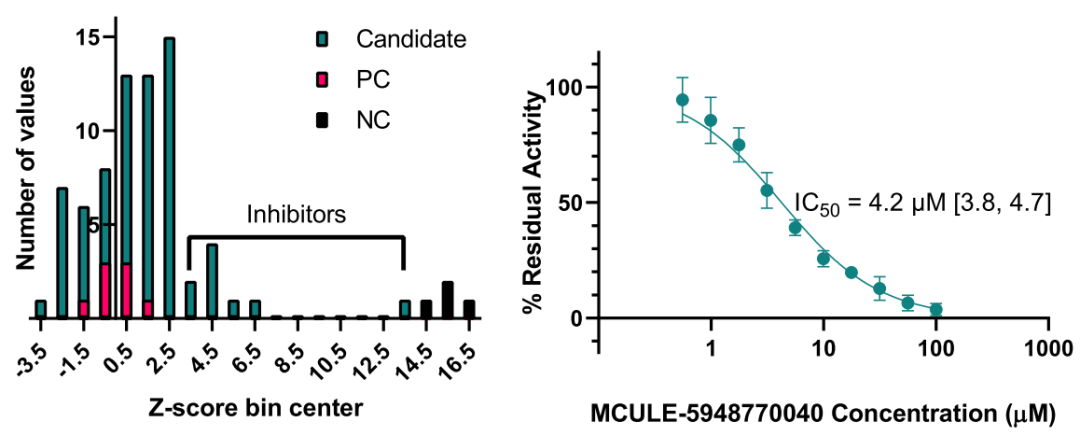
图2. 候选化合物的抑制活性。图片来源:JCIM
Mpro与MCULE-5948770040配合物的
X射线晶体结构
为了阐明抑制的分子基础,作者解析了在近生理(室温)温度下Mpro与MCULE-5948770040的1.8Å的X射线晶体结构。复合物结晶为生物学相关的同源二聚体,如图3。每个原聚体由三个结构域组成(I−III)。底物结合的裂缝在催化结构域I和II的界面上形成,而α-螺旋结构域III形成一个二聚界面。底物结合裂缝位于酶表面,分别在S1’−S5亚位点的P1’−P5位置容纳氨基酸残基或抑制基。由Cys145和His41组成的非典型催化二分体位于底物结合裂缝的深处,在C端P1’和N端P1位置之间进行肽键裂解。
根据晶体结构,MCULE-5948770040非共价结合到Mpro的活性位点,占据S1和S2的亚位点(图3a,b)。该配体的尿嘧啶P1基团位于由极性接触驱动的S1亚位点。His163咪唑侧链的Nϵ2与尿嘧啶取代基第4位的碳基形成紧密的2.6Å H键。His163之前被中子晶体学确定为在Nδ1上的单质子化,表明在配体结合时,His163侧链的质子化态可能被重排。Mpro的特征是由Gly143、Ser144和Cys145的主链酰胺NH基团在S1亚位点碱基上形成的氧阴离子空穴。将P1尿嘧啶连接到MCULE-5948770040的中心哌嗪连接体的碳基位于氧阴离子孔的外围,与Gly143形成直接的2.8Å氢键。
MCULE-5948770040复合物与Mpro的LF联合中子/X射线晶体结构的叠加显示,P2-二氯苄基进入疏水S2口袋,改变了Met49侧链的位置 (图3c)。MCULE-5948770040与Mpro活性位点的结合导致His41侧链翻转和χ2角旋转,为复杂结构中与P2-二氯苯的π−π堆积创造了有利的几何结构。His41上的构象变化使得His 41 Nδ1与Mpro结构中常见的保守的催化水分子(H2O cat)之间形成保守的氢键,但直接用连接His164的主链羰基的氢键取代它会导致额外的水分子从大体积溶剂中招募,与His咪唑环形成氢键。
计算预测的MCULE-5948770040与Mpro活性位点的结合姿势与实验确定的取向一致(图3d)。仅在哌嗪linker和p2-二氯苄基中存在微小的差异。哌嗪linker在晶体结构中被最优化为一个椅式构象,而对接的几何形状得分最高的采用了一个扭曲的船式构象。
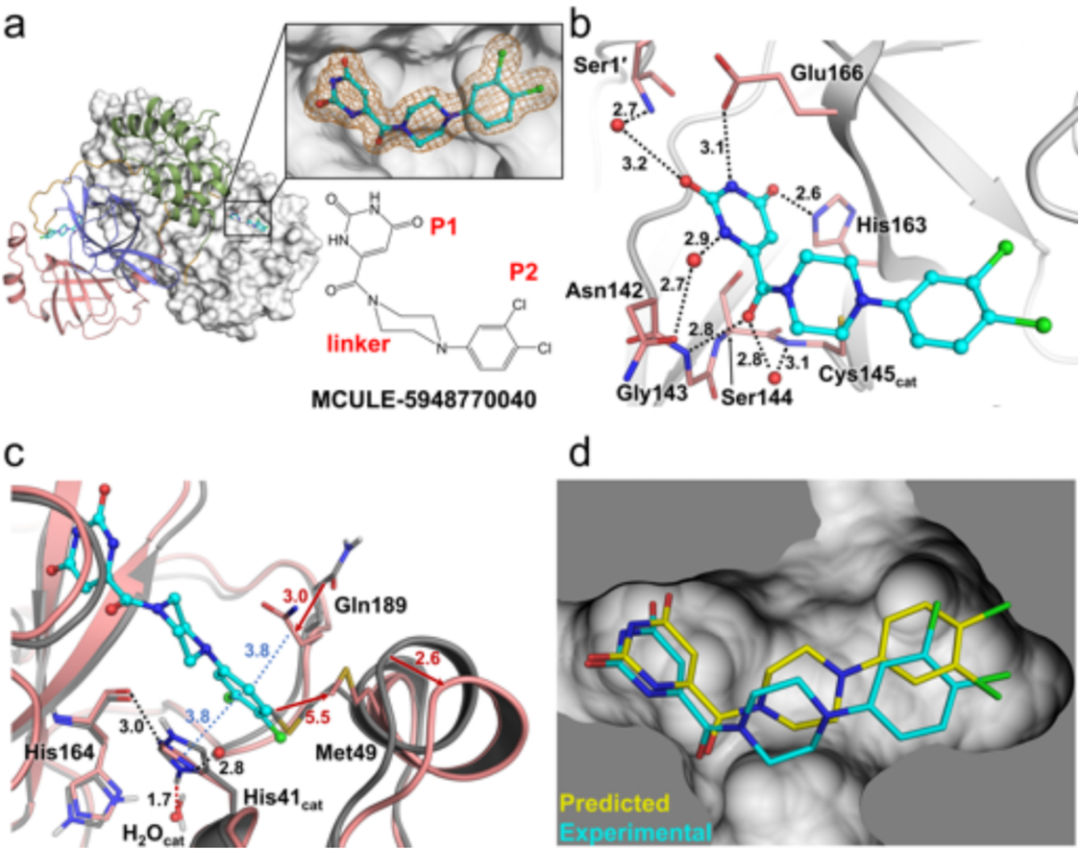
图3.Mpro与MCULE-5948770040复合物晶体结构,以及与LF和对接结构的比较。图片来源:JCIM
MD 揭示Mpro与MCULE-5948770040的互作机理
为了解分子如何与Mpro结合位点相互作用,作者进行了μs时间尺度的原子MD模拟。对于LB态模拟,模拟了两种Mpro原聚体中存在的配体。作者观察到配体仍然与主要结合位点结合(对于二聚体中的A链和B链),该蛋白也没有发生任何较大的构象变化。
作者使用LF和LB Mpro模拟中的均方根波动(RMSFs)(图4a)来了解配体是如何影响构象动力学的。为了方便,作者单独考虑了每个原聚体(模拟是以配体结合活性二聚体形式进行的),并观察到Mpro的几个不同区域表现出改变的波动。在作者所有的副本中,二聚体A链中的RMSFs略高于B链。值得注意的是,并不是所有的区域在每个原聚体中都表现出稳定性(例如,S4区域,原聚体链A表现出与LF状态相似的波动)。有趣的是,远离结合位点的区域,包括每个原聚体的结构域III(图4中的R5),在LB模拟中表现出较低的波动。
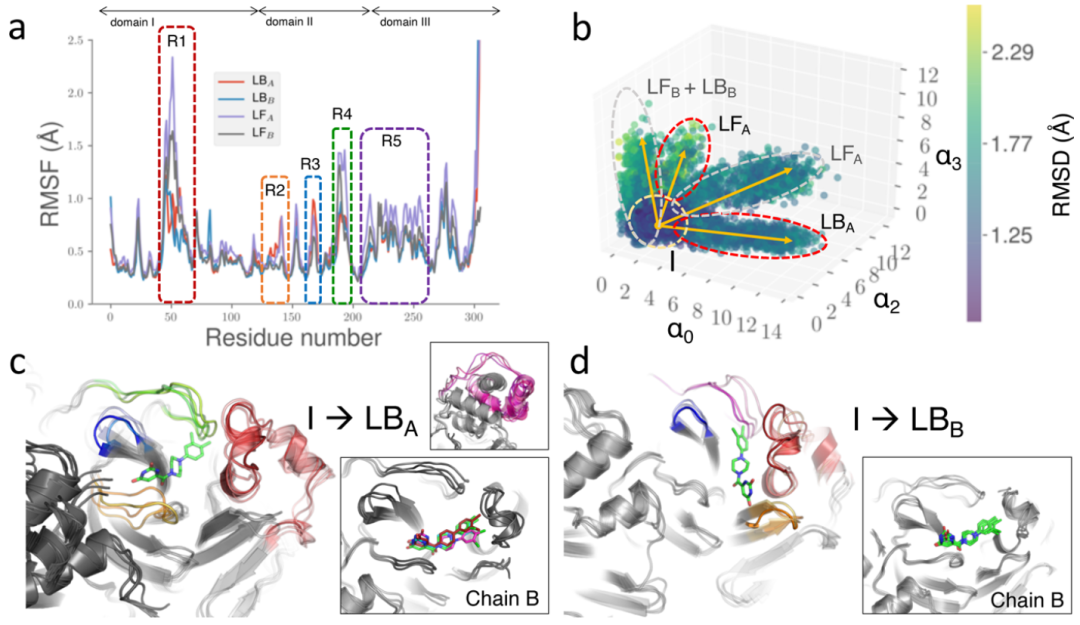
图4. MCULE-5948770040与Mpro结合时的构象变化表明了不同区域内的变化。图片来源:JCIM
为了阐明受配体的结合影响的运动,作者使用了支持非谐构象分析的自编码器(ANCA-AE),将LF和LB模拟所跨越的构象嵌入到一个低维集合中(图4b)。作者的模拟可以嵌入到一个10维的集合中,这很好地解释了蛋白质所经历的构象波动。在这些嵌入中,LF和LB模拟占据了不同的投射,LF模拟采样了不同的构象状态(通过RMSD对LB状态进行量化)。LB模拟中主要的构象变化局限于跨越S1—S4和R5的结合口袋,如图4c,d中原聚体A所示,没有观察到相对于原聚体B的显著运动。观察到的波动主要是由于原聚体A内的配体从其主要相互作用位点(P1和P2)重新定位到互补位点。P1-尿嘧啶与S4区域(残基)形成了新的相互作用,而P2-二氯苄基则保持结合在疏水口袋内。
虽然作者没有在晶体结构中观察到这种配体的位置,但这些配体运动在作者模拟的多个副本中普遍存在,并可以在尿嘧啶和蛋白质侧链之间形成稳定的相互作用。与这些变化相对应的是,蛋白质结构域III的运动(图4c,d)也被抑制,表明该区域可能在配体结合时稳定下来。作者还从LB模拟中检测了配体与蛋白质之间的氢键模式,发现原聚体B链中配体与蛋白质之间的氢键比A链中的氢键更稳定。
讨论
本文描述了发现新型Mpro非共价小分子抑制剂的过程。化合物采用可扩展的HTVS框架和MCULE靶向性化合物库获得。生化分析验证了该Mpro抑制剂的抑制常数(Ki)为2.9μM。X射线晶体学确证了化合物与Mpro的结合模式,以及稳定的氢键和疏水相互作用。分子动力学模拟和机器学习阐明了结合配体是如何改变Mpro的构象状态的,包括结合位点的近端和远端运动。为Mpro非共价抑制剂的进一步设计奠定了基础。
参考文献
Clyde A, Galanie S, Kneller DW, Ma H, Babuji Y, Blaiszik B, Brace A, Brettin T, Chard K, Chard R, Coates L, Foster I, Hauner D, Kertesz V, Kumar N, Lee H, Li Z, Merzky A, Schmidt JG, Tan L, Titov M, Trifan A, Turilli M, Van Dam H, Chennubhotla SC, Jha S, Kovalevsky A, Ramanathan A, Head MS, Stevens R. High-Throughput Virtual Screening and Validation of a SARS-CoV-2 Main Protease Noncovalent Inhibitor. J Chem Inf Model. 2022 Jan 10;62(1):116-128. doi: 10.1021/acs.jcim.1c00851. Epub 2021 Nov 18. PMID: 34793155; PMCID: PMC8610012.