

Polyply包含了:1)一种多尺度图匹配算法,用于快速生成参数,并适用于任意复杂的聚合物拓扑;2)通用的多尺度随机行走协议,能够高效地建立复杂系统,且不依赖于目标力场或模型分辨率。
背景介绍
生物大分子的动力学模拟已经成为研究人员补充实验分析的有力工具。自现代医学出现以来,人们一直在模拟单聚合物熔体或混合物,最近的趋势是研究更复杂的多组分体系,包括纯合成材料和生物合成的杂化大分子。通过MD对聚合物进行HT(高通量)筛选,有望成为实验性HT方法的补充,因为它通常比合成探索成本更低,并且具有实验性HT方法难以获得的性质。
尽管HT模拟方法很有前途,但它需要程序用快速、可靠和一致的方式构建拓扑和模拟盒子。此外,考虑到基于聚合物的系统行为背后的时空尺度层次结构,需要具有全原子和粗粒度(CG)分辨率的模型。虽然有大量的程序可以用于生物相关系统的MD模拟,如蛋白质、脂质膜和DNA等,但对合成和生物合成杂化大分子模拟的支持,很大程度上是有限的。据目前所知,还没有程序能够为任意复杂的聚合物体系生成输入参数和坐标,而不依赖于力场,并与HT方法兼容。
由于缺乏支持全原子和CG聚合物模拟的程序,限制了MD模拟在大型通用系统和(生物)大分子系统的HT研究中的应用。因此目前需要克服以下五大挑战:
a. 该程序需要能够生成坐标和参数,分辨率和力场独立。
b. 需要一个易于使用的管道,来生成基于系统组成的输入文件和坐标。
c. 该程序需要能够结合各种生物分子结构的聚合物系统的输入参数和坐标,如蛋白质、脂质双分子层和核苷酸。
d. 它需要能够建立复杂的系统,而不需要降低对模型的要求。
e. 坐标和参数文件的生成都需要足够快,才能进行HT研究。
主要内容
荷兰格罗宁根大学的Siewert J. Marrink等研究者,报告了一个可解决上述五大挑战的开源的polyply软件套件。它有助于生成(生物)大分子和纳米材料MD模拟的输入参数和坐标。polyply使用基于图的算法,允许用户从简单的库文件和残基图中生成任意组合和分支聚合物的参数文件。残基图不仅包含了聚合物的残基序列,而且还记录了哪些残基连接在一起。使用多尺度随机游走,polyply也可以用于生成任意力场和任意目标分辨率的起始坐标。此外,还包括复合物的重排,如微相分离聚合物系统或包在脂质囊泡中的多组分聚合物溶液。为了最大化模型和代码的可访问性,polyply通过python包索引来分发。相关的研究成果以“Polyply; a python suite for facilitating simulations of macromolecules and nanomaterials”为题发布在国际著名期刊Nature Communications上。
参数文件的生成
polyply生成参数文件的一般输入是目标分子的残基图。此外,还需要与目标分子中残基相对应的块和链接(参见图1虚线框)。目前,polyply提供了包含一些力场和聚合物参数的库,这个数据库将随着时间的推移而扩展。polyply根据残基图、块和链接的定义,分三步生成参数文件(参见图1):
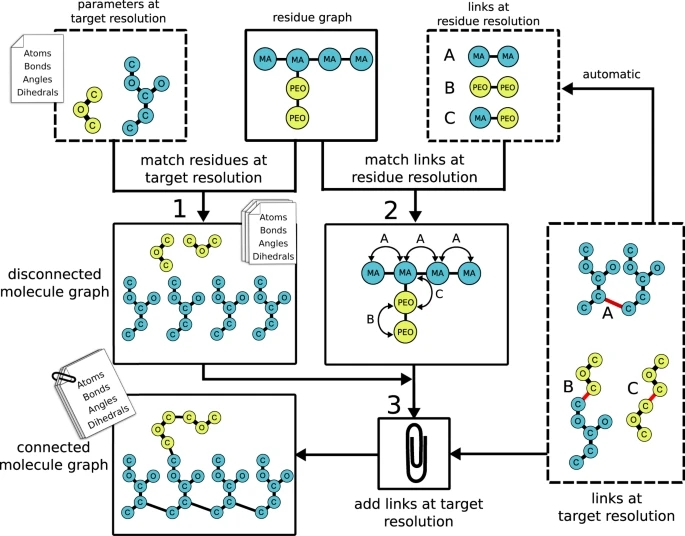
图 1. 参数文件生成工作流程示意图。图片来源于Nat. Commun.
步骤1:生成残基的非连通图:
读取输入文件后,对输入残基图的所有残基进行多重迭代。对于每个残基,将匹配块添加到空图中,从而生成目标分辨率处的残基断开图。这张图包含了目标分子的所有原子以及残基内的相互作用。因此,分配连接两个或更多残基的适当相互作用的问题仍然存在。
步骤2:找到残基层的所有链接:
为了生成跨越多个残基的所有相互作用,选择在两个或多个残基之间应用链接。为了用一般的方式来解决这个问题,研究者将其作为残基图层的子图同构问题:一个链接可以适合于残基图的约束,如匹配节点属性。
步骤3:将通用链接与特定残基匹配:
利用链接和残基之间的匹配,程序在目标分辨率处建立链接的原子和未连接图中的原子之间的对应关系。为此,链接中给出的原子名称和相对残基指数,只需与第1步生成的未连接图中残基的原子相匹配。它还可以扩展到考虑其他原子特性。当添加一个链接时,该链接的边也被添加到断开的残基图中。这样,在目标分辨率级,断开连接的图逐渐变成连接图。这就完成了图的转换,分子包括了所有的相互作用,而只需要写入一个文件。
系统构建
系统构建过程分为以下五个步骤:
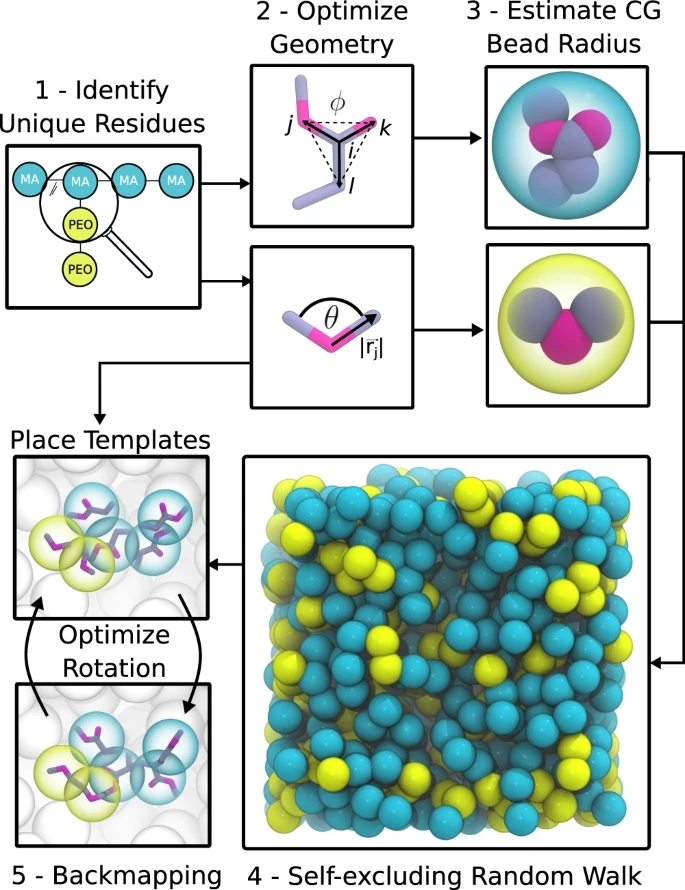
图2. 系统构建的多尺度算法原理。图片来源于Nat. Commun.
步骤1:将所有分子映射到每个残基的一个珠上:
第一步,分析拓扑文件,检测系统中所有分子类型。对于每个分子,所有独特的残基都被识别并转化为块。进而创建了一个一般的一粒一残基超级CG模型并以图的形式存储。残基图的连接性是从分子的键合图中提取出来的。
步骤2:生成残基坐标:
每个块是单个残基的图,使用图嵌入来生成该残基的坐标。由于分子几何的特殊要求,研究者采用了两步图嵌入的方法。先使用在NetworkX库中实现了的Kamada-Kawai嵌入生成初始坐标。随后,研究者利用有限内存Broyden Fletcher Goldfarb Shanno极小化器的scipy,对残基内的键合相互作用进行了几何优化。
步骤3:派生通用CG模型的参数:
在自排除随机游走中,采用了单残基珠近似CG模型。它基于Lennard-Jones (LJ)势作为交互函数。σ参数决定了整体的堆积密度,反映了残基的体积。研究者将这个概念移植到单个残基的分子几何上。此外,研究者还考虑了单原子具有的体积。
步骤4:约束随机游走:
为了在目标系统中为每个残基分子生成一个珠子的坐标,研究者执行了一个自排除的随机游走。如果在被放置的珠上的力超过了最大值,那么在随机游走中放置珠(步)的尝试将被拒绝。默认情况下,自排除随机游走是沿着分子图的宽度优先遍历进行的。这意味着彼此接近的节点(即残基)首先放置,然后算法沿着链继续前进。分子彼此分开放置,起始点从一个网格中随机选择。
这个网格可以是用户指定的,也可以被认为是跨盒子的矩形网格。当随机游走算法超过一定的步数时,默认情况下,它会在宽度优先的路径上后退10个残基,并试图替换这10个残基。为了满足距离限制,polyply实现了一个基于图的算法,该算法为每一步设置上下界。
步骤5:回映(Backmapping):
通过基于残基模板的backmapping程序,将低分辨率坐标转换为高分辨率目标坐标,类似于那些用于生物分子的程序。首先,将残基模板的几何中心移至重心位置;随后,优化围绕几何中心的模板旋转,这样,与其他残基有键的原子就被放置在那些残基附近。
为了实现手性,研究者目前使用了一个特殊的反常二面体,在能量最小化步骤和/或模板生成过程中强迫手性。
单链DNA和环状单链DNA
DNA是一种重要的生物大分子,在细胞中表达和调控遗传信息。虽然大多数遗传信息编码在双链DNA (dsDNA),单链DNA (ssDNA)参与重要的复制和修复过程。能否生成真实的ssDNA结构的多尺度随机游走协议,是另一个层面的挑战。一方面,DNA碱基是在全原子水平上约有30个原子的大残基。另一方面,ssDNA的持久性长度大约是大多数柔性聚合物的3-10倍。最后,ssDNA是高度带电的,可以协调周围的离子,从而增加持久性长度。
为了验证polyply能否生成这些大分子的构型,研究者在polyply中实现了Parmbsc1力场,并构建了不同长度(8、16、50、65、100个碱基)的poly-T ssDNA分布。对于每个长度,生成100个副本。对于每个链长,研究者设置了两个实验确定的持久性长度(3.2 nm, 1.4 nm),分别对应于低盐浓度(12.4 mM/L)和高盐浓度(1 M/L)。从而,获得了由polyply产生的1000个DNA结构。
图3a显示了与SAXS值相比,两组持久性长度的回转半径可作为链长的函数。随机游走方案对旋转半径没有偏好。生成的结构与实验测量值较好地吻合。在低盐浓度(v = 0.71± 0.01)下,用polyply测量的比例与实验中发现的比例一致(v = 0.72± 0.01),而在高盐浓度(v = 0.55± 0.01)下,比例稍低一些,但仍接近实验(v = 0.57± 0.02)。图3b显示了两条DNA链(100个碱基),对应于图3a所示的平均旋转半径。链的主要组成部分是对齐的,表明两者都是延伸的,但具有较高持久性长度的链明显是更长的。
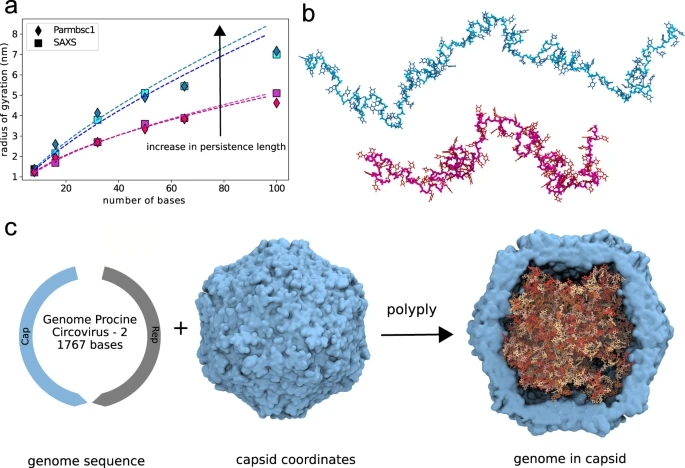
图3. 单链DNA测试案例。图片来源于Nat. Commun.
内部液-液相分离的脂质囊泡
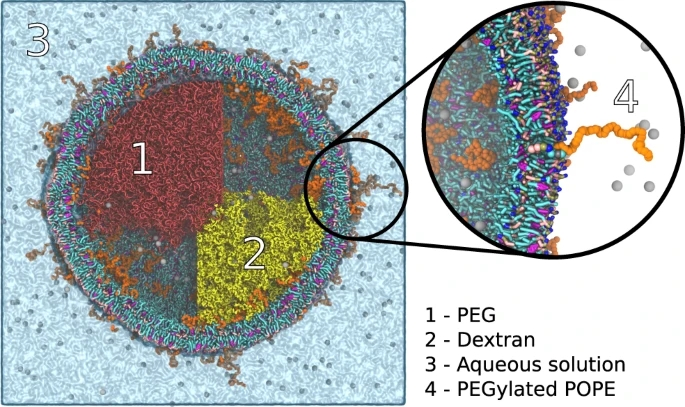
图4. 囊泡内的液-液相分离。图片来源于Nat. Commun.
研究者建立了一个系统组成的多组分脂质囊泡,由dioleoyl-phosphatidylcholine (DOPC) 、dipalmitoyl-PC (DPPC)和胆固醇组成,含有聚乙二醇和1-palmitoyl-2-oleoyl-phosphatidylethanolamine(POPE)脂质, 内部充满由PEO和葡聚糖组成的相分离水溶液。这个系统已经被实验证明可以诱导囊泡裂变,因此可用于洞察早期生命的形成。
为了生成这个系统的起始坐标,研究者首先使用TS2CG获得一个囊泡。将TS2CG生成的脂质坐标,作为起始结构进行聚合。此外,使用了一个几何约束来指定PEO和葡聚糖只能占据囊泡的一半,将其近似为一个球体,有一个2nm的重叠区域以允许一些间相混合。有了这个输入,系统由通用超级CG随机游走生成,然后是一个backmapping步骤。
生成整个系统大约需要30分钟。一旦获得初始坐标,就进行能量最小化,然后进行短暂的平衡和50 ns的生产运行。系统运行稳定,最终构型如图4所示。可以清楚地看到,系统至少在模拟的时间尺度上保持相分离,聚乙二醇化脂质均匀地分布在双分子层的内外。
结论总结
本文介绍的polyply旨在促进模拟涉及聚合物的任何目标分辨率和所需的力场。除了支持大型和复杂的聚合物之外,生成输入参数文件也很快,这使得它适合HT应用。总的来说,众多例子表明polyply能够设置大型和复杂的系统,将生物分子世界与材料科学连接起来。
参考文献
Grünewald, F., Alessandri, R., Kroon, P.C. et al. Polyply; a python suite for facilitating simulations of macromolecules and nanomaterials. Nat Commun 13, 68 (2022). https://doi.org/10.1038/s41467-021-27627-4