

Pharmacoprin是一种高分辨率的药效团指纹,可对分子的呈现、类型和药效团特征之间的关系进行编码。
背景介绍
目前,指纹已成为编码化合物结构最流行的方式之一,在化学信息学被广泛应用于相似度搜索、聚类和分类等任务。分子指纹的基本思想是对分子应用一个函数来生成一个位向量或计数向量。
另一种化学信息学尤其是虚拟筛选中常用的技术是药效团模型。药效团是与生物靶点相互作用,并触发生物效应的分子的各种结构特征的空间取向。药效团过滤器,本身可以作为筛选的一部分,甚至可以作为一个独立的工具。指纹也经常在机器学习方法中扮演输入的角色;因此,创建一个广泛描述化学结构药效团特性的指纹图谱,可极大扩展该方法的应用领域。
将指纹图谱与药效团模型相结合的做法并不新鲜。McGregor和Muskal等人使用10549比特的指纹来描述三点药效团,Wood等人的四点药效团将其扩展到超过30万比特,应用起来非常耗时且消耗CPU。ChemAxon在软件中引入了一种基于原子对的二维药效团指纹,这个指纹被定义为所有原子-原子药效团特征对及拓扑距离的集合,然而该指纹无法被转换成二进制向量。尽管前人已创建了高分辨率的表示,但该主题仍留有进一步探索的空间。
主要内容
在本研究中,来自波兰科学院药理学研究所的Rafał Kurczab等人提出了药效团指纹(以下称为Pharmacoprint)的概念和构建,以二进制形式描述药效团特征。Pharmacoprint在分类实验中使用ML算法(逻辑回归、支持向量机、线性支持向量机和神经网络)进行了评估,其性能优于其他流行的分子指纹(如ECFP4、Estate、MACCS、PubChem、Substructure、klekotra – roth、CDK、Extended和GraphOnly)和ChemAxon药效特征指纹图谱。Pharmacoprint由39973比特组成;采用多种方法进行降维,其中最优的算法不仅缩短了位串长度,而且提高了ML测试的效率。最后,Pharmacoprint产生了具有明确氢原子的三维(3D)结构作为输入文件应用到神经网络中,并使用监督自动编码器选择最重要的比特,使得可以最大化Matthews相关系数,最高可达0.962。相关的研究成果以 “Pharmacoprint: A Combination of a Pharmacophore Fingerprint and Artificial Intelligence as a Tool for Computer-Aided Drug Design” 为题发布在国际著名期刊Journal of Chemical Information and Modeling上。
Pharmacoprint考虑并评估了指纹作为ML实验输入数据的应用(图1)。
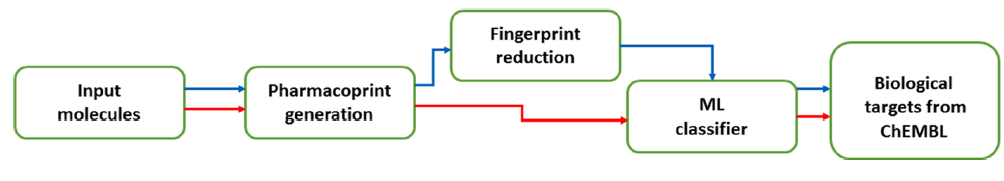
图 1. 本研究中所进行实验的一般方案。图片来源于JCIM
项目地址:
https://github.com/lstruski/Pharmacoprint
Pharmacoprint的工作流程
该算法的主要作用是识别分子中的药效团特征,并确定以键为单位的拓扑距离测量的内部特征距离。用户可自定义特征定义和距离仓。
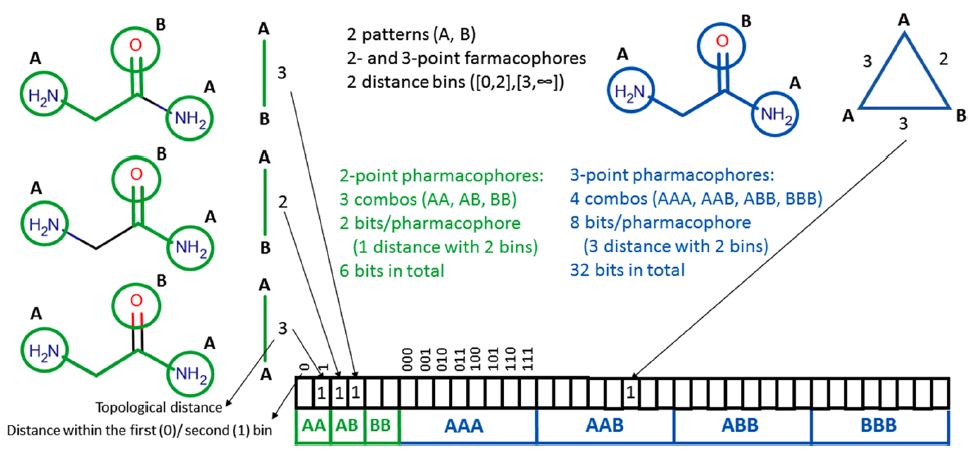
图2. Pharmacoprint生成方案,其中两个特征,两点和三点药效团,和两个距离仓定义一个38比特指纹。图片来自JCIM
为了说明算法是如何工作的,研究者假设只有两个药效团特征(图2中的A和B),两个或三个特征的所有可能组合,以及两个距离仓(两个或更少的键和两个以上的键)。两种药效团特征(A和B)可以组合成三对(AA、AB和BB)和四对三联(AAA、AAB、ABB和BBB)。特征对之间的距离可以认为是在第一个仓内(两个或更少的键;bin “0”)或第二个仓内(两个以上键;bin “1”),这意味着一个单一的两点药效团是由两个比特来描述的。三点药效团由8比特描述,因为三个内部距离,可以在第一或第二距离仓,可以产生8种可能的解决方案。这种表示的整个指纹由38比特组成。
在本研究中,为了测试Pharmacoprint的性能,研究者人员进行了以下设置:采用Gobbi和Poppinger最初列出的8种药效团特征类型(氢键受体、氢键供体、碱性基团、酸性基团、疏水基团、卤素、脂肪环和芳香环的连接点)和七个距离仓[(<1,3), (<3,4), (<4,5), (<5,6), (<6,7), (<7,8), (<8,100),以键为单位度量所有距离]。8个药效团特征、7个距离仓和所有的2点和3点药效团的组合,共产生了39973比特的指纹。
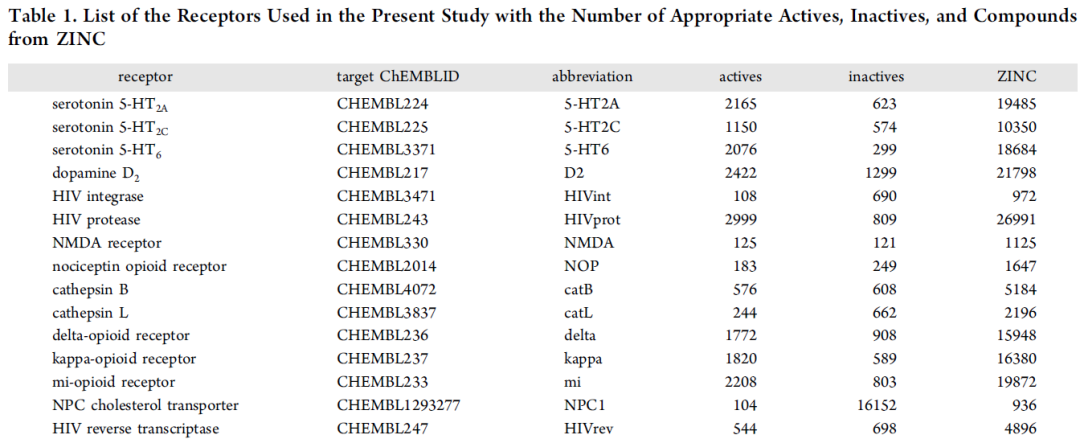
表1. 本研究中所使用的受体列表,以及ZINC中的活性数据和化合物的数量。图片来自JCIM
指纹比较
研究者比较了不同分子指纹图谱和药物学指纹图谱的效率。使用了两种非活性物质(ChEMBL的真正非活性物质和ZINC的假定非活性物质)。Pharmacoprint是通过以下设置生成的:无约简算法(所有39973比特指纹全部使用) ,生成氢原子位置确定的三维表征。所有计算均使用三种ML方法(SVM、LSVM和LR)进行。
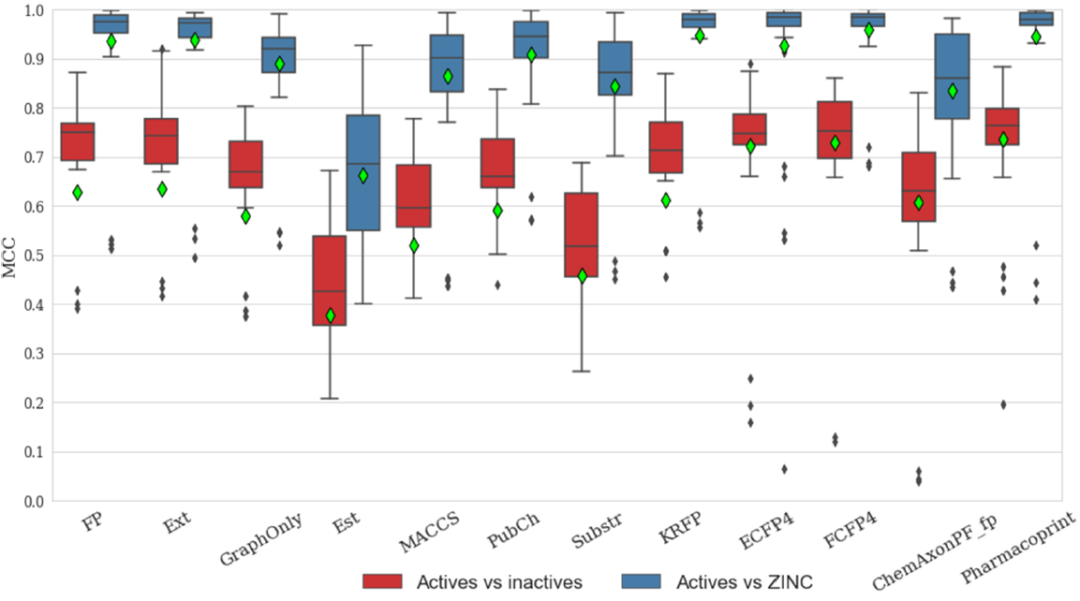
图3. 本研究分析的所有指纹图谱的MCC值箱图。图片来自JCIM
结果(图3)显示,Pharmacoprint在真假阳性化合物分类方面优于所有测试指纹。Pharmacoprint的中位MCC(Matthews相关系数,范围为-1~1)和平均MCC均最高(分别为0.766和0.736);然而,ECFP4和FCFP4的结果相似(ECFP4的中位MCC = 0.754,平均MCC = 0.729, FCFP4的中位MCC = 0.749,平均MCC = 0.722)。Pharmacoprint的性能与广泛使用的KRFP、ECFP4和FCFP4具有可比性(KRFP和Pharmacoprint的中位MCC为0.980,FCFP4为0.981,ECFP4为0.983)。
Pharmacoprint与ChemAxon PF fp比较(分别针对靶点和ML方法),结果显示出了Pharmacoprint的优势,45例中有37例MCC值较高。
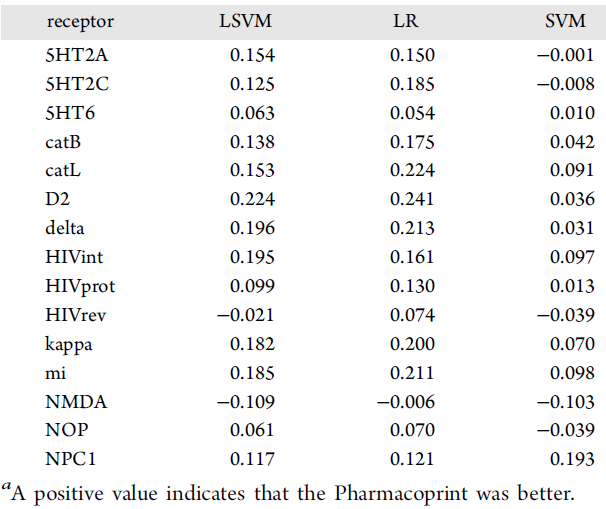
表2. 比较Pharmacoprint和ChemAxon PF fpa的MCC值(3种ML方法和15种不同靶点计算)的差异。图片来自JCIM
所有结果在p = 0.05的显著性水平下,经Friedman检验进行了统计学分析。结果证明,ECFP4和FCFP4与Pharmacoprint的差异在统计学上并不显著。Pharmacoprint与其他三种结构指纹图谱(Ext、FP、KRFP)的差异也无统计学意义,但在等级上差异较大。六个剩余的指纹图谱,已证明在统计上比Pharmacoprint差。
输入分子表示的影响
随后的实验只进行了Pharmacoprint表示,没有用无约简算法来阐明化学结构,结果(图4)表明在Pharmacoprint生成前,不管应用何种ML方法,适当准备化学结构是获得高质量结果的关键。因此,为了保证结果的最高质量,化合物应制备为具有明确氢原子位置的三维结构。而在Pharmacoprint中,当药效团特征的数量增加2倍时,比特数就会增加10倍。这样的分子会引入很多噪音。当化合物从2D转换为3D时,观察到结果的质量得以提升,从而确保了高分子量化合物的消除。因此,为了获得高质量的结果,自动截断是必须的步骤,但转换为3D数据本身并不是必须的。综上所述,初始数据准备是耗时的;然而,必须最大限度地利用这些数据,来创建机器学习预测模型的统计参数。
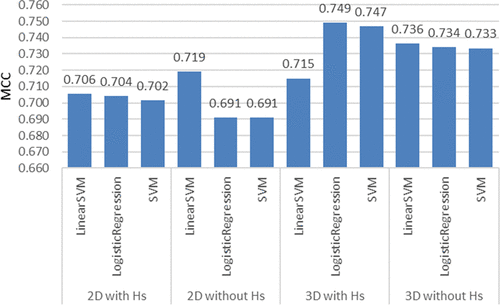
图 4. 在三种不同的机器学习方法的分类实验中获得的输入分子的不同表示的平均MCC值(15个不同的生物靶点计算)。图片来源于JCIM
靶点依赖性
在分析不同靶点的结果时(图5),可以发现无论采用哪种ML/约简算法组合,一些靶点的结果都优于其他靶点的结果。特别是,对活性化合物数量最多的生物靶点(如delta阿片类受体或5-羟色胺-HT6受体)的检测结果较好。NMDA和NPC1受体的活性成分相对较少,结果最差。
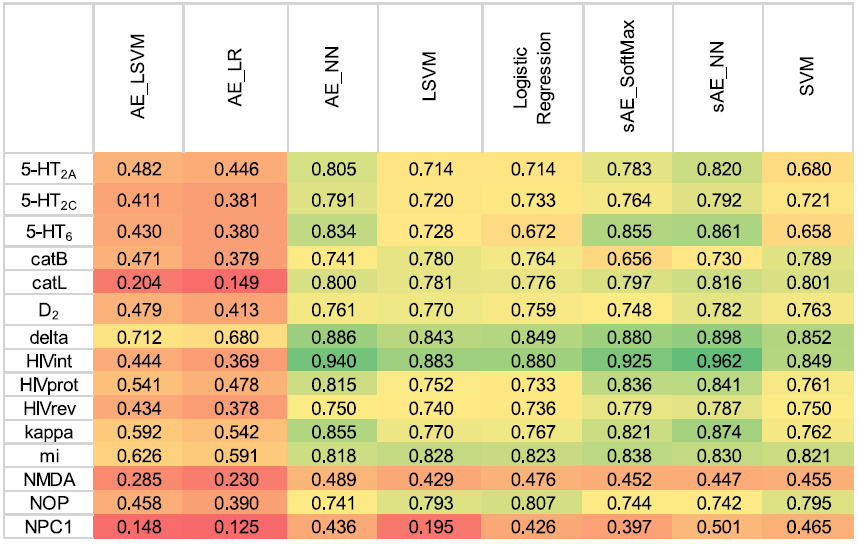
图 5. 通过不同的机器学习方法和约简算法获得不同生物靶点的MCC值。图片来源于JCIM
然而,HIV整合酶抑制剂却没有观察到类似的结果,尽管和较差靶标的活性化合物的数量相似,但创建的分类模型却非常有效 (MCC值达到了0.962,这是本研究中所实现的最大值)。与HIV整合酶抑制剂相比,在NPC1活性和非活性之间观察到更高的内部差异,但在这种情况下,不良结果是由于活性和非活性之间的数量失衡(约1:161)。
以上结果表明,采用监督式自动编码器对指纹进行约简是最佳的方法;对于大多数靶点,该方法获得的MCC值最高,与神经网络结合尤其有效。
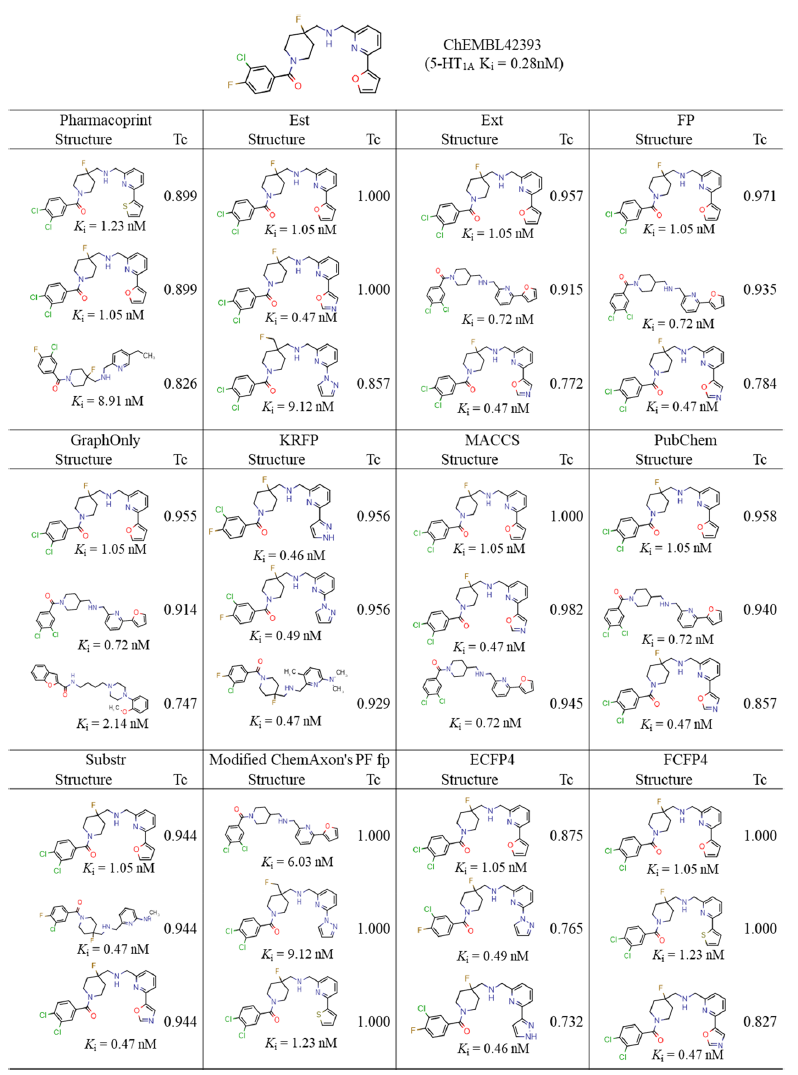
图 6. 本研究分析的所有5-HT1AR活性化合物中活性最好的化合物ChEMBL42393的结构以及相似性搜索的每个指纹类型中最相似的三个化合物。图片来源于JCIM
结论总结
本文介绍了一种描述化合物结构中药效团特征呈现的指纹图谱。Pharmacoprint是最长指纹之一;在一定设置下生成的完整指纹由39973比特组成,但这是很容易缩减的。当作为ML方法的输入时,这种表示优于其他11种常用的分子指纹中的9种,并在分离真正的活性和非活性上返回与ECFP4和FCFP4相似的结果。
如果Pharmacoprint是由定义了氢原子的3D输入结构生成的,并将其作为具有监督自动编码器功能的神经网络的训练数据,则在分类测试中可以获得高达0.962的MCC值。
参考文献
Dawid Warszycki, Łukasz Struski, Marek Śmieja, Rafał Kafel, and Rafał Kurczab, Pharmacoprint: A Combination of a Pharmacophore Fingerprint and Artificial Intelligence as a Tool for Computer-Aided Drug Design, Journal of Chemical Information and Modeling Article ASAP. DOI: 10.1021/acs.jcim.1c00589.