

IsAb是一个专注于抗体重新设计的计算协议,主要是优化抗体序列。协议全面且免费,方便来自不同领域的用户快速上手。
背景介绍
抗体重新设计是在已知抗体序列及结构的基础上对其结构进行修饰。由于重新设计是基于已有的抗体,特别是经FDA验证的抗体,因此设计的抗体产生免疫原性的可能性较小,可以更快地进行临床试验。重新设计很大程度上依赖于天然蛋白或交界面,主要侧重于对抗体-抗原复合物的表面或内部进行改造,以获得更好的亲和力或提高复合物的稳定性。体内亲和力成熟是获得更好的抗体性能的重要方法,在免疫应答期间通过高频突变(SHM)可将其亲和力提高数倍。匹兹堡大学的谢祥群教授团队开发的IsAb突破了结构的限制,即使在抗体-抗原复合物结构信息有限时,也可快速、免费地进行抗体设计。
抗体设计协议的工作流程
1. 获得抗体和抗原的结构。这些结构可以来自PDB/自主解析。否则,可以从IMGT检索抗体序列。
2. 使用RosettaAntibody web服务器进行抗体建模。可通过抗体序列生成其结构,并输出一个松驰的结构。
3. 采用RosettaRelax方案,精修抗体和抗原的结构并提高对接精度。
4. 抗体与抗原对接。如果有抗体和抗原之间的结合信息,用户可以跳转到第5步。如果结构信息有限,抗体和抗原的结构可以提交到ClusPro来进行全局对接。
5. 局部对接。基于全局对接结果,使用SnugDock进行局部精修对接,允许交界面侧链和CDR loop环的柔性,并输出最终的抗体-抗原复合物。
6. 基于精修的抗体-抗原复合物进行丙氨酸扫描:(i)先将抗体和抗原界面上的残基突变为丙氨酸,然后(ii)计算突变过程中残基的能量变化,找出抗体上的热点(hotspots),这有助于后续的抗体设计或为未来的研究提供思路。
7. 采用抗体亲和力成熟方案设计抗体。基于Rosetta评分函数,抗体亲和力成熟方案将生成比原抗体具有更好亲和力和稳定性的最佳突变抗体。IsAb方案中所有的代码均进行了优化,以适应特殊要求。
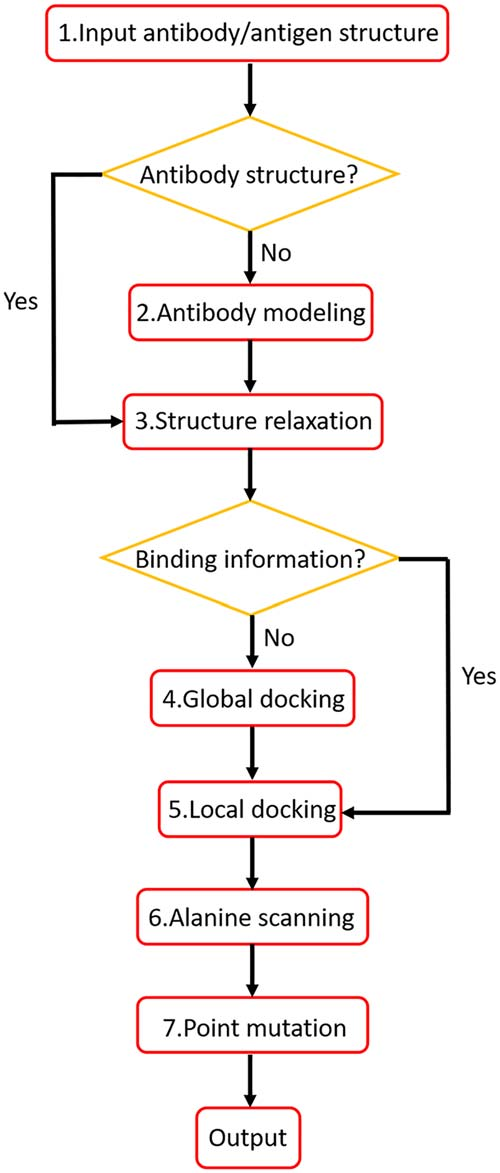
图1 工作流程。图片来源:BIB
方案设计完成后,作者首先使用IsAb重新设计抗体D44.1,并将预测结果与报道数据进行比较,结果显示,IsAb设计的结果可以较好地与实验数据重叠,说明IsAb是一种可靠的抗体设计方案。随后,作者以PD-1检查点抑制剂Cemiplimab为例进行了重新设计。
案例学习
Cemiplimab(西米普利单抗)设计步骤
Cemiplimab是FDA批准的一款PD-1免疫检查点抑制剂,用于治疗皮肤鳞状细胞癌。然而,Cemiplimab没有任何报道的三维晶体或者冷冻电镜结构。另外,PD-1在不同情况下解析的结构显示出其结构具有很大的柔性(图2)。PD-1的C’D,N和FG loop环在与不同的抗体发生相互作用后可能发生巨大的变化。其与Cemiplimab的结合数据也暂无报道。因此,Cemiplimab的例子是处于没有结构也没有结合数据的极端情况。
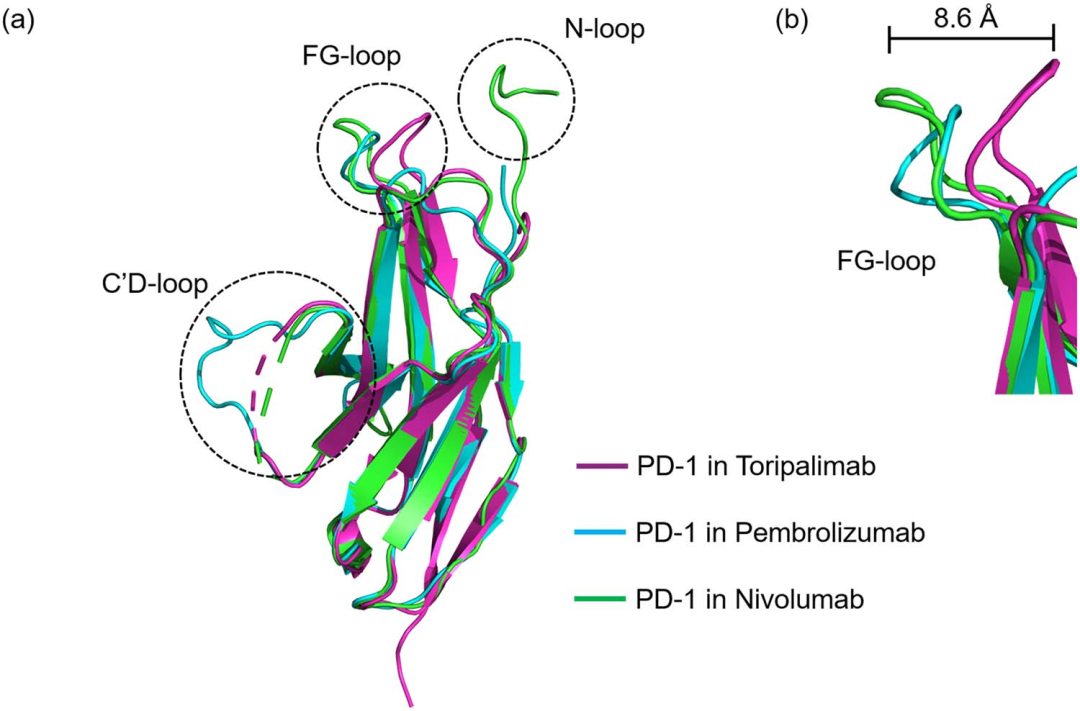
图2. 三个不同PD-1/抗体复合物的PD-1结构的重叠图。图片来源:BIB
第1-3步:PD-1和Cemiplimab结构的生成和准备
首先,作者收集了PDB数据库中的PD1-抗体复合物晶体结构,并从其复合物晶体结构中提取PD-1结构,生成了三个代表性的结构。从IMGT获得Cemiplimab的Fv序列,并提交至ROSIE。利用RosettaAntibody功能模拟Cemiplimab的三维结构,使用RosettaRelax方案准备抗原结构。随后,将三个结构分别与Cemiplimab(Fv区域)进行对接。选择10个输出结果中得分最好的结果作为松弛结构。
步骤4:Cemiplimab -PD1全局对接
由于没有Cemiplimab-PD1复合物的实验数据,作者首先使用全局对接来生成表位信息和可能的结合位点。为了提高全局对接的准确性,作者将Cemiplimab的序列提交到Robin等人开发的抗体结合部位预测数据库中预测位点。随后,在Cluspro中输入3个可能的PD-1结构以及预测的抗体结合位点,进行全局对接。作者从10个得分最高的结果中选取了可能的结合构象。每个对接特征将选择一个可能的结合构象用于局部对接输入。共选择了三个结果,分别命名为模型1、2和3(图3)。
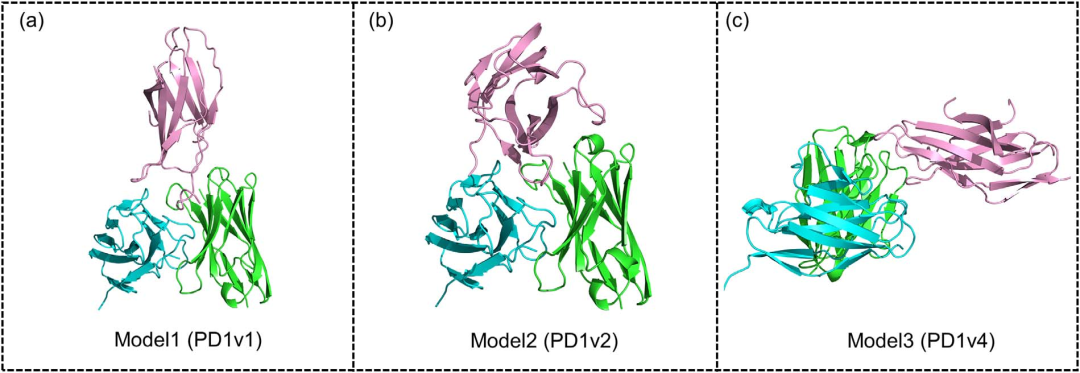
图3. 全局对接生成的三种可能的结合姿势。 图片来源:BIB
如果已知抗体结合部位和表位,则可用PyMol构建局部对接的起始姿势。对于计算方法来说, Parapred、Antibody i-patch以及一些像IEDB、Robetta等的web服务器是预测结合位点及表位的有效方法。结合部位以及表位的信息对于全局对接来说并非必不可少,但这些信息在对接中可以提高对接的准确性。结合部位和表位预测得越准确,起始构象的质量就越好。
第5步:Cemiplimab-PD1 局部对接
为了精修全局对接的结果,使用ROSIE web服务器的SnugDock 功能进一步进行局部对接。对接结果的接受标准为局部对接形成一个对接漏斗(N5>3)。如果对接成功,并且最低I_sc 结果达到全局对接步骤提及的三个标准,那么最低的I_sc结果就是最终的局部对接结果。综合以上规则,作者认为模型1和模型2的局部对接是成功的。
随后,作者将模型1和模型2的序列与已发表的PD-1抗体结构进行了比较。结果表明,模型1的序列与nivolumab具有较高的相似性,轻重链相似性分别为64%和76%。重链具有更高的结合频率,并在与抗原结合时提供更高的能量。因此,作者选择模型1作为最终的结合构象。
结构分析表明PD-1的BC、N、FG和DE loop环均参与了与cemiplimab的相互作用。复合物交界面包含9个氢键和疏水相互作用。与跟nivolumab的结合不同,PD-1的 N loop环在与cemiplimab结合时没有起到主导作用。相反,BC loop环在与Cemiplimab的相互作用中贡献了大量的氢键相互作用(图4)。
尽管N loop环没有在结合中扮演主要角色,但其依旧贡献了三个氢键作用。由残基能量贡献可知,重链上的残基Y59、D57和N31对cemiplimab-PD-1复合物的能量贡献较大(-3.697 kcal/mol、-2.721 kcal/mol、-2.118 kcal/mol)。此外, cemiplimab的Y59、D57、N31与PD-1的R30、N58/N102之间的氢键相互作用很强,其距离分别为2.6 Å、2.9/3.0 Å和3.4/3.0 Å。另一方面,N loop环上的P28和BCloop环上的R104与LCDR3和HCDR1间形成了强疏水作用。
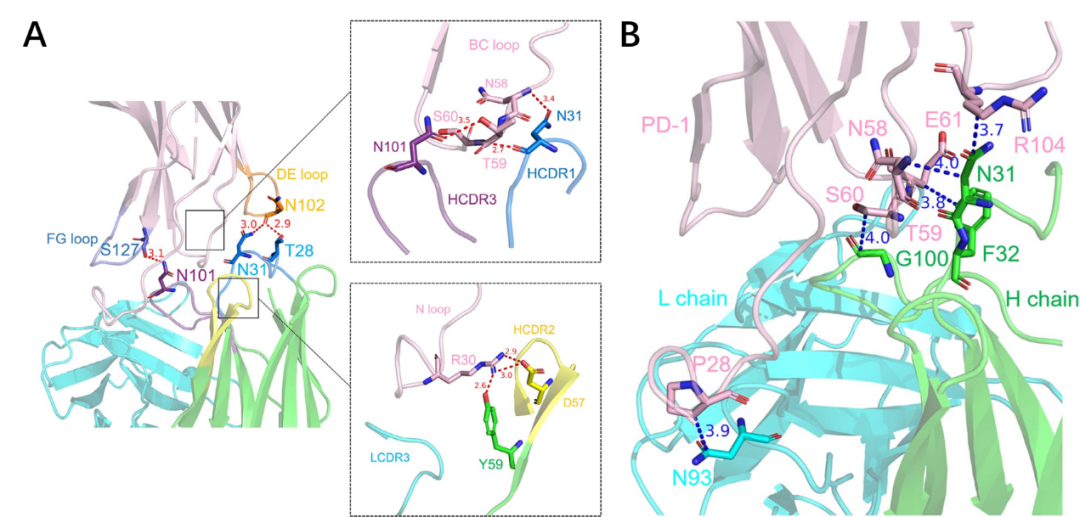
图4. Cemiplimab-PD1复合物间(A)氢键和(B)疏水相互作用。图片来源:BIB
无论采取何种方式获取输入结构,输入结构与天然结构越相似、对接的结果就越好。为了在Cemiplimab例子中进行对接结果过滤,序列比对可以帮助预测可能的最终复合物结合姿势。在某些情况下,可能会有不同类型的对接结果(抗原结合在抗体的不同位置上,或者抗原以不同的结合姿势结合在相同位置)。序列比对可能是决定最终构象的一个选择。此外,如前所述,重链在抗体-抗原相互作用中起着重要作用。因此,作者建议在序列比对中优先考虑重链的序列相似性,特别是CDR区域的序列比对。
第6步:计算丙氨酸扫描
采用丙氨酸扫描预测cemiplimab-PD-1交界面热点后,作者获得了多个交界面氨基酸残基的△△G。位于HCDR3上的两个热点残基ASN101和ILE102,其△△G分别为1.799和1.552 kcal/mol。突变为Ala使得N101失去两个氢键,这可能是结合能发生较大变化的原因。相反,I102与PD-1没有形成氢键,但仍然导致了很大的能量变化。这意味着I102可能对复合物的稳定做出了贡献。
第7步:Cemiplimab抗体计算的亲和力成熟
基于对cemiplimab的了解,按照抗体亲和力成熟方案进行计算的突变。作者首先尝试将交界面残基突变为20个蛋白氨基酸。cemiplimab上有17个残基被突变,其中11个残基来自重链。为了更清楚地查看结果,作者计算了突变的氨基酸残基的分数变化。Y103A、I102T、G100D及F27S的突变在重链的突变残基中具有最高的分数变化,N93Y、F32K的突变在轻链的突变残基中具有最高的分数变化,大多数突变残基位于cemiplimab的CDR区。这一结果与体内SHM的大部分突变主要积聚在cemiplimab的CDR和框架区域的现象一致。作者选择分数变化最高的残基来进行点突变。由于CDR H3区域在结合中起着重要作用,作者决定对H:Y103、I102、N101及G100残基进行点突变。预测的结果显示,两种突变相对于原始氨基酸来说,其总体得分、结合亲和力以及稳定性均更优。为了验证这些预测,作者对这四种突变进行了MD模拟和MM/GBSA计算。
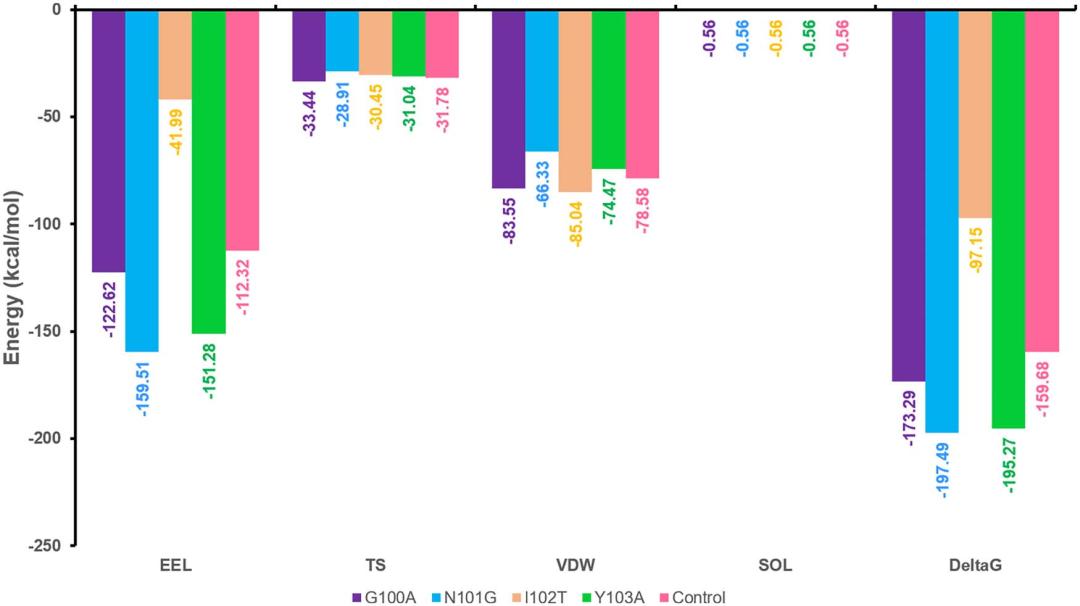
图5. 四种突变体的MD模拟结果和Cemiplimab对照的对比。图片来源:BIB
图5显示,突变体G100A、N101G和Y103A提高了cemiplimab的结合能(总结合能-159.68 kcal/mol),而I102T降低了cemiplimab的总结合能。有趣的是,结果表明,这三种体系比突变前的cemiplimab具有更好的静电属性,表明突变结构可能形成更多的极性的短程相互作用。G100A相对于对照组具有更高的构象熵变化和范德华能,这意味着G100A允许cemiplimab与PD1之间形成强的范德华相互作用。根据结构分析,作者发现N101G突变丢失两个氢键(图6)。对于疏水相互作用来说,G100A突变体依旧与PD1之间形成4Å的疏水相互作用。IsAb预测与MD模拟结果之间的重叠表明了作者的方案是可靠的。
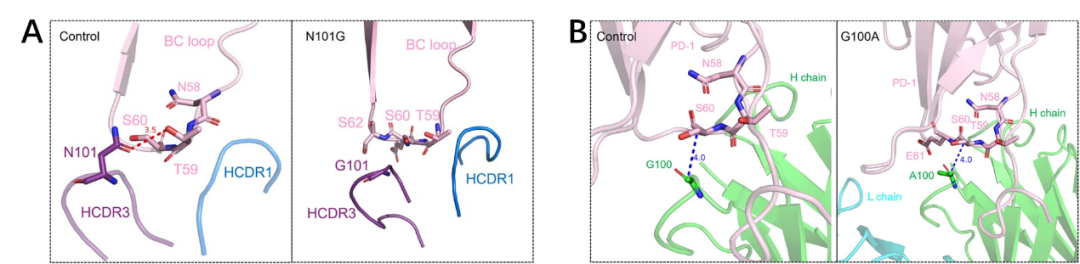
图6. 对照组和设计组之间(A)氢键和(B)疏水相互作用的比较。图片来源:BIB
参考文献
Liang T, Chen H, Yuan J, Jiang C, Hao Y, Wang Y, Feng Z, Xie XQ. IsAb: a computational protocol for antibody design. Brief Bioinform. 2021 Sep 2;22(5):bbab143. doi: 10.1093/bib/bbab143. PMID: 33876197.