
一种适用于大规模虚拟筛选的机器学习管道,可处理不断增加的类药化合物库,并区分真阳性和假阳性。
背景介绍
在过去的数十年里,虚拟筛选技术在药物发现中越来越普及,这很大程度上是由类药分子的不断增长和计算机技术的进步共同推动的,但至今其发展仍受到两个方面的阻碍。首先,目前商业化合物的数量将很快达到1011-1012个分子,而人类预估类药物分子的数量约为1060。将如此庞大的化合物库与特定的药物靶点对接是一项艰巨的任务。其次,对接能很好地筛选出非活性化合物,但会产生过多的假阳性。
处理这类庞大化合物的策略之一是库修整,这样不会损失潜在的命中率。机器学习虽然已在药物发现应用中,尤其是化合物分类及辨别对接结果的真假阳性方面(例:vScreenML)显示出强大的功能,但据目前所知,它们还没有被用于库修整。
如今,分子动力学(MD)模拟越来越多地被用于否决对接的假阳性结果。混合的QM/MM模型兼具了量子力学的精确性和分子力学的高效性,为研究蛋白-药物复合物提供了强有力的工具。本文作者之前的工作已经证明了QM/MM MD模拟在选择对抗SARS-CoV-2主要蛋白酶Mpro的抑制剂方面的成功。QM力场是ANI-2x,它是由一个神经网络根据密度泛函理论对数百万小分子进行训练的。研究者的ANI/MM MD模拟,获得了药物发现工作流的完整过程。该工作流从对接的1615个FDA批准的针对Mpro的药物开始,首先通过经典MD模拟,然后通过ANI/MM MD模拟将对接命中进一步过滤,最后预测了9个Mpro抑制剂,其中至少有3个是已报道的活性抑制剂。
主要内容
本文中,来自伊利诺伊大学芝加哥分校的Huan-Xiang Zhou教授等人报道了一个用于大规模虚拟筛选的机器学习管道。该管道的两个核心部分是(1)通过聚类进行库修整;(2)通过密集神经网络(DNN)将对接选择的命中结果分离为真阳性和假阳性。研究者通过筛选RPN11抑制剂、蛋白酶体的去泛素酶亚基(图1)和乳腺癌的药物靶点来阐述这一管道。作者调整了之前的工作流程,从而得到了8种RPN11抑制剂。相比之下,在显著降低计算成本的情况下,机器学习管道发现了其中的6个抑制剂。相关的研究成果以 “Machine Learning-Enabled Pipeline for Large-Scale Virtual Drug Screening”为题发布在国际著名期刊Journal of Chemical Information and Modeling上。
通过全对接和昂贵的MD模拟筛选RPN11抑制剂
这个工作流程涉及对接的240万个化合物,并通过昂贵的经典和混合量子/经典MD模拟来评估命中,获得了8个真阳性(图1A)。然后,研究者开发了一个机器学习管道,其中库在对接前缩减了10倍,DNN被训练来区分真阳性和假阳性。
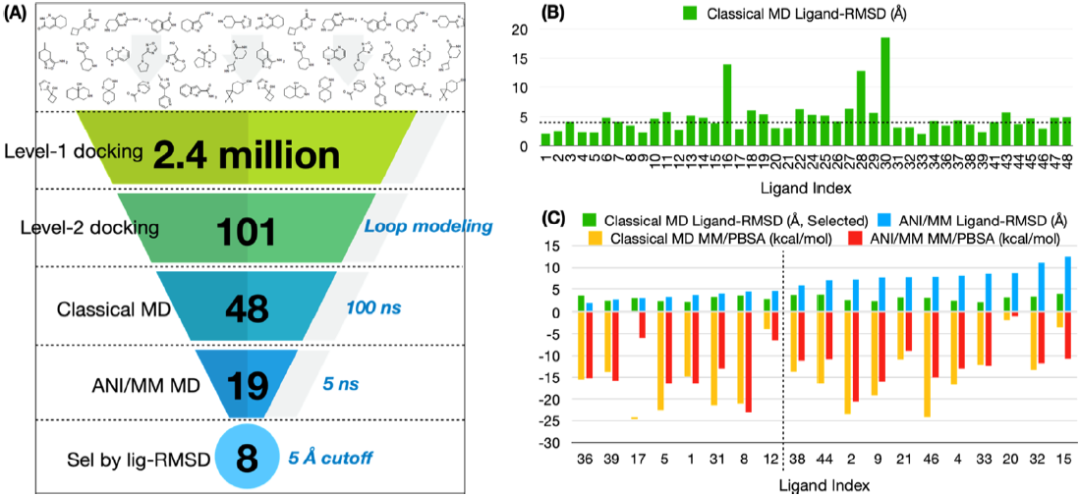
图1. 通过全对接和经典MD模拟筛选RPN11抑制剂。图片来自JCIM
研究者使用Autodock Vina将240万化合物中的每一个与从ZINC15网站上的ChemDiv和Asinex库中提取的可对接的化学结构与RPN11刚性对接。在这个“一级”对接中,对每个化合物生成的10个构象选出Vina评分最好的一个。根据Vina评分(最佳评分为−9.9 kcal/mol)对化合物进行排名,并以−9.2 kcal/mol为截断值筛选出101个化合物。
其次,研究者为Ins-1环生成了19种额外的构象,并进行了二级对接,其中一级对接选择的101种化合物中的每一种都和其他19种Ins-1构象一起与RPN11对接。在101种化合物中,研究者选择了48种化合物,它们在20种Ins-1构象中至少有6种的Vina评分高于−9 kcal/mol。
剩下的任务是在48次测试中区分真阳性和假阳性。这是分两步完成的。首先,研究者进行了100 ns的经典MD模拟。在MD模拟中,真阳性预期是稳定的,而假阳性预期是在结合位点移动或离开结合位点,导致高的配体-RMSD值。因此,研究者计算了模拟的20到100ns部分的平均配体-RMSD(图1B),并使用4 Å的截断值将19个命中定义为阳性,其余25个为阴性。在第二步中,对19例阳性样本进行了5 ns混合ANI/MM MD模拟。最后,基于5 Å的配体-RMSD截断,研究者选择了8个配体作为真阳性(图1C)。与之前对SARS-CoV-2主要蛋白酶的研究类似,ANI/MM MD模拟改善了大多数真阳性(8个中的5个)的结合自由能,但削弱了大多数假阳性(11个中的9个)的结合自由能(图1C)。
用k-均值聚类对全库进行修整
研究者从ZINC15网站的101种配体中的97种的一级对接中(图2A)提取PDARB。其余四种配体,研究者使用RDKit包获得了PDARB。根据PDARB计算的距离进行层次聚类,101个配体分为3种聚类,分别为25个、48个和28个配体(图2B)。通过对二维结构和物理化学性质的考察,研究者验证了同一组中配体是相似的 (图2A,C)。组I是高HBA和RB;组II log P高,HBA低;组III在HBD和Ring中含量较高。由此,验证了通过对接选择的阳性确实形成了少量聚类且PDARB用于聚类是有效的。
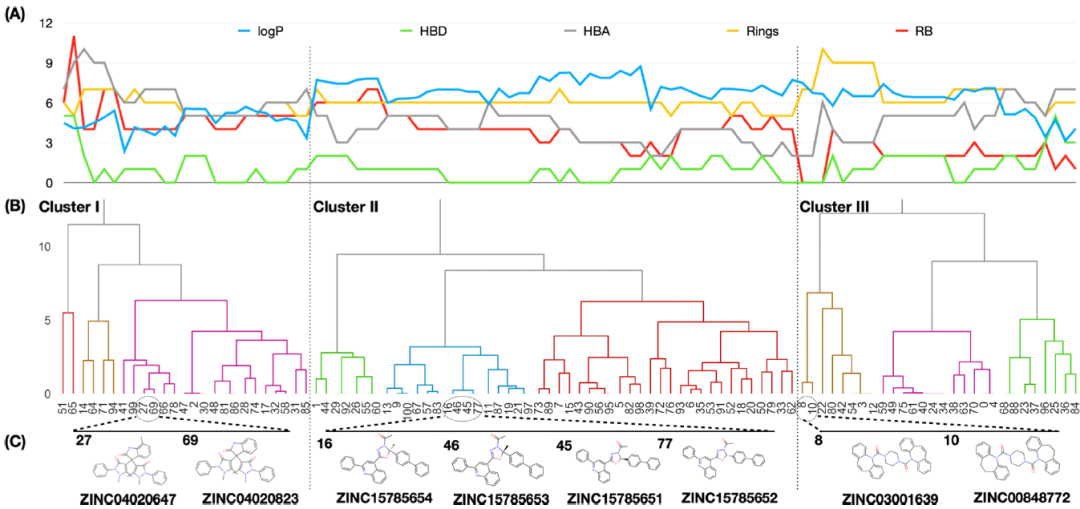
图 2. 101种化合物的层次聚类。图片来源于JCIM
随后,研究者从ZINC15网站上提取PDARB,以获得初始库中的240万个化合物,并成功提取了130万个化合物。研究者设定了10倍缩减的目标,并使用k-均值聚类化合物(图3A)。
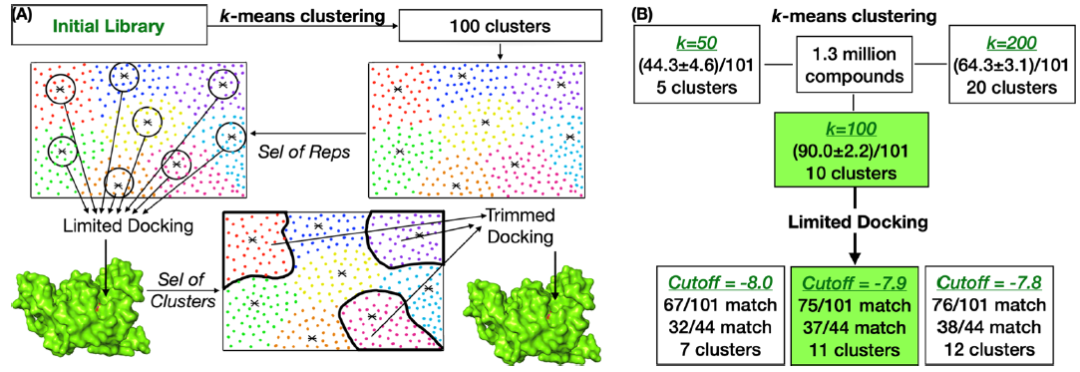
图 3. 在RPN11靶标上的库修整和工作流程阐述。图片来源于JCIM
第一步是找到最优的组数量(“k”)。为此,研究者将130万个化合物聚成50、100或200个组,并根据找到的101个配体的数量对组进行排序(图3B)。然后,计算了前10%的聚类的总召回率。当k = 50、100和200时,总召回率分别为44.3±4.6、90.0±2.2和64.3±3.1。这些结果表明,k = 100是最优选择。
接下来,当k = 100时,研究者根据每个群组中少数配体的Vina评分,选择10个左右(即k的10%)的组。具体来说,在每个组中,选择最接近簇心的10个配体,并获得它们的Vina分数。然后,为组调整了Vina分数的截断值。当10个配体中Vina得分低于截断值时,选择一个组。当截断值分别为−8.0、−7.9和−7.8 kcal/mol时,选择的簇数分别为7、11和12,由此产生的11个组召回了101个配体中的75个。在101个配体中,44个通过二级对接,并通过100 ns MD模拟进行了评估。在这44个配体中,有37个被11个选定的组召回。有趣的是,7个没有被10倍缩减库召回的配体,最终被100 ns MD模拟(6/7)或ANI/MM MD模拟(1/7)消除。因此,基于聚类的裁剪将库的大小减少了10倍,且不损失任何真阳性。
用DNN分离真阳性和假阳性
通过对接10倍缩减后的库以选择命中并分离真阳性和假阳性,仍然是一个重大的挑战。DNN可以解决此问题,作者准备了两个截然不同的化合物子集来训练DNN。A组由顶级的Vina评分者组成;它们的阴、阳性分类是基于配体-RMSD的一个短时(10 ns) MD模拟。B组是好的和坏的Vina得分的配体的混合;他们的阳性或阴性的分类是基于Vina的分数。
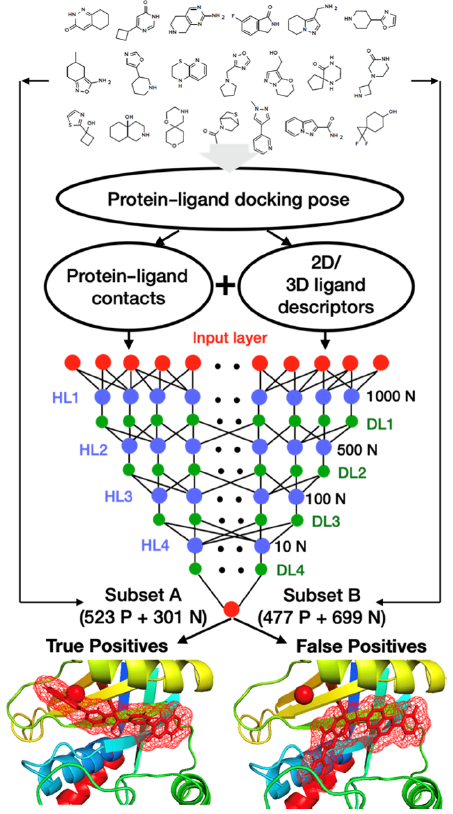
图 4. DNN用于命中分类。图片来源于JCIM
研究者通过100 ns MD模拟评估了44种配体,其中19种为真阳性,其余25种为假阳性。以这44个配体作为测试集,结合2000个配体训练的DNN预测了13个真阳性和31个假阳性,其中9个和21个是正确的,准确率为68.1%。此外,根据ANI/MM MD模拟19个配体的评估,8个预测的真阳性有6个是正确的,11个预测的假阳性有8个是正确的,在19个ANI/MM评估的配体的测试集中,准确率为73.7%。相比之下,仅在子集A上训练DNN时,44 个100 ns MD评价配体的测试集的预测准确率较低,为61.4%,这再次表明子集B被纳入训练DNN时的准确率有提高。以19个ANI/MM评估的配体作为测试集,在训练中省略子集B并不影响整体准确性,但预测的真阳性少了一个,多了一个正确的假阳性预测来补偿。
结论总结
本文报道了一个可用于大规模虚拟筛选的机器学习管道。通过混合ANI/MM MD模拟,研究者确定了8个新的化合物可作为潜在的RPN11抑制剂。此文提出的机器学习流程,将有助于开发针对RPN11和其他靶点的潜在活性化合物。
参考文献
Aayush Gupta and Huan-Xiang Zhou, Machine Learning-Enabled Pipeline for Large-Scale Virtual Drug Screening, Journal of Chemical Information and Modeling Article ASAP. DOI: 10.1021/acs.jcim.1c00710.