

一种集成了多个深度学习和基于协同进化的接触图方法——C-QUARK,在非同源蛋白结构建模领域取得了极大进展,正确折叠的蛋白数量是其他方法的6倍。
背景介绍
从头蛋白结构预测,通常指的是不使用PDB中的同源模板来建模蛋白结构的方法,在过去的几十年里引起了人们极大的兴趣。CASP(蛋白质结构预测的关键评估)实验,见证了这一方向的长足进展。例如直到10年前,从头算模型还只能折叠长度小于100个残基的小蛋白质,但在最近的CASP实验中,包括Rosetta和QUARK在内的几个先进的pipelines,对长度大于100个残基的靶标都生成了正确的折叠。这些进展主要归功于能量力场和搜索引擎的发展,这些引擎有助于在模拟折叠过程中获得接近自然状态的全局最小能量。但目前的力场和搜索引擎,往往无法捕捉到蛋白质中精确的远程原子相互作用。因此,基于从头算折叠方法的复杂拓扑大蛋白建模的精度还有待商榷,而且远远不能令人满意。
克服从头算模型局限性的有效方法之一,是引入远程接触,即把在蛋白质序列中被至少24个残基分开的接触作为折叠模拟中的约束。残基间接触的先验知识有助于将构象搜索限制为接近自然态,但需要足够高的接触预测精度。接触图预测的早期研究,主要集中在协同进化和机器学习上,但由于接触图预测的准确性有限,接触图预测对从头构造折叠的影响不大。直接耦合分析(DCA)和深度神经网络学习技术的引入,使接触预测的精度有了质的飞跃。DCA有助于从多序列比对(MSAs)中去除平移接触噪声,而监督深度学习技术则从PDB结构中学习内在接触模式,这些结构是从MSAs衍生的协同进化特征开始的。尽管接触预测取得了显著的进展,但除非预测的接触与折叠模拟有效地结合起来,否则当前从头算建模协议无法发挥其全部潜力。特别是当序列较长时,基于序列的接触预测精度会偏低,如何平衡嘈杂的接触图与先进的折叠模拟力场,构造正确的从头算结构折叠,仍是一个重要且具有挑战性的问题。
主要内容
基于序列的接触预测,在辅助非同源结构建模方面显示了相当广的前景,但它往往需要足够数量的正确接触才能实现正确的折叠。为此,来自美国密歇根大学的Yang Zhang等研究者,在QUARK的基础上进行了扩展,开发了接触引导的从头算折叠程序C-QUARK;QUARK是自2010年以来,一直被评为CASP实验的顶级方法之一。采用该方法对247个非冗余蛋白进行检测,在TM评分(模板建模评分)≥0.5的情况下,C-QUARK可以折叠75%的范例,是QUARK的2.6倍。对于接触精度低或同源序列少的59个案例,C-QUARK正确折叠的蛋白质数量比其他基于接触的折叠方法高6倍。同时,C-QUARK在第13次CASP(蛋白结构预测的关键评估)实验中的64个自由建模靶点上进行了测试,其平均GDT_TS(全局距离测试)得分比最佳CASP预测因子高5%。相关的研究成果以 “Improving fragment-based ab initio protein structure assembly using low-accuracy contact-map predictions” 为题发布在国际著名期刊Nature Communications上。
C-QUARK在从头算结构预测方面明显优于QUARK
基于最先进的从头算蛋白质折叠模拟程序QUARK,C-QUARK首先从全基因组和宏基因组序列数据库中收集多序列比对(MSA),其中两种接触图由深度学习和基于协同进化的预测器创建。其次,从不相关的PDB结构中收集连续序列长度(1-20 AA)的结构片段,并在由基于知识的能量项组成的复合力场的指导下,通过副本交换蒙特卡罗(REMC)模拟组装全长结构模型,根据距离分布,从结构碎片中收集残基间接触,以及基于序列的接触图预测(图1)。
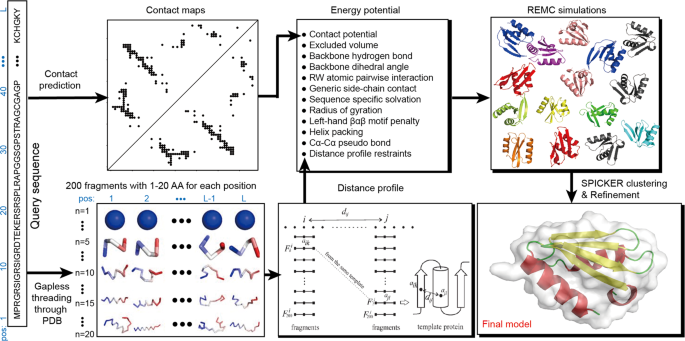
图 1. 用于接触引导从头算蛋白质结构预测的C-QUARK流程图。图片来源于Nature Communications
表1总结了折叠结果,其中C-QUARK的第一个模型的平均TM-评分(模板建模得分)(0.606)比QUARK(0.423)高43%。这一差异对应的p值为6.8× 10-51,这表明从接触图预测的改善在统计学上是高度显著的。表1还列出了前5个模型中最好的结果,这些结果是基于SPICKER的decoy大小进行排名的,其中C-QUARK再次显著优于QUARK,其平均TM评分(=0.629)比后者(=0.468)高34%,p值为6.8× 10-47。
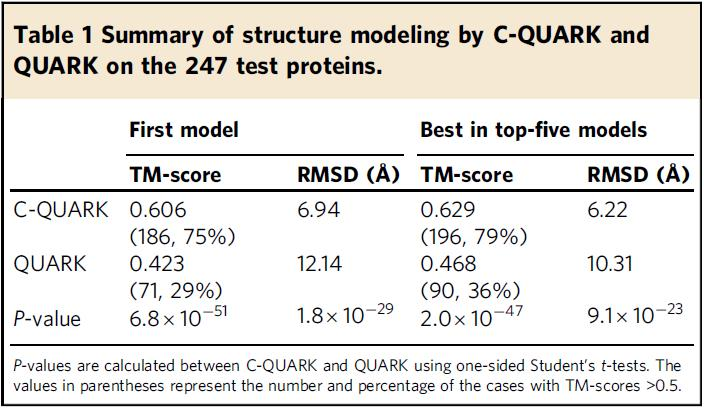
表1. 247个检测蛋白的C-QUARK和QUARK结构建模综述。表格来源于Nature Communications
为了检验C-QUARK在特定目标上的优势,研究者给出了图2a中与QUARK的头对头的TM评分比较。数据表明,在247个靶点中,C-QUARK生成的模型中有224个(212个)比QUARK生成的模型具有更高的TM评分(或更低的RMSDs)。当考虑前5个模型中的最佳模型时,两个程序生成的正确折叠数分别增加到196和90。相反,在46%的QUARK无法折叠的情况下,C-QUARK产生了正确的折叠,这表明接触限制在C-QUARK从头折叠蛋白质结构的过程中起着主要的积极作用。
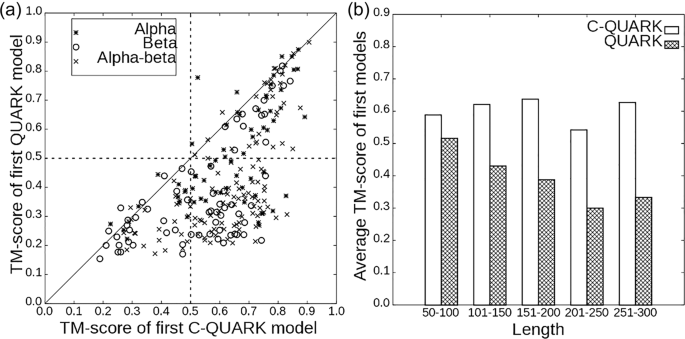
图2. 247个检测蛋白的C-QUARK模型和QUARK模型比较。图片来自Nature Communications
在图2b中,研究者进一步考察了C-QUARK和QUARK对不同长度蛋白质的折叠能力。对于长度在50-100、101-150、151-200、201-250和251-300之间的蛋白,C-QUARK模型的平均TM值分别为0.588、0.621、0.638、0.542和0.627,而QUARK模型的平均TM值分别为0.516、0.431、0.388、0.300和0.333。对于156个小蛋白,C-QUARK模型和QUARK模型的平均TM评分分别为0.607和0.467,对应C-QUARK模型的TM评分仅提高了30%。而91个大蛋白的改进率为74%,其中C-QUARK和QUARK的平均TM评分分别为0.604和0.347。此外,C-QUARK对小蛋白质和大蛋白质的平均TM评分具有很大的可比性(0.607 vs 0.604),而QUARK对大蛋白质的建模精度显著差(0.467 vs 0.347)。
案例研究揭示了中、长接触对具有复杂拓扑结构的折叠蛋白的重要作用
为了研究显著改进的原因,研究者给出了C-QUARK和QUARK模型与相应的本地结构的结构比较,以及图3中三个测试用例的接触图预测。第一个例子(PDBID: 2d7jA)是一个有188个残基的α – β蛋白,由11条β链、5条α -螺旋和一个310-螺旋组成。该结构域的核心是一个七股β片,其两侧被α螺旋包围(图3a)。图3d中的原生接触图显示,由于N-端(HN)和C-端(HC)的残基之间的长程相互作用(图3d中用矩形标记),螺旋在N-端(HN)和C端(HC)上的距离很近。大多数原生接触,包括保持两端螺旋在一起的远程接触,都能够正确预测,其中接触图预测精度为0.648,如图3d左三角形中的红色圆圈所示。这些预测接触的约束主要导致残基在C-QUARK模型中与原生结构具有相同的接触网络的排列,如图C-QUARK模型和图3d左三角形中带有蓝色圆圈的接触图所示。核心区域的接触限制,也有助于维持该区域的7条β链的整体拓扑结构。
因此,C-QUARK模型与本地模型非常相似,TM评分=0.793。另一方面,在QUARK模型中,由于缺乏N-和C-末端之间的远距离接触约束,在QUARK模型中,末端的两个α螺旋彼此距离较远。因此,QUARK模型的整体折叠和对应的接触图(图3d直角三角形中的蓝色圆圈)与本地有显著差异,导致TM评分较低,为0.295。这个范例强调了接触的重要性,特别是长期接触,可用于正确建模大型α-β蛋白。
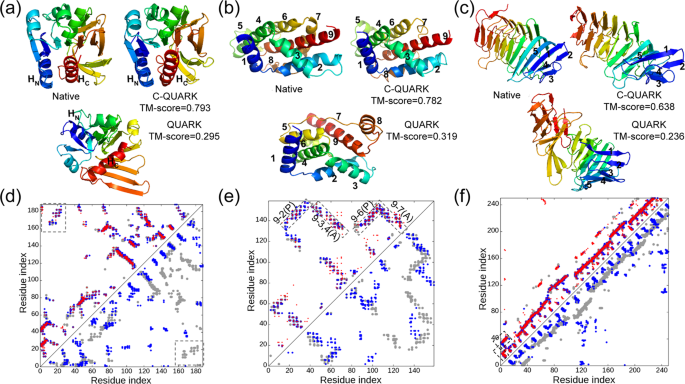
图3. 接触引导从头构造折叠的说明性范例。图片来自Nature Communications
结论总结
本文介绍了一个接触引导的从头算折叠程序,C-QUARK,它显示出了对在PDB中没有同源模板的“硬”蛋白建模能力的显著提高。重要的是,C-QUARK正确生成折叠的总体成功率约为75%,是QUARK(29%)的2.6倍,表明接触图预测在改进从头构造建模中的重要性。此外,C-QUARK显示出对长度为>150残基的大中型蛋白具有一致的折叠能力,这是几十年来从头算建模领域的挑战之一。
总体来说,虽然C-QUARK还有很大的改进空间,许多策略/组件正在开发中,但本研究的结果表明它能有效地将接触图约束与折叠非同源蛋白和远端同源蛋白的前沿折叠组装模拟相结合,具有强大和显著的应用优势。
参考文献
Mortuza, S.M., Zheng, W., Zhang, C. et al. Improving fragment-based ab initio protein structure assembly using low-accuracy contact-map predictions. Nat Commun 12, 5011 (2021). https://doi.org/10.1038/s41467-021-25316-w.