

全新Nsp1/SL1复合物结构的解出,首次揭露了Nsp1如何进一步通过招募40S核糖体来识别自身RNA进而参与翻译调控的方式,有助于开发新冠药物及减毒疫苗。
与其他冠状病毒(α-、β-、γ-和δ-冠状病毒)相似,SARS-CoV-2是一种包膜、单链、正义RNA病毒,已知可感染大量脊椎动物。SARS-CoV-2的基因组编码多达14个开放阅读框(ORFs)。ORF1ab编码了很多病毒蛋白,参与病毒形状或结构形成的结构蛋白负责感染宿主细胞,非结构蛋白则通过与宿主细胞间蛋白的相互作用参与其他细胞内的病毒运输。其中一个重要蛋白是非结构蛋白1(Nsp1),在大多数感染人类的冠状病毒中,包括SARS和MERS,Nsp1被证明是病毒复制和翻译调控所必需的。它通过两种方式攻击宿主mRNA:通过阻断核糖体的40S亚基来终止宿主蛋白的翻译,或在宿主mRNA的5’UTR附近进行内切,使其不能翻译。然而,由于Nsp1和病毒RNA在5’UTR区域的独特的结构性相互作用,病毒RNA对这种切割具有抗性。虽然近年来有关Nsp1在阻断宿主核糖体方面的报道较多,但有关SARS-CoV-2中Nsp1如何识别病毒RNA的关键区域(5‘UTR’)的机制尚不明确。另一方面,Nsp1在抑制宿主防御成分(如I型干扰素干扰素γ、白介素)的产生方面也发挥关键作用,它作为被感染细胞分泌的关键信号分子,可以提醒其他细胞病毒进入。
Nsp1在宿主细胞抗病毒防御机制(调节干扰素应答)中发挥了关键作用,并改变固有免疫系统,有利于病毒复制和免疫逃避,是一个潜在的治疗靶点。澳大利亚莫纳什大学的Wilma Jerom Lopes团队通过酶动力学及分子动力学明确了SARS-CoV-2的Nsp1和5’UTR之间的分子相互作用和复合物结构稳定性并通过对接等计算模拟手段探讨了潜在的抑制剂结构。
文章摘要图。图片来源于:JPCL
首先,作者使用在线比对工具Clustal Omega对SARS-CoV-2、SARS-CoV-1和MERS的5’UTR区域进行的多序列比对(MSA)证明了三者的相似性,推测可能存在相似的物理结合模式。接下来,作者使用两个独立的程序:SPOT-RNA和RNAfold 以理解完整的5’UTR的二级结构特征。
已有实验证实SARS-CoV-1和MERS的5’UTR的10-28号核苷酸与Nsp1相互作用。作者结合MSA,分别使用RNAComposer和simRNA程序对SARS-CoV-2的5′ UTR-SL1区域(7-33号核苷酸,以及延伸区域)的三维结构建模,初步分析二者可能的结合位点。随后,作者再次使用Clustal Omega进一步分析SARS-CoV-2、SARS-CoV和MERS之间的Nsp1蛋白序列的相似性和差异性。结果发现SARS-CoV-2与SARS-CoV-1的相似性高达84.4%,与MERS的序列相似度仅为17.7%,表明SARS-CoV-2的Nsp1其结构、功能、及物理和化学结合特性与同一分支的SARS-CoV-1相同,很可能与MERS不同。这暗示着可以用SARS-CoV-1的晶体结构作为同源模板来构建稳健的和经验证 (能量最小化和物理结合力优化)的虚拟SARS-CoV-2 Nsp1全长结构。作者以低分辨率的SARS-CoV-1 NMR结构为模板,使用SWISS-MODEL和I-TASSER从头建模。已经发表的SARS-CoV-1的Nsp1的部分结构以及Nsp1-FL结构的RMSD为0.38,说明了氨基酸端结构域总体结构的准确性。再辅以观察结果对SARS-CoV-2 Nsp1的RNA结合活性进行实验测试,强调其物理上的相互作用。
不同分支的病毒Nsp1同源物对不同RNA片段(茎环区域)结合的亲和力具有一定的范围,一些依赖于核苷酸长度而另一些具有序列特异性。同时,SARS-CoV-2 5’UTR区域的哪个RNA片段与Nsp1结合尚不清楚。SARS-CoV-2和SARS-CoV-1的Nsp1在表面电荷分布上具有最高的同源性,文献中同源蛋白SL1与Nsp1结合数据,结合文章的序列比对结果,作者试图利用酶迁移位移分析(EMSA)来评估SARS-CoV-2的Nsp1和RNA的SL1区域之间的潜在相互作用。如预期,体外翻译的5’UTR的SL1 RNA片段直接的、物理结合于Nsp1,全长纯化蛋白的亲和力较好,为0.18μM (图1A,C)。进一步证明SARS-CoV-2 Nsp1直接结合于病毒RNA的SL1区域。随后,研究者使用C端截短的Nsp1(1-133)(Nsp1△C)来测试其与相同RNA片段之间的亲和力,探索C端结构域对RNA结合的必要性。实验结果表明,Nsp1△C与SL1之间的亲和力降低近一倍,为0.31 μM,且在摩尔浓度比为1:2时结合趋于饱和(图1B,C)。Nsp1△C形成的RNA/蛋白符合物的拖尾更少,这可能是因为截短的形式更稳定,因为连接于linker区域的C端结构域高度柔性,可能导致结合亲和力较低。
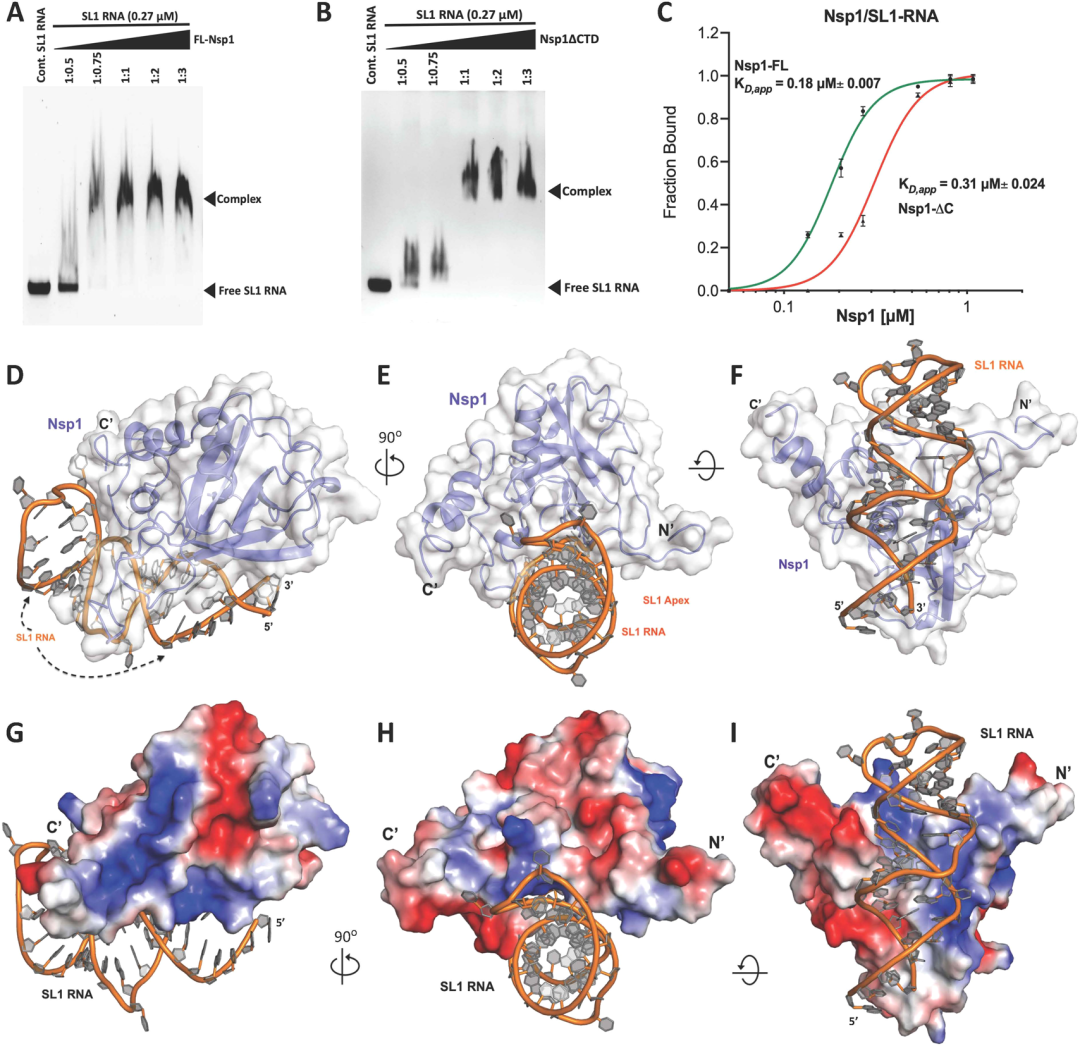
图1. SARS-CoV-2的Nsp1/SL1 RNA复合物的结构、表面电荷分布和亲和力的EMSA计算。图片来源于:JPCL
为了更好地理解SARS-CoV-2 Nsp1和SL1之间相互作用的结构和分子模式,作者使用分子动力学和基于选择性对接和计算模拟进行了虚拟结构研究。作者分别使用ModelX、MDock和HADDOCK 2.4进一步验证和精确对接复合物以明确二者之间的结合模式和相互作用。综合HADDOCK分数、可重复聚类规模、RMSD以及结合自用能(氢键、范德华力以及离子相互作用),选择可能的最佳模型。再使用Coot对该模型进行了验证及修正,以确保残基占据在最佳Ramachandran区域。随后将最后的对接复合物结构与初始的Nsp1单独模型结构进行比较,它们的Cα(中心碳原子)的总体RMSD为0.32 Å 。这表明对接时没有大的构象变化。
对接的Nsp1-RNA复合物结构显示Nsp1以一种类似拍手(clap-like)的方式直接与SL1 RNA结合(图1D-F),其中两个长的反平行β片界面位于SL1的RNA螺旋之上。还观察到Nsp1诱导了RNA的物理弯曲(14.3°),这可能有助于增强相互作用获得更高的亲和力。SL1的顶端/loop环区朝向N端和C端一侧(图1F)。此外,使用PISA服务器计算,Nsp1在SL1 RNA的结合创造了一个大的约943 Å2的掩埋界面(burried interface)。这表明5’UTR的Nsp1和SL1 RNA之间存在真正的紧密相互作用,功能结合研究也证实了这一点(图1A,B)。
另一方面,Nsp1/SL1复合物的表面电荷分布表明,大多数RNA结合在蛋白质的正电荷区域(图1G-I) 。C端区域包含一个带负电荷的表面,其相关性最小,且远离RNA结合槽,观察到的Nsp1的正电荷区域以及其以拍手样方式与凹槽/口袋结合和实验结合研究证明了Nsp1对其翻译调控的病毒RNA识别模式。
随后,作者仔细研究了在Nsp1和SL1之间形成相互作用的关键残基(图2)。从复杂的结构可以明显看出,RNA分子插入到Nsp1蛋白的沟槽状结构中,在参与相互作用的残基中,T12、Y118、R124、K125、N128、K129、L141和D147与SL1 RNA形成氢键, 而Nsp1的其他几个残基与RNA的距离很近,产生强烈的离子相互作用。特别的是,带正电的长链Arg和Lys氨基酸残基(K11, R124、K125和K141)插入到RNA沟槽(图2),与RNA的负电磷酸骨架建立起强大的离子相互作用,因此导致如图1A,C中所见的更高亲和力。此前关于SARS-CoV-1的研究也通过实验表明R124和K125在病毒RNA识别中起着关键作用,这与作者在复合物结构中观察到的及其他参与相互作用的潜在残基一致。随后作者通过突变实验对结合自由能的影响从侧面验证了关键残基。动力学实验表明,野生型Nsp1结合于SL1 RNA的自由能为-68 kcal,大多数突变体的与RNA的结合亲和力明显降低。其中,Y118、R124、K125和K141上的突变表现出2 ~ 3倍的亲和力减少,表明与突变蛋白的物理相互作用减弱或中断。这进一步证实了Nsp1与RNA的真正结合亲和力和有效性,并强调了在Nsp1/SL1-RNA复合物中参与这种物理相互作用的关键氨基酸残基。
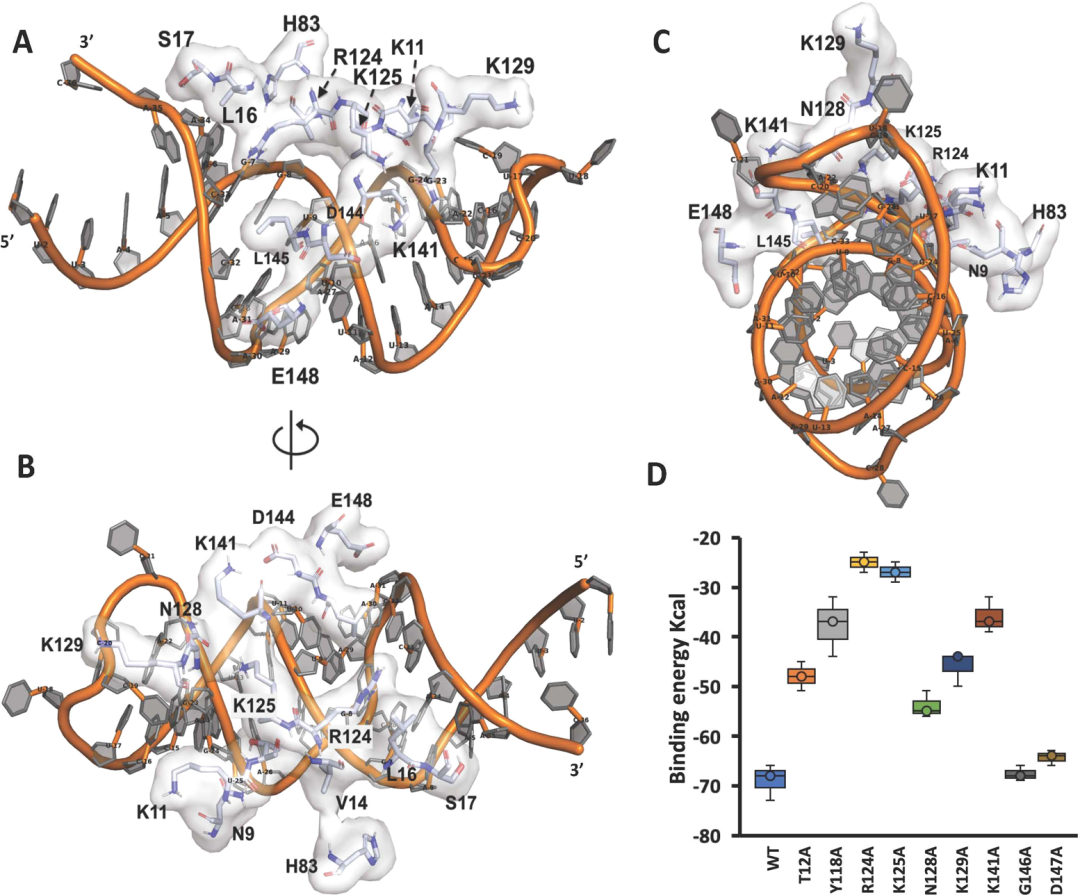
图2. Nsp1/SL1复合物之间详细的氨基酸相互作用及Nsp1单一氨基酸突变对结合自由能的影响。图片来源于:JPCL
为了验证Nsp1-SL1 RNA复合物的整体复合物结构和实时行为,作者使用LARMD进行10 ns的分子动力学模拟。利用DynOmics服务器进行了B因子要素以及结构域分离分析和结合模拟研究,并使用Schrodinger分子动力学同步验证。结果表明Nsp1 – SL1 RNA复合物在相互作用和动态运动方面是稳定的(图3)。最重要的是,Nsp1-SL1 RNA复合物的残基间接触图谱显示,即使在动态或溶液状态下,分子之间也存在清晰而稳健的物理相互作用(图3A)。在动态状态下,复合物之间至少有七个特定区域是稳定的,其中复合物被允许振荡到7.3 Å。复合物中所有残基的B因子值均低于0.5 Å2(图3C),进一步证明了配合物的物理稳定性较好。Nsp1-CTD的B因子分数的增加与大结构域的移动直接相关,这可以促进Nsp1-CTD插入40S核糖体RNA结合通道。Nsp1-CTD在该区域的结合对宿主的翻译调控和Nsp1介导的免疫逃逸起着至关重要的作用。作者随后试图通过域分离动力学来检验Nsp1和SL1-RNA的域分离可能性。如图3D,整个复合物构成的区域和Nsp1的N段区域观察到十分小的特征向量,特征向量的得分分别为0±0.01和0±0.027,这表明复合物具有高稳定性和最小的物理分离可能性。
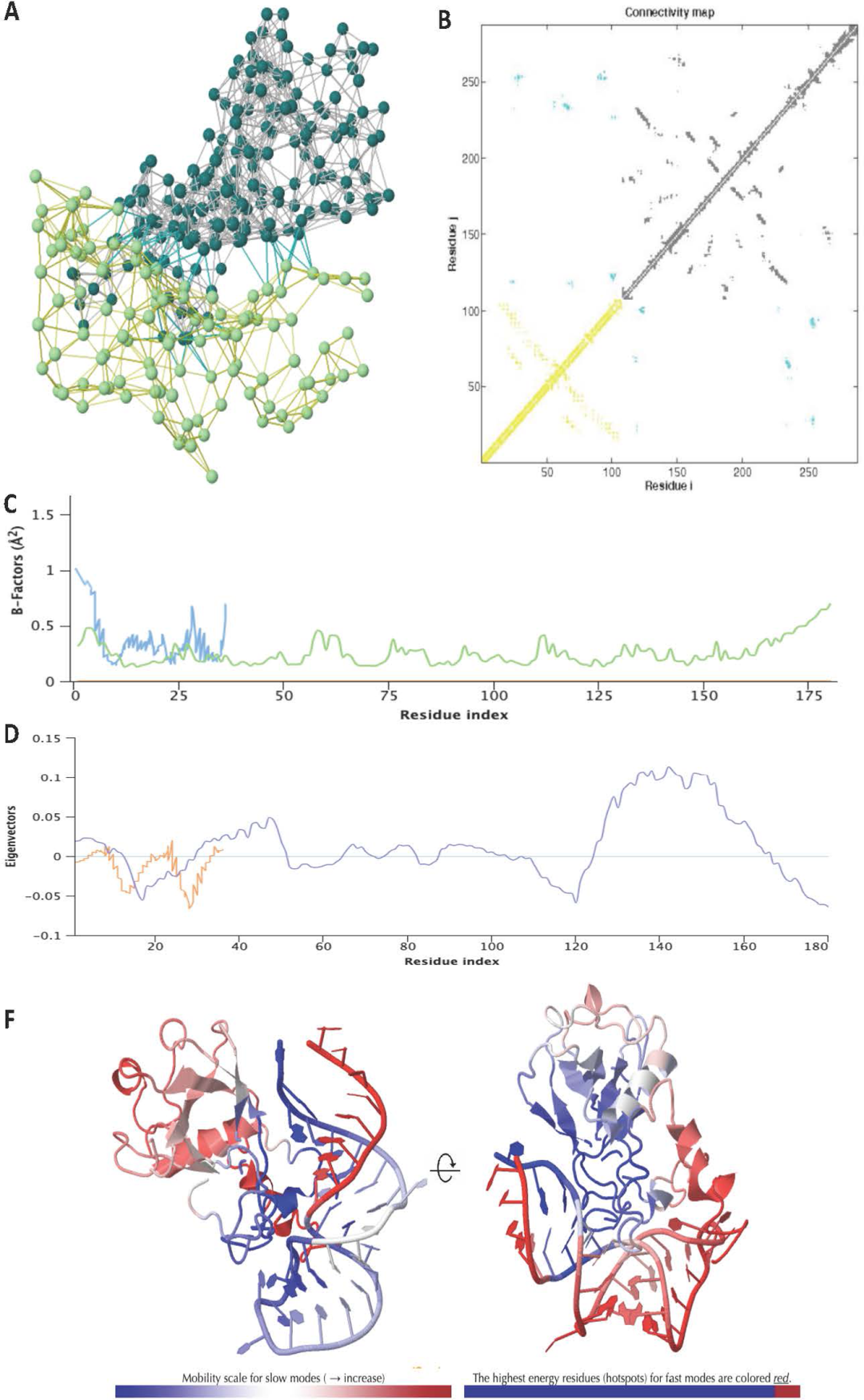
图3. Nsp1/SL1复合物的结构稳定性研究。图片来源于:JPCL
抑制或阻断Nsp1的功能对调节病毒复制的治疗有多个意义。但目前通过计算研究的药物特异性极低,缺乏利用SARS-CoV-2的Nsp1-FL阻断RNA相互作用功能的潜在和特异性药物的结构和功能分析。根据抑制剂的化学性质,Nsp1处表面电荷分布及观察到的RNA和Nsp1的结合模式,作者首次通过对接Nsp1-FL筛选了具有高结合潜力的抑制剂(图4)。甘草酸、 肺衣酸、藤黄酸和替拉扎德能以更高的亲和力与Nsp1结合,并在结构上阻碍Nsp1和SL1之间的物理相互作用。潜在Nsp1抑制剂间相互作用是由几个极性、疏水和盐桥相互作用介导的。候选药物和靶标RNA具有相同的结合区域,且以高亲和力结合。经计算,这些药物与Nsp1-FL的结合△G值在-10.4 ~ -8.6 kcal/mol范围内。这表明,这些药物可能通过抑制Nsp1与SARS-CoV-2基因组5’UTR的SL1的相互作用,或抑制与任何其他目标宿主或病毒RNA的相互作用而发挥作用。
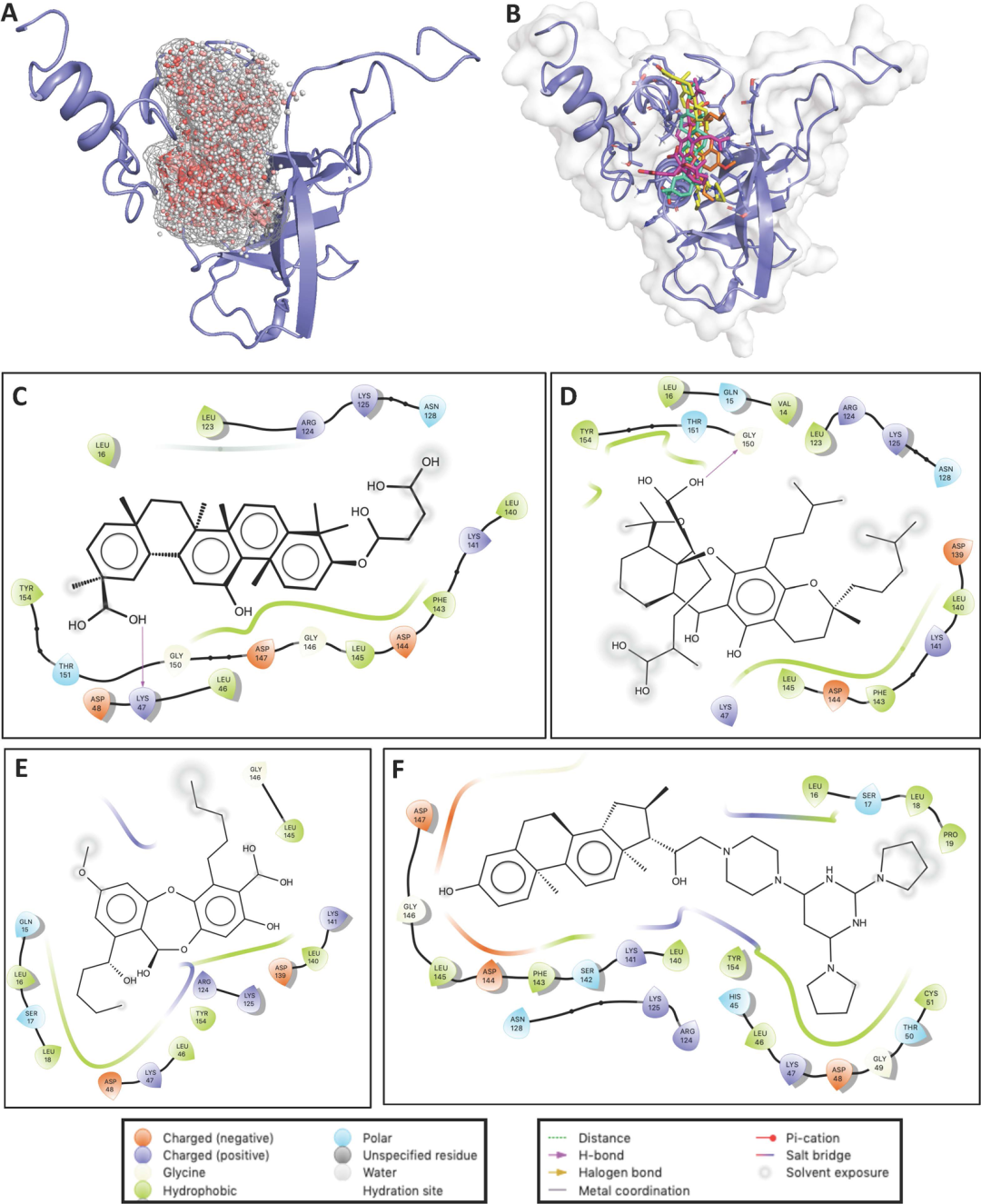
图4.可能阻止Nsp1/SL1复合物形成的潜在药物。图片来源于:JPCL
在疫苗开发方面,已有研究表明,小鼠肝炎病毒的Nsp1上的部分突变能产生减毒病毒,此外,SARS-CoV-2 Nsp1的精准结构和机制及其与病毒RNA和核糖体的相互作用将为探索开发减毒活疫苗打开思路。可能与针对脊髓灰质炎病毒的Sabin疫苗的开发相似,在5’UTR区域的突变导致病毒翻译的失效,从而引导目前脊髓灰质炎疫苗的开发。由于Nsp1也物理且直接的结合于其病毒RNA的5’UTR区域,其5’UTR区域或SARS的Nsp1可能的突变也具有使病毒弱化的能力,这可能有助于疫苗的开发。这一策略可用于开发SARS-CoV-2疫苗。作者设计了Nsp1/SL1复合物在翻译调控中的潜在作用的示意图以供疫苗开发人员参考。
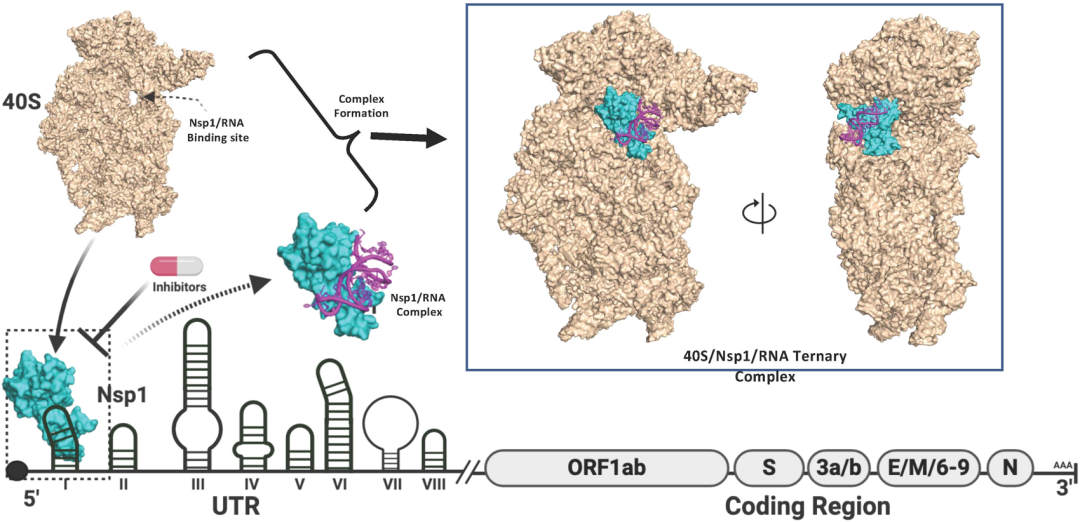
图5. Nsp1/SL1复合体潜在作用的示意图。图片来源于:JPCL
参考文献
Vankadari N, Jeyasankar NN, Lopes WJ. Structure of the SARS-CoV-2 Nsp1/5′-Untranslated Region Complex and Implications for Potential Therapeutic Targets, a Vaccine, and Virulence. J Phys Chem Lett. 2020 Nov 19;11(22):9659-9668. doi: 10.1021/acs.jpclett.0c02818. Epub 2020 Nov 2.