

iANP-EC是一种集成计算框架,结合机器学习和进化计算可识别天然抗癌化合物。
背景介绍
在癌症治疗中,尽管化疗和放疗有严重的副作用,但它们仍是最普遍应用的方法。发现具有更高疗效、更好适应性和更持久疗效的新型抗癌药物至关重要。计算模型与实验平台相结合可以有效提高探索新型潜在抗癌化合物的成功率,确定更多的潜在候选药进入试验阶段。除了合成化合物外,人们还对天然产物进行了严格的研究,以提高抗癌治疗的有效性和质量。
天然产物是由各种生物来源产生的生物合成代谢物,包括植物、细菌和真菌等。科学证据表明,它们是开发治疗各种疾病(包括癌症)的新药的关键化合物来源。天然分子支架的多样性和独特性,有望启发科学家设计新的药物分子。此外,人们还发现,天然的抗癌物质可以触发癌细胞的凋亡诱导活性。此外,人们已发现许多天然产物具有广泛的抑制活性,通过多靶点机制杀死癌细胞。
除了实验方法外,最近人们还发展了多种计算方法来确定抗癌药物。然而,当前计算方法主要集中在抗癌肽上。尽管这些方法取得了阶段性成果,但确定具有抗癌活性的天然产物的模型仍有很大的改进空间。
数据下载链接
https://github.com/mldlproject/2021-iANP-EC
主要内容
越南胡志明国立大学的Ly Le 和新西兰惠灵顿维多利亚大学的Binh P. Nguyen团队提出了一个更有效的计算框架:iANP-EC,该框架使用集成学习结合进化计算来识别抗癌天然产物。为了构建集成模型的基分类器,研究者使用k-最近邻(k-NN)、支持向量机(SVM)、随机森林(RF)和极端梯度增强(XGB)算法,并结合RDkit分子描述符集(RDkit MD)和Mol2Vec编码两种扩展连接指纹(ECFPs),获得了16个分类器。接着利用粒子群优化算法(PSO),对四个顶级分类器的组合权重进行优化。结果表明,iANP-EC是一个稳定、稳健、有效的框架,其AUC-ROC值为0.9193,AUC-PR值为0.8366。通过对天然抗癌药和非抗癌药分子亚结构的比较分析,揭示了驱动抗癌活性的几个关键亚结构。相关的研究成果以“iANP-EC:Identifying Anticancer Natural Products Using Ensemble Learning Incorporated with Evolutionary Computation”为题发布在国际著名期刊Journal of Chemical Information and Modeling上。
iANP-EC概述
图1概括了iANP-EC研究中的主要步骤。首先,研究者将经过优化的数据集随机分成训练集、验证集和独立测试集,然后使用不同的分子表示方案将其转换为相应的数字形式。对特征训练数据的5倍交叉验证结果被用来为每个分类器找到最佳的超参数。每个分类器由一种分类算法和一种分子表示组合而成。研究者使用了四种分类算法,包括k-NN、SVM、RF和XGB,并结合了四种分子表示方案,因此,得到了16个分类器。选取前4个分类器作为基分类器,构建集成模型。
利用整个训练集上的最佳超参数,对前四个分类器进行再训练。研究者提出的模型是使用加权集成学习方法建立的,其中每个样本的预测概率,由单个基分类器返回,分配权重。样本对集合模型的预测概率为Pensemble = w1P1 + w2P2 + w3P3 + w4P4。利用粒子群优化算法(PSO)对验证集的权重w1、w2、w3和w4进行优化。最后,利用特征化后的测试数据对模型性能进行评价。
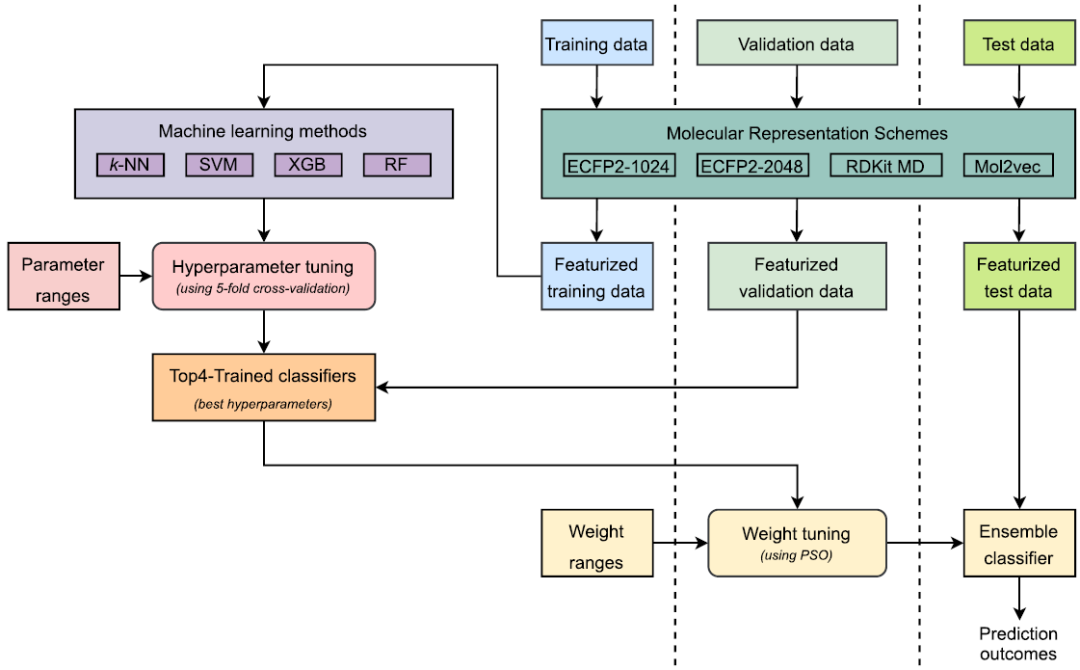
图1. 开发iANP-EC主要步骤的图表描述。图片来源:JCIM
数据管理
数据管理过程遵循Fourches等人的流程,并进行了少量修改,包括四个主要阶段:(1)验证,(2)清理,(3)规范化,(4)最终检查。验证阶段除去了混合物、无机和有机金属。清洗阶段除去了盐和带电化合物。正规化阶段包括去自动化和立体异构化。在非自动化子阶段,化合物被非自动化变为正则非自动化SMILESs的形式。检查阶段识别出有冲突标签的样品,或简称为冲突样品。最后,研究者完成了数据整理阶段,得到了997个独特样本的细化数据集,其中包括367个抗癌和630个非抗癌。策划的数据被分成三个分层集:一个训练集,一个验证集和一个独立的测试集。训练集用于5倍交叉验证和模型训练。权值调优使用验证集,独立测试集评估模型性能(表1)。
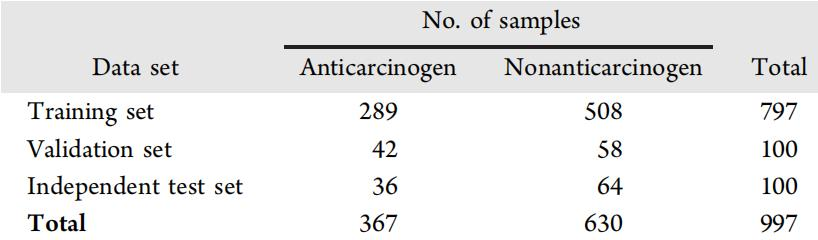
表1. 用于预测模型开发和评估的数据。表格来源:JCIM
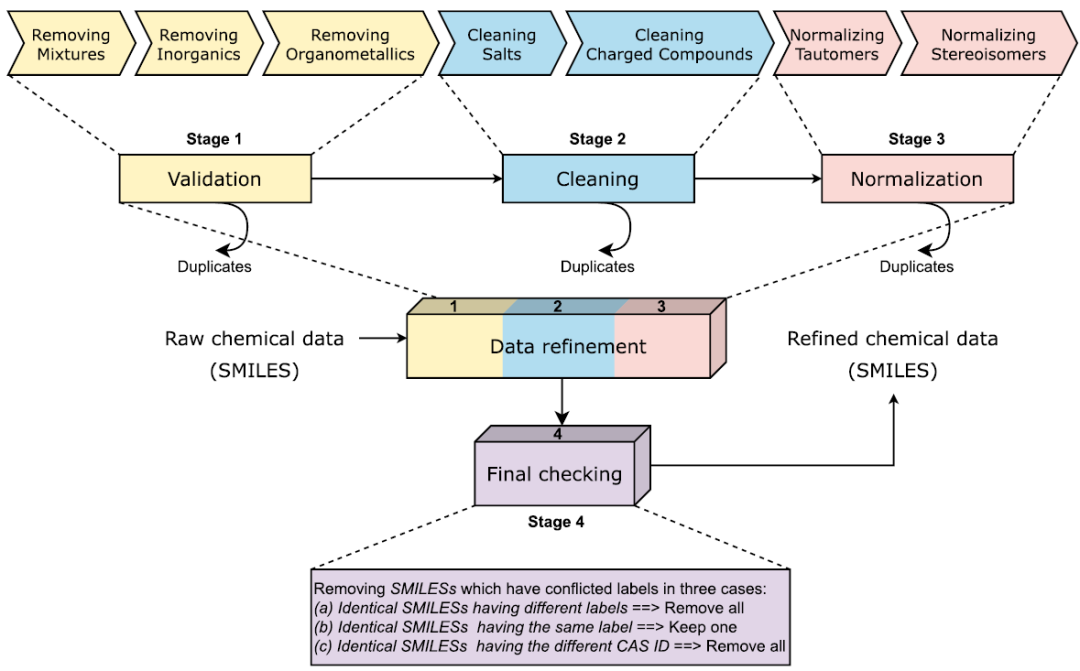
图2. 数据管理中的处理步骤。图片来源:JCIM
模型构造
图3显示了加权集成模型是如何构建的。研究者将所提出的方法与其他两种集成方法(平均法和多数投票法)而不是最好的单个模型进行比较,以显示iANP-EC的优势,因为集成模型通常比单个模型表现出更好的性能。
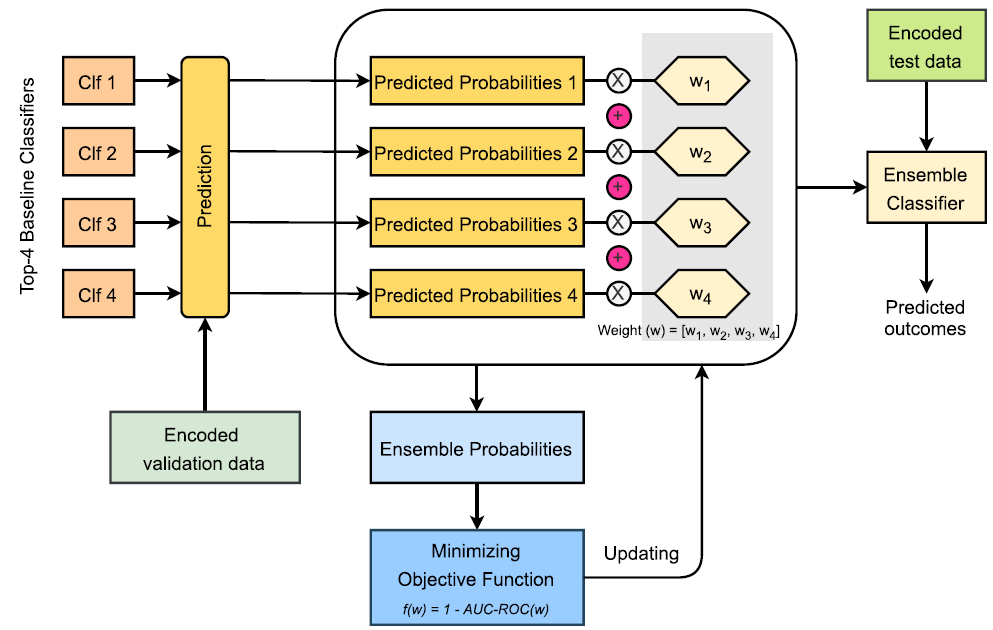
图3. 用粒子群优化方法构建集成模型。图片来源:JCIM
化学多样性分析
本研究中,研究者采用了主成分分析对RDKit MD特征化的数据进行分析和可视化。图4显示了数据集的化学多样性和适用性领域。由于训练集、验证集和测试集是使用基于结构的分层抽样方法创建的,它们的覆盖空间是相似的(图4a)。此外,还应用PCA观察了抗癌和非抗癌物质所覆盖的化学空间(图4b)。结果发现大多数抗癌物质相互靠近,在主簇旁边形成许多小簇。
此外,为了阐明本研究的适用范围,研究者还对本研究使用的所有数据和更大的一组天然产物进行了PCA。研究者选择NPASS数据库作为天然产物的代表性来源,有超过35 000个代谢物条目;同时,研究者收集并整理了超过28000种天然产物(不包括在当前数据集中)。PCA图显示,研究者的数据集覆盖了天然产物空间的一个大区域。
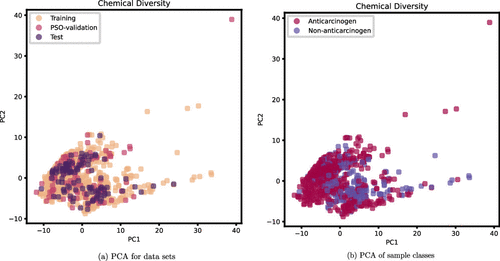
图4. 主成分分析的化学多样性及其应用领域。图片来源:JCIM
子结构的重要性分析
为了进一步了解有助于模型鉴别能力的关键分子亚结构的重要性,研究者使用SHAP (SHapley Additive explained)方法,对两组抗癌药和非抗癌药的子结构进行了比较分析。图5显示了哪些子结构向预测模型添加了更多的值。该图列出了有助于从基础值提升模型性能的关键特性(子结构)。促进预测任务的子结构用一个红色右尖蜜蜂图来表示,而降低预测任务的子结构则表示为一个蓝色左尖蜜蜂图。分析结果表明,bit-36子结构对提高模型性能的贡献最大,且变化较小;bit-484、bit-420、bit-960、bit-35、bit-202、bit-503子结构对提高模型性能的贡献最大,且变化较大;而其他子结构可以在一定程度上降低或提高模型性能。
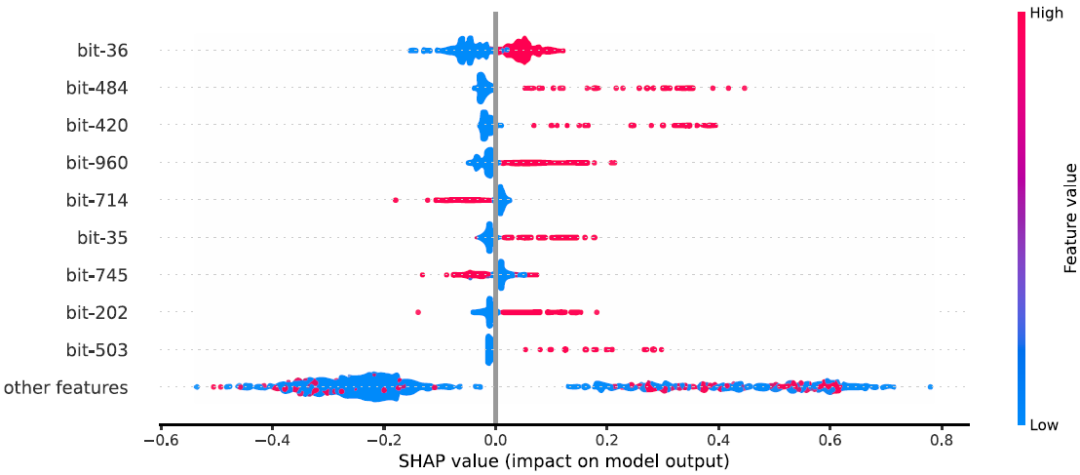
图5. 在抗癌药和非抗癌药分类中具有重要意义的关键亚结构。图片来源:JCIM
同时,研究者还对两组天然产物的子结构分布进行了比较分析。条形图显示了抗癌药和非抗癌药分子亚结构分布的差异(图6)。对其分子亚结构的分析表明,其亚结构有一些相似和不同之处,这可以部分解释其抗癌活性的性质。x轴是子结构标识符(位),而y轴是整个复合集的截止水平。化合物的子结构标识符为Morgan指纹位(使用1024位集)。根据SHAP分析结果选择子结构(图5)。bit-36亚结构存在于60%以上的抗癌药物中,而这种亚结构仅存在于40%的非抗癌药物中。两组子结构中bit-484、-420、-714、-35、-745、-503的分布较为明显。具有bit-484、-420、-35、-745和-503亚结构的抗癌药部分均超过总集合的10%,而对于非抗癌药,这些部分几乎可以忽略不计。相反,大约10%的非抗癌药具有bit-714亚结构,而具有bit-714亚结构的抗癌药所占比例非常小。此外,bit-960和-202子结构在两组中分布相当均匀。
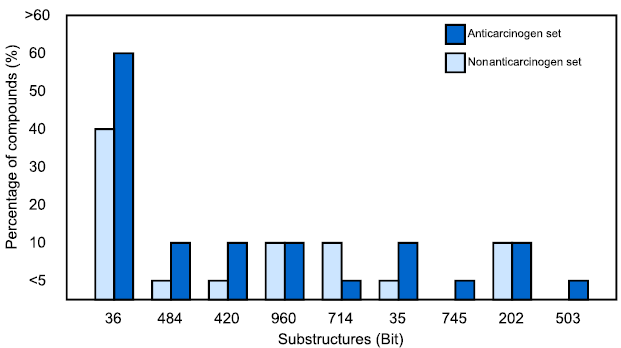
图6. 抗癌和非抗癌基团中亚结构的分布。图片来源:JCIM
总结
本文介绍的iANP-EC方法是一种有效的抗癌天然产物预测计算框架。与单独使用机器学习相比,将机器学习方法和进化计算相结合可以获得更高的模型性能。该集成模型从16个初始分类器中选出4个性能最佳的分类器,有效地提取了基本分子特征,提高了分类模型的预测能力。
参考文献
Loc Nguyen, Thanh-Hoang Nguyen Vo, Quang H. Trinh, Bach Hoai Nguyen, Phuong-Uyen Nguyen-Hoang, Ly Le, and Binh P. Nguyen, iANP-EC:Identifying Anticancer Natural Products Using Ensemble Learning Incorporated with Evolutionary Computation, Journal of Chemical Information and Modeling Article ASAP DOI: 10.1021/acs.jcim.1c00920