

对Specs库进行虚拟筛选得到的两个靶向GyrB的抗结核杆菌抑制剂具有IC50 值分别为2.69 和 2.46 μM的生物学活性,并通过分子动力学模拟分析药效团明确了抑制作用,为优化抗结核药物的开发提供了起始模板。
背景
结核病(TB)是一种由结核分枝杆菌(M. tuberculosis)引起的具有全球影响的传染病,它主要攻击肺部(肺结核),但也会影响其他部位(肺外结核病),主要传播途径为呼吸道传播和消化道传播。据估计,全球约有四分之一的人口感染了结核分枝杆菌。结核病的治疗受到耐药菌株传播的阻碍,包括耐利福平结核病和多药耐药结核病,因此急需新的抗结合药物来解决耐药性。2020年,两种新的化合物(TBAJ-876和GSK-286)已进入结核病临床开发管道的I期试验,结核病临床开发的化合物总数达到14个,其中8种作用于5个新的结核病靶点。
DNA拓扑异构酶II在许多在原核生物和真核生物中管理DNA(重)组织的细胞事件中发挥着重要作用。在大多数细菌中有两种II型拓扑异构酶,DNA促旋酶( Gyrase )和DNA拓扑异构酶IV(Topo IV)。但结核分枝杆菌只编码DNA促旋酶,并通过使用ATP作为辅助因子将负超螺旋引入DNA来控制DNA拓扑结构。DNA促旋酶由两个亚基组成,DNA促旋酶A(GyrA)和DNA促旋酶B(GyrB),它们结合形成一个具有催化活性的异源四聚体复合物(GyrA2GyrB2)。近期,结核分枝杆菌DNA促旋酶的四聚体复合物结构被解析,为基于此酶结构的小分子药物设计提供了结构信息。
DNA促旋酶已被验证为氟喹诺酮类药物(FQs)的酶靶点,FQs是用于治疗多药耐药结核病的二线抗生素。DNA促旋酶DNA结合位点周围的突变导致结核分枝杆菌出现FQ耐药性;抑制DNA促旋酶ATP酶活性是克服这一问题的一种策略。本文利用虚拟筛选结合湿实验的方法,从化合物库(www.specs.net)中筛选出了结核分枝杆菌DNA促旋酶ATP酶活性的候选抑制剂。随后使用分子动力学模拟分析了其药效团,为向DNA促旋酶发挥作用的抗结核药物设计奠定了基础。
虚拟筛选
虚拟筛选的流程如图1 所示。得分最高的30个化合物与GyrB ATP结合口袋结合的Glide XP得分在-3.2~-7.2 kcal/mol范围内。作者先将它们作为命中(Hit)化合物收集。其中,10个化合物(G01、G07、G08、G10、G13、G19、G24−G27)是同时具有S和R构型的手性分子。对这些分子也计算了S和R构型分子的Glide XP分数。
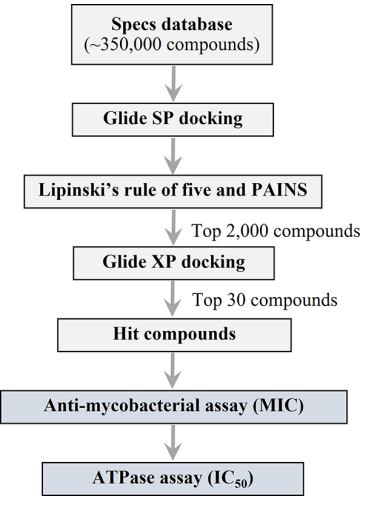
图1. 发现有效DNA促旋酶抑制剂的虚拟筛选工作流程。图片来源:JCIM
抗结核活性
以DNA促旋酶抑制剂新生霉素为参考化合物,作者对30种命中化合物对无致病性的H37Ra结核分枝杆菌的最低抑制浓度(MIC)进行了初步评估,以降低实验成本和生物安全的严格要求。不幸的是,所有命中化合物都在7H9GC培养基中沉淀,推测溶解度可能受到其高亲脂性的限制。Swiss ADME计算的单个命中化合物的MlogP值在1.19−4.15范围内,高于可溶于7H9GC培养基中的新生霉素(0.65)。检测的化合物中,G24和G26(图2)的MIC值均为12.5μg/mL,表明它们对结核分枝杆菌的活性明显高于新生霉素 (MIC>100μg/mL)。据此,作者测定了二者对H37Rv结核分枝杆菌生长抑制作用的MIC值。结果表明,G24和G26对H37Ra、H37Rv结核分枝杆菌表现出相同的MIC值(12.5μg/mL)。因为30个命中的化合物是基于Glide XP评分选择的,所以作者随后考虑了Glide XP评分和MIC值之间的相关性。结果发现,数据并没有确定Glide XP得分和MIC值之间的相关性。作者推测这可能是由于大多数Hit化合物在MIC检测中的低溶解度妨碍了对其抗细菌活性的准确评估,以及/或Hit化合物对分枝杆菌细胞的渗透能力较差等原因。
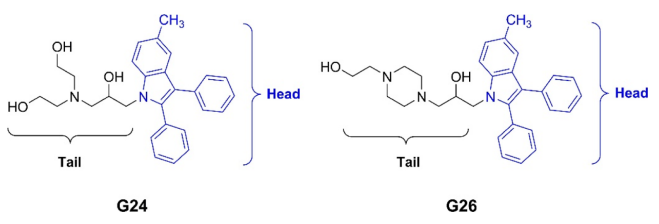
图2. 化合物G24和G26的化学结构。图片来源:JCIM
抑制结核分枝杆菌DNA促旋酶ATP酶活性
由于化合物G24和G26对结核分枝杆菌H37Rv和H37Ra菌株的生长有抑制作用,并且在该试验中比新生霉素更有效。作者随后以新生霉素作为参照,测定化合物抑制结核分枝杆菌DNA促旋酶ATP酶活性的IC50。结果显示,G24和G26对ATP酶抑制的IC50分别为2.69和2.46μM(图3),与新生霉素(IC50 1.27μM)表现出相当的ATP酶抑制活性。由于这两种化合物在细胞实验和酶抑制试验中都具有活性,作者推测它们的抗结核活性可能是由于抑制了DNA促旋酶的ATP酶。因此,G24和G26被认为是可进一步开发新型抗结核药物的很有前景的先导化合物。
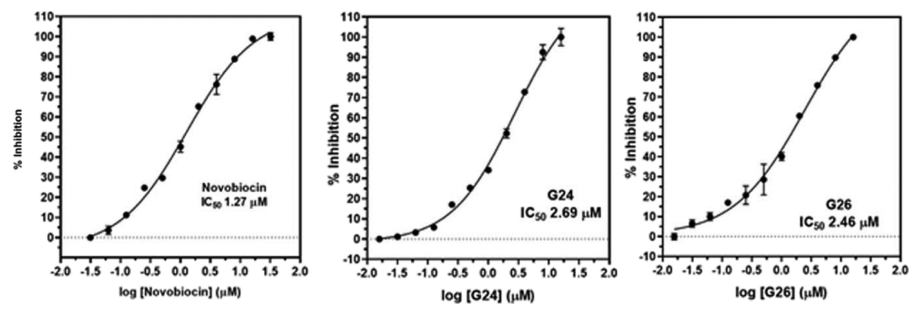
图3. 抑制结核分枝杆菌DNA促旋酶ATP酶活性。图片来源:JCIM
抑制剂结合在ATP结合位点
随后,作者对G24和G26与结核分枝杆菌DNA促旋酶GyrB亚基中ATP结合位点的结合模式进行了分析。这些化合物的化学结构包括等效的5-甲基-2、3-二苯基-1H-吲哚疏水头部,具有不同的羟乙基/氨基(G24)和羟乙基/哌嗪基(G26)极性尾部(图2)。由于由Specs数据库提供的两种化合物G24和G26的精确立体化学构型尚未被确定,所检测的化合物可能是外消旋体,因此作者进一步考虑了它们在R和S构型中的结合模式。G24在R和S构型的预测结合模式不同,分子对接计算得到的不同的Glide XP评分(−7.5和−5.7kcal/mol)。而预测结果表明,R构型中的化合物G24可能更有利地结合GyrB ATP结合位点。化合物G26的R和S构型的预测结合模式则相似,Glide XP评分相当(−6.9和−7.4kcal/mol)。作者在比较化合物G24的R构型和G26的S构型这些更有利的结合模式时,发现两者预测的结合模型更为相似, 疏水头部基团很好地重叠。此外Glide XP评分也具有可比性。这些数据表明,G24和G26对GyrB ATP结合位点的结合亲和力没有显著差异,与IC50测试结果一致。模拟的结构也表明,二者很可能都结合在一个与ATP腺苷基团所利用的结合位点部分重叠的结合位点上。
随后利用MD进一步分析了化合物G24(R构型)和G26(S构型)与GyrB ATP结合位点的结合。化合物在 GyrB ATP结合位点的结合模式由分子对接得到,在经过了三个重复MD模拟后,其RMSD值表示与初始对接结构相比,在平衡态上略有不同(G24和G26分别为∼1.6−2.5 Å和∼1.4−1.7Å)。在配合物的MD模拟中观察到涉及化合物极性尾部的氢键(图4),在分子对接结果中没有体现。G24的极性尾部与Glu56形成氢键,这在三次重复的MD模拟中都可以观察到,而G26的极性尾部则与Asn52和Arg82形成氢键。在三次重复MD模拟中,两种化合物与GyrB ATP结合位点的平均结合自由能(ΔGPBSA和ΔGGBSA)是相似的。这些结果表明, G24和G26对结核分枝杆菌DNA促旋酶GyrB具有相似的结合亲和力,与二者IC50值相似的结果一致。
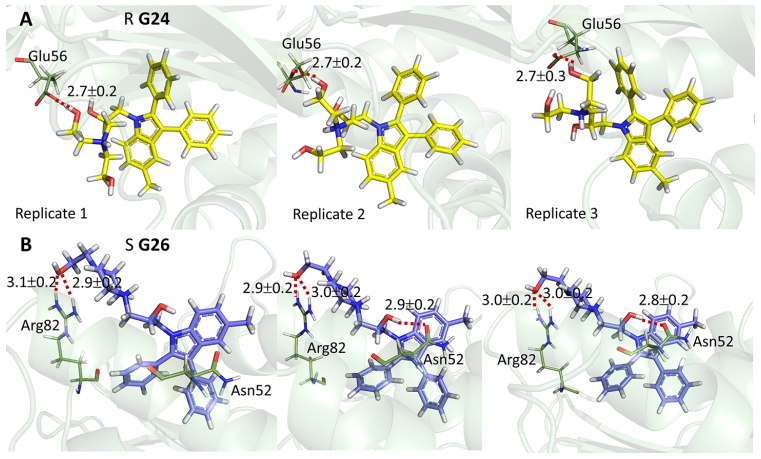
图4. 化合物G24和G26在三次重复MD与GyrB ATP结合位点的结合模式。图片来源:JCIM
药效团映射分析
用LigandScout 4.4生成的2D药效团模型来分别表示结核分枝杆菌DNA促旋酶与G24和G26之间的主要相互作用(图5)。生成的两个模型识别了三种不同的药效团特征:(i)化合物头部基团的疏水特点,使其结合于由Val49、Ala53、Ile84、Val99、Met100、Val125和Val171形成的疏水口袋;(ii)极性尾部带有正电的部分指向Glu56;(iii)G24和G26的氢键供体分别与Glu56和Asn52相互作用,G26的氢键受体与Arg82相互作用。分析表明,这两种化合物的疏水头部和极性尾部可能对它们抑制结核分枝杆菌DNA促旋酶的ATP酶活性有重要作用。其中,G24和G26的极性尾部,可能通过增强与GyrB ATP酶位点的相互作用和提高溶解度来提高抗结核活性。
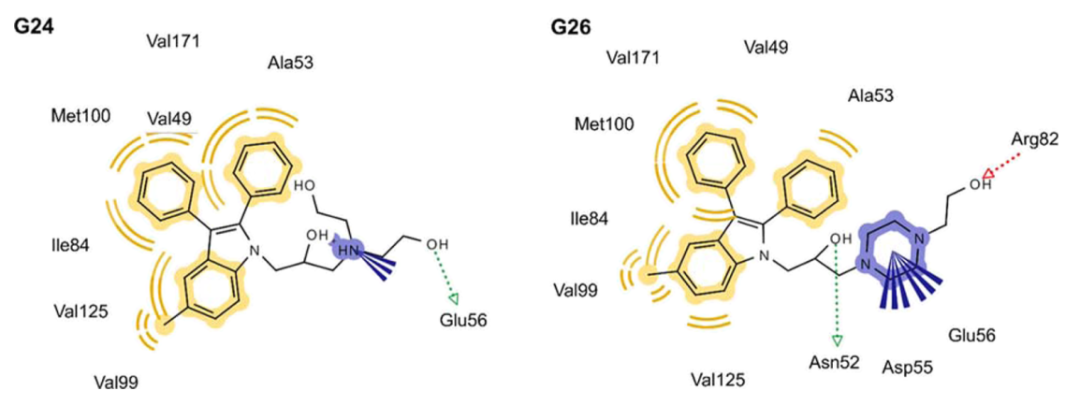
图5. G24和G26的药效团模型。图片来源:JCIM
结论
本文介绍了虚拟筛选如何结合生物活性实验识别新的具有抗结核剂潜力的结核分枝杆菌DNA促旋酶ATP酶抑制剂。筛选得到的化合物G24和G26对结核分枝杆菌菌株表现出比新生霉素更强的抗结核活性,IC50分别为2.69和2.46μM。通过MD模拟发现,这些化合物可以结合到GyrB亚基的ATP结合位点,而药效团定位分析也进一步表明了疏水、静电和氢键相互作用对结合的重要性。研究为后续抗结核药物的研发奠定了基础。
参考文献
Pakamwong B, Thongdee P, Kamsri B, Phusi N, Kamsri P, Punkvang A, Ketrat S, Saparpakorn P, Hannongbua S, Ariyachaokun K, Suttisintong K, Sureram S, Kittakoop P, Hongmanee P, Santanirand P, Spencer J, Mulholland AJ, Pungpo P. Identification of Potent DNA Gyrase Inhibitors Active against Mycobacterium tuberculosis. J Chem Inf Model. 2022 Apr 11;62(7):1680-1690. doi: 10.1021/acs.jcim.1c01390. Epub 2022 Mar 29. PMID: 35347987.