

光谱质量(SPEQ)评估工具可用于鉴别并消除低质量光谱,同时突出显示高质量的光谱。本文用深度神经网络建立的光谱质量判定模型SPEQ与其他预测工具相比,展现出优秀的预测准确性。
背景介绍
串联质谱(MS/MS)将高效液相色谱和MS相结合,可从蛋白质混合物中鉴定出肽链,是蛋白质组学中鉴定蛋白质的主要手段。首先用酶将蛋白质消化成更小的肽,然后对肽进行LC-MS/MS分析。实验中会产生许多肽的谱图,通常借助数据库搜索引擎鉴定各个肽的结构。但是由于光谱质量、软件参数等因素,半数以上的谱图不能被数据库搜索引擎正确识别,常被误判成假阳性或假阴性。这些光谱中可能含有的高质量的光谱需要使用SPEQ识别出来并交由研究人员进一步分析。
质量评估工具有助于科研人员安全地丢弃低质量光谱,提高数据分析速度。当前工具主要是人工提取特征加传统的机器学习的方式,比如用一组人工设计的特征和支持向量机建立质量评估模型。后续也出现了决策树、随机森林等模型。如今这些工具有的不再维护,有的不适用于当前质谱仪的数据。
图 1 介绍了如何对不同质量的光谱进行分类。如果将显著峰定义为峰相对强度约为 5% 或更高的峰,则高质量光谱中显著峰的数量比低质量更多更密集。高质量光谱中显著峰之间的 m/z 差异蕴含了氨基酸残基质量的信息。这些差异是构建光谱鉴定程序的基础。
本文介绍的SPEQ工具使用深度神经网络 (DNN) 模型来预测光谱质量。模型从训练数据中学习特征,所提取的特征对于每种类型的数据集都不同。
图1. 第一行三个样本为被MS-GF+正确识别的高质量样本,第二行为未被识别的低质量样本。图片来源:Bioinformatics
主要内容
SPEQ的建模
训练数据来自用不同高分辨率仪器生成的四个数据集:Quadrupole time-of-flight、Orbitrap human、NIST和Orbitrap mouse数据集。
SPEQ 用MGF格式的频谱作为输入,每个光谱的峰列表用元组形式表示。首先将光谱中最丰富的峰强度变换到0 -100 区间,然后对数据分段,各段的最大的峰强度代表该段的数值,如果区间中无峰值则取0,最后将光谱表示成一个向量。
DNN以向量作为输入来预测光谱质量,模型架构如图 2 所示,核心部分由一维卷积层组成,最后连接全连接层。模型以处理后的向量作为输入,然后历经三个卷积块。每个卷积块都有一个dropout层、卷积层和池化层。它们可以从光谱的向量中提取出特征,此外还有电荷状态和荷质比,它们与第三个卷积块的输出合并,共同输入到三层全连接神经网络。最后一层经过sigmoid函数变换,输出预测概率P,代表的是高质量光谱的概率。
作者基于伯尔尼的描述重现了Bern模型。其中SVR模型的c经过了参数调优给出了最佳的数值,模型表现优于文献中的数值。
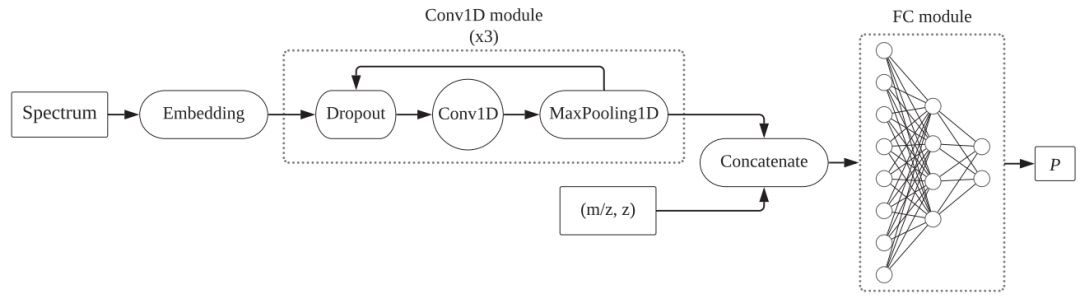
图2. 深度学习模型的架构图。图片来源:Bioinformatics
SPEQ模型的评价方式
模型的表现从三个方面进行评价。
首先比较SPEQ与其他模型在预测精度上的差异,用5折交叉验证来衡量预测精度,划分过程确保具有相同m/z和z的光谱在同一部分。此外,还进行了跨物种验证,模型在 Orbitrap human 数据集上训练,用独立的 Orbitrap mouse数据集测试。
其次,测试未识别出的光谱能否用其他分析方法解释。在这个测试中,作者首先使用 MS-GF+ 搜索 Orbitrap human 数据集,用从 MS-GF+ 搜索生成的标签训练了 SPEQ 模型,为所有光谱赋予预测的SPEQ 分数。作者使用 Comet 软件以相同的参数进一步搜索未识别的光谱。前两次分析无法识别的光谱使用 Novor 软件从头测序。包含至少五个具有高置信度分数(> 70)的氨基酸的从头序列被视为置信的从头标签。预计第一次搜索未识别的光谱中较高的 SPEQ 分数与较高百分比的由 Comet 或从头测序分配的光谱相关。
第三个测试证明SPEQ分数在“排除”数据分析中的有用性。首先使用 MS-GF+ 搜索 Orbitrap human数据集。研究人员猜测许多未识别的光谱是因为它们搜索参数中未指定 PTM。但是,在整个数据集上使用太多可变 PTM 进行搜索的速度非常慢。作者使用 SPEQ 对未识别的光谱评分,根据 SPEQ 分数排序,将未识别光谱的前 1% 用于其他变量 PTM 搜索。从鉴定的肽中,作者选择了一些最常见的 PTM进行第三轮搜索,使用所有未识别的光谱和这几个额外选择的可变 PTM 来识别更多的肽。
SPEQ预测具有高精度
图3中显示了每个模型在 Q-TOF、Orbitrap human和 NIST 数据集上做出的预测的 ROC 曲线,SPEQ 的预测准确性优于所有其他工具。NIST 数据集中,所有模型的表现都很差。这可能是因为 NIST 数据集中缺少极低质量的光谱,而且区分高质量和中等质量的光谱比区分高质量和低质量的光谱更难。
在 Orbitrap mouse数据集上不同方法的 ROC 曲线及其 AUC如图3所示。Spectrum Quality 工具在此处未能做出任何有效预测,因此未包含在图中。SPEQ 和 Bern 的方法在同一数据集上训练并使用 5 折交叉验证,或者在 Orbitrap human数据集上进行训练,然后绘制出ROC曲线。两个模型在同一数据集上的交叉验证测试比在不同数据集上的训练预测效果更好。在不同数据集上训练和测试时,SPEQ 具有良好的预测准确度,而且均优于 Bern 方法和基线方法。
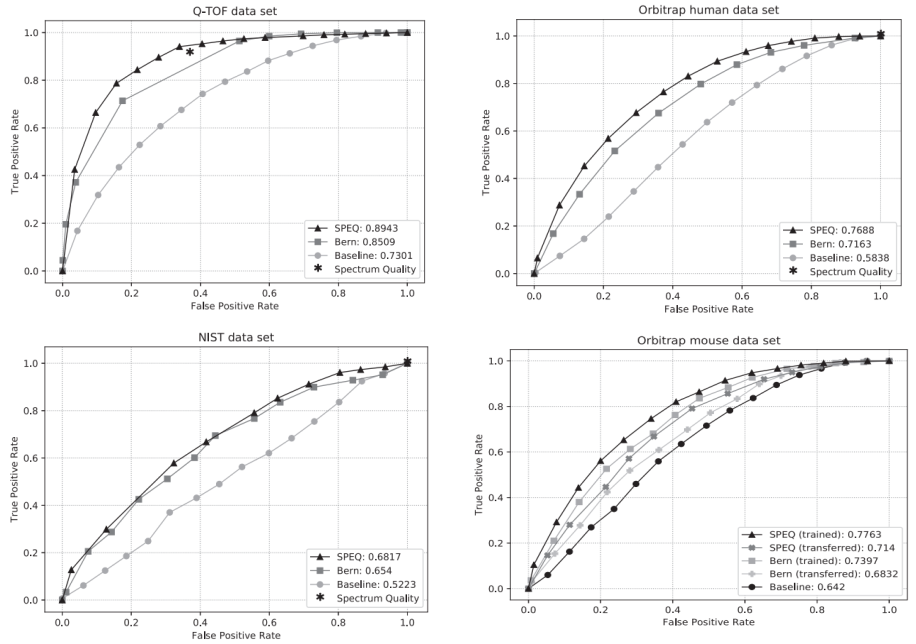
图3. 左上:Q-Tof数据集上的ROC-AUC曲线图;右上:Orbitrap human数据集上的ROC-AUC曲线图;左下:NIST数据集上的ROC-AUC曲线图;右下:Orbitrap mouse数据集上的ROC-AUC曲线图。图片来源:Bioinformatics
多种质量评估工具联用提高识别比例
图 4 显示了未知光谱的测试结果。在Orbitrap human数据集中的 52 285 个光谱,20 885 个光谱不能被正确识别。这些未识别的光谱使用 Comet 进行第二次数据库搜索,并使用 Novor 进行从头测序。图 4A 的直方图显示了每个 SPEQ 评分区间中的光谱数量,以及四类光谱的分布:在第一次搜索中被 MS-GF+ 识别;在第一次搜索中未识别但在第二次搜索中被 Comet 识别;未由前两次搜索识别,但被Novor识别的光谱;没有被任何一种工具识别的光谱。
图 4B 与图 4A 相同,只是 y 轴成为每个 SPEQ 得分区间中的百分比。为了绘制这些数字,取SPEQ 预测的概率的对数被用作 SPEQ 分数。可以看出,随着 SPEQ 提供的质量分数的增加,由至少一个模型正确识别的光谱百分比增加,第一个模型未识别的高分光谱通常可以在其他分析中得到解释。包含有价值信息的光谱通常获得更高的分数。
此外,当分数足够高时,两个数据库搜索工具都没有识别出的大多数光谱都包含从头测序标签。这有力地表明这些光谱确实是由肽产生的,但由于数据分析不充分而无法识别。
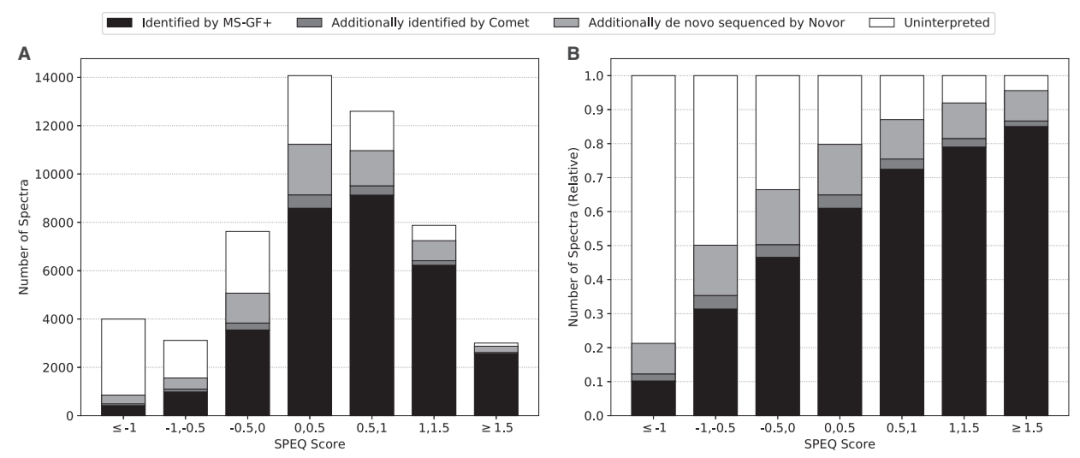
图4. (A)SPEQ不同预测分数区间中识别和未识别的光谱数量统计;(B) SPEQ不同预测分数区间中识别和未识别的光谱比例。图片来源:Bioinformatics
SPEQ与检修错误原因
MS-GF+ 第一次搜索Orbitrap human数据集,使用以下变量 PTM:M 上的氧化,蛋白质 N 端的乙酰基, N 和 Q 上的脱酰胺。
该数据集的原始文献仅使用了上述PTM的前两个变量。添加第三个 PTM 可识别出更多光谱。在 52 285 个光谱中可以识别出 31 400 个。在 20 885 个未识别的光谱中,前 1% 的光谱(根据它们的 SPEQ分数)再次使用新的 PTM 进行搜索,确定了 24 个可靠肽。第三次搜索这三个最常见的 PTM 和 20 885 个未识别的光谱,确定了 829 个光谱。
结果表明,SPEQ 分数确实可以用于选择一小部分 (1%) 的未识别光谱分析错误原因,调整搜索策略识别更多肽。
总结
SPEQ是一种使用深度学习来预测 MS/MS 光谱质量的软件工具。与其他方法相比,SPEQ具有更高的 AUC,即使在跨物种实验中依然表现良好。模型自动提取重要特征且适用于不同类型的质谱仪,这大大节省了开发人员的时间,有助于筛选出高质量的光谱。
参考文献
Soroosh Gholamizoj and Bin Ma. SPEQ: quality assessment of peptide tandem mass spectra with deep learning, Bioinformatics, 38(6), 2022, 1568–1574.