引言
量子力学方法在理论上准确度高,但是面对生物大分子体系,计算代价很大,无法满足实际应用需求,引入经验参数的半经验量子力学(SQM)方法是一种以近似方法,在时间和准确性之间折中,可用于评分以及估算配体与蛋白质的亲和力,在计算机辅助药物设计中具有重要意义。在CADD实际应用中如何选择合适的半经验量子力学方法?本文通过蛋白-配体晶体结构数据构建基准测试集,测试不同半经验量子力学方法的准确度以及溶剂化效应。

背景
在过去的几十年中,信息技术的飞速发展为科学研究开辟了许多新领域。其中之一是计算机辅助药物设计,它利用了现在强大的计算能力来预估基于靶标相互作用的各种药物的结合特性。其中的一种常用方法是基于解Schrodinger方程的量子力学方法。量子力学方法(QM)方法在理论上无疑是精确的,但当药物设计中经常遇到较大的体系(如蛋白质),QM需要更大的计算资源才能在合理的时间内完成计算,因此面对较大体系时无法同时兼顾准确度及计算时间。引入经验参数的半经验量子力学(SQM)方法是一种可以在时间和准确性之间进行折中,以换取近似方法。例如SQM方法可用于评分,以估算配体对蛋白质的亲和力。但是,此类方法的参数测试通常基于传统化学模型的数据集,尽管该模型系统包含基础及不同类型的测试集,但它们可能无法准确真实地代表药物设计中的蛋白质-配体相互作用。对于非共价相互作用,这些数据集通常仅包含简单的有机分子。即从实际的蛋白质-配体复合物的结构中建立了一个很小的非共价复合物数据集,但是其中的相互作用是由非常小的片段代表的。为了使模型系统与药物设计应用更加接近,数据集必须变得更大,更多样化。由此提出了两个新型数据集,它们来源于片段化的蛋白质-配体复合物。
方法
01
数据集构建
用于PLF547数据集构建的17种蛋白质配体复合物基于PDB数据库中的以下晶体结构:10GS,2CET,2FVD,2OBF,2P4Y,2VOT,2VW5,2XB8、2YKI,2ZX6、3G0W,3GCU ,3GNW,3JVS,3NOX,3PE2和4GID。从参考文献中获得了用于QM计算的结构(包含氢原子)。这些配合物中的配体尺寸范围为37至95个原子,其净电荷为-1、0或+1。它们都含有芳香族杂环。五个配体包含二价的四面体硫原子,其中四个和三个分别包含F和Cl原子。为了生成与配体相互作用的蛋白质片段,C和Cα之间的键被切割并被H原子以0.767Å的距离封端(保证其不带电),将主链片段化为代表主链段的N个甲基甲酰胺残基。Cα和Cβ之间的键断裂(脯氨酸和甘氨酸除外),并在0.769Å处加氢原子以将侧链与主链分开。片段化通过Cuby4框架的片段化界面实现自动化。片段化后,选择侧链和主链的片段,该片段具有至少一个距配体4Å的原子。此外,由于空间问题(非物理上紧密接触),去除了对应于2YKI的酪氨酸和2VW5的赖氨酸的两个片段。生成了构成PLF547数据集的547个蛋白片段-配体复合物。为了测试溶剂化能的方法,省去了另外8个碎片。这些片段包括以下内容(列出的数据集中的pdb文件的名称):2P4Y_27_tyr,2VOT_12_glu,2XB8_20_arg,3JVS_15_asp,3JVS_18_glu,3JVS_21_glu,3NOX_17_asp,3PE2_15_arg。除了每个配体的这些片段外,与配体(片段的并集)相距4Å之内的蛋白质残基的相同选择也用作完整活性位点的模型。在这17种蛋白质-配体活性位点复合物中,有2种被排除在外, 其余15种配合物及其周围的蛋白质构成了我们数据库PLA15数据集的活性位点模型部分。用于构建PLA15数据集的蛋白由259到584个原子组成,可提供有关实际应用中SQM方法性能的独特信息。
02
配体-片段相互作用的分类
PLF547数据集根据相互作用的片段(蛋白质片段和配体)是否带电荷,相互作用之间的距离以及蛋白质衍生部分的性质而分为几组。基于碎片模型电荷的基团是离子-离子(例如天冬氨酸侧链与非零净电荷的配体相互作用),离子-中性(相互作用的配偶体之一具有非零净电荷,另一个为中性)或中性。我们还将系统根据相互作用之间的距离进行分类,分为“短”(片段与配体之间的最短接触低于范德华半径总和的90%),“平衡”(介于90%和110%之间) ),“长”(从110%到130%)和“远处”(片段模型,其中相互作用的伙伴的最接近原子大于范德华半径之和的130%)。根据蛋白质部分的基团为“骨架”(蛋白质骨架片段,以N甲基甲酰胺模型表示),“芳香族”(组氨酸,色氨酸,苯丙氨酸和酪氨酸的芳香族侧链),“非极性”(丙氨酸的非极性侧链) ,亮氨酸,异亮氨酸,缬氨酸和蛋氨酸),“极性”(丝氨酸,苏氨酸,天冬酰胺,谷氨酰胺,半胱氨酸和脯氨酸的侧链),“阴离子”(天冬氨酸和谷氨酸的侧链)和“阳离子”(侧链)赖氨酸,精氨酸和质子化组氨酸的链)。一些结果以箱形图的形式表示数据集中误差的分布。用中线划分的框包含50%的数据点(中心两个四分位数)。其宽度是相应中央四分位数宽度的1.5倍。超出此误差范围的其余复合物(离群值)用点表示。
03
基准检测方法
对于PLF547数据集中片段复合物相互作用能的基准检验方法,我们选择了基于显式相关MP2-F12计算的复合方案,并采用ΔCCSD(T)方法校正。一个片段的总基准相互作用能为:
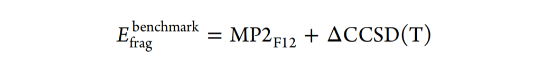
由于体系的大小(其中一些超过100个原子),MP2-F12方法以cc-pVDZ-F12基组进行计算。研究表明使用较小的基组计算的结果要优于传统的MP2/CBS外推法。高阶校正项ΔCCSD(T)通过CCSD(T)的相关性弥补了MP2/CBS对相互作用能计算的误差。ΔCCSD(T) 部分为DLPNO-CCSD(T)/ aug-cc-pVDZ相互作用能与MP2/cc-pVDZ相互作用能之差。MP2和MP2-F12的计算是在TURBOMOLE 7.3中进行的,DLPNO-CCSD(T)的计算是在ORCA 4.0.1中进行的。
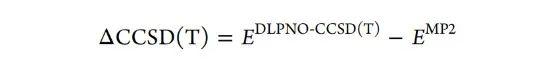
配体与完整的活性位点模型(PLA15数据集)之间的相互作用无法使用上述方案直接计算,因为这些系统包含500多个原子。因此,采用了多体效应通过DFT-D3级别(B3LYP-D3/ DZVP-DFT)计算的片段方案,其中i遍历了活性位点的所有片段,因此所有成对的贡献都在MP2-F12 + DLPNO-CCSD(T)级别上进行处理,整个系统的DFT-D3计算仅考虑了非可加性。后者主要受多体极化效应的影响,应该在DFT级别上对此进行充分说明。由于碎片相互作用能的基准是溶剂,因此我们使用基于BP/def2-TZVPD计算的COSMO-RS溶剂模型。实验通过将Turbomole与COSMOTHERM X17程序结合进行,计算公式如下:
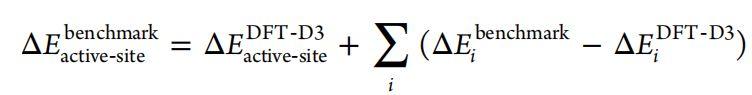
04
SQM方法测试
在数据集的片段-配体复合物部分,对QM和DFT方法进行了测试:包括MP2/ aug-cc-pVDZ,B3LYP-D3/def2-QZVP,BLYP-D3/def2-QZVP和BLYP-D3/DZVP-DFT。在D3校正中,使用了Becke- Johnson阻尼。对于以DZVP-DFT为基础的计算,我们使用了重新参数化进行矫正。计算在TURBOMOLE 7.0程序下进行。在SQM方法中,选择了以下方法进行测试:AM1,PM6,具有D3H4校正的PM6,PM7,具有D3H4和D3H5校正的DFTB和GFN2-xTB。我们在半经验方法中添加了HF-3c进行比较,AM1,PM6和PM7已在MOPAC中进行了计算,DFTB +软件中的DFTB,GFN2-xTB使用了方法作者提供的XTB代码在ORCA进行了基于HF-3c的计算。并在Cuby4下进行数据集的计算;对于溶剂化,将HF/6-31G * /SMD方法作为另一种高级方法,并将以下SQM方法作为测试对象:PM6/COSMO,PM6/COSMO2,PM7/COSMO和PM7/COSMO2,DFTB3/SMD和DFTB3/PCM。
结果
01
基于活性位点模型PLA15数据集的测试结果

图1.相互作用能的相对误差分布
图片来源JCIM
本文的主要结果是对PLA15数据集中的活性位点模型的近似方法进行了评估。AM1和PM6相比带有校正的SQM方法的性能差很多,平均相对误差分别为55.9%和31.5%。PM7相对误差为-24.1%,PM6-D3H4和DFTB3-D3H4的误差分别为-9.4%和-11.1%。其中最好的方法GFN2-xTB误差为(8.1%),它提供了SQM最小的平均误差,但误差分布较大。第二好的方法DFTB3-D3H5(-8.4%)具有最小误差分布,并在较小的基组上达到了DFT-D3的精度(B3LYP-D3/DZVP-DFT,平均误差为-7.2%)。仅考虑误差的随机部分(相对于平均值的平均绝对偏差)时,DFTB3-D3H5在SQM方法中表现最佳,其次是DFTB3-D3H4,然后是PM6-D3H4和GFN2-xTB。
02
基于PLF547数据集的测试结果
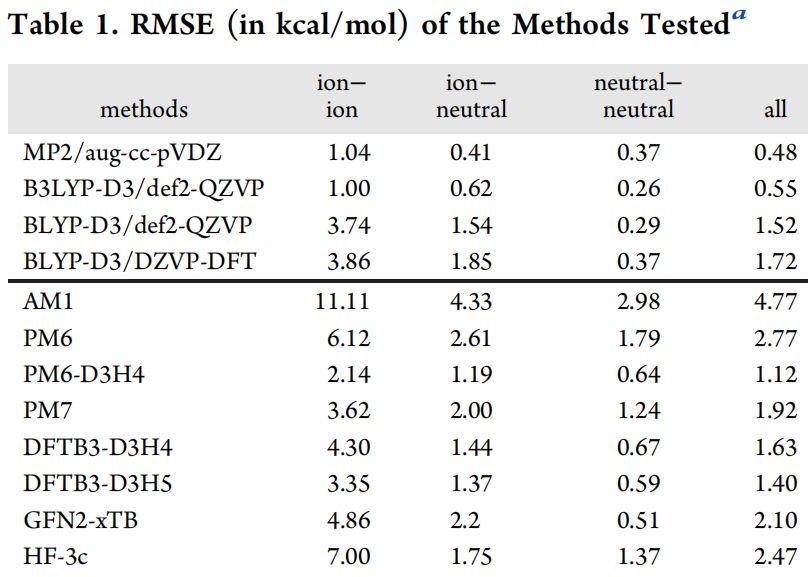
表1.测试方法的RMSE(以kcal/mol为单位)
图片来源JCIM
总相互作用能准确度和不同电荷下能量的准确度:
对QM方法,MP2和B3LYP-D3/def2-QZVP较准确的结果。相比之下,BLYP-D3/def2-QZVP准确性有所降低,最明显的是对带电物质进行计算的结果 结果表明对大多数体系的精度很好,但对于复杂的系统具有明显的误差。其中BLYP-D3/DZVP-DFT的准确度更低, 所有这些QM方法都为中性片段复合物提供了准确结果。但是比SQM计算昂贵。PLF547数据集(体系包含从44到114个原子)上采用BLYP-D3/DZVP-DFT计算一个体系的平均CPU时间是540 s,使用HF-3c的CPU时间是170 s(此处使用的是较小的基组,但是HF比GGA DFT更昂贵),而使用PM6或PM7则不到一秒钟。
对于SQM方法,PM6-D3H4,DFTB3-D3H4和DFTB3 D3H5对于中性配合物计算最为准确,GFN2-xTB在所有SQM方法中均具有最低的RMSE,但总体而言效果不佳。它仅胜过AM1,PM6和HF-3c方法。没有进行任何校正的PM6严重低估了整体的相互作用能,但PM7在高估了相互作用,这可以归因于高估了长程色散作用,DFTB3-D3H5误差最少,其次是DFTB3-D3H4,PM6-D3H4和GFN2-xTB。
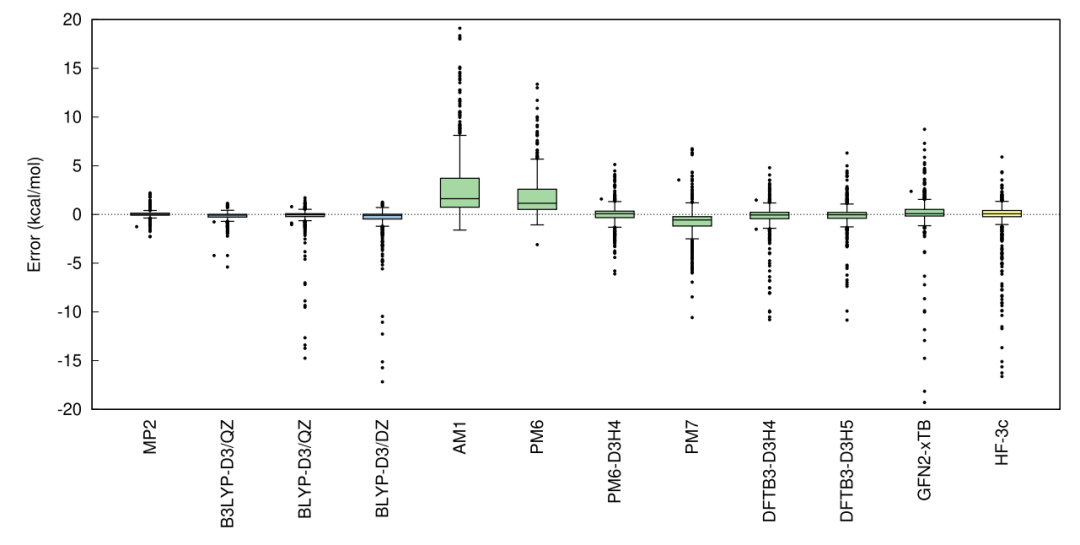
图2.相对于基准方法,片段数据集上测试方法的误差分布
图片来源JCIM
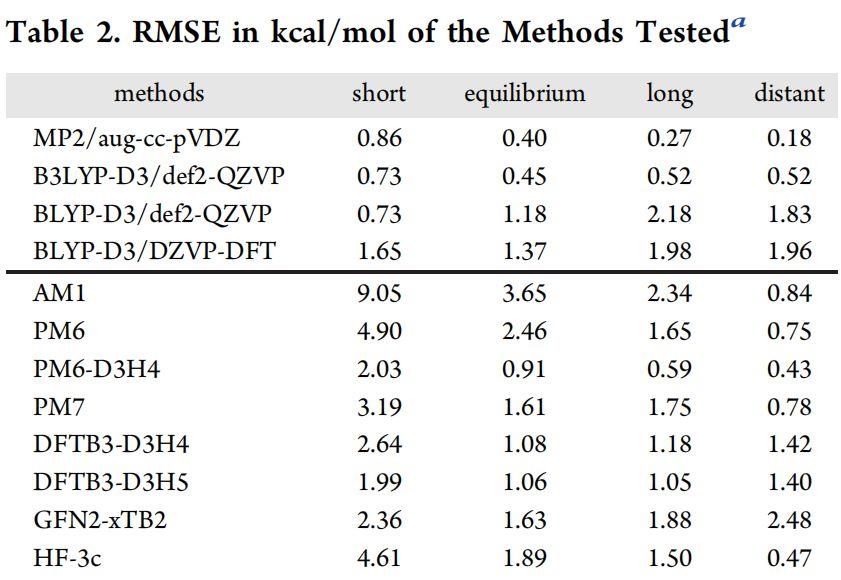
表2.按照相互作用片段间距离排序的测试方法的RMSE
图片来源JCIM
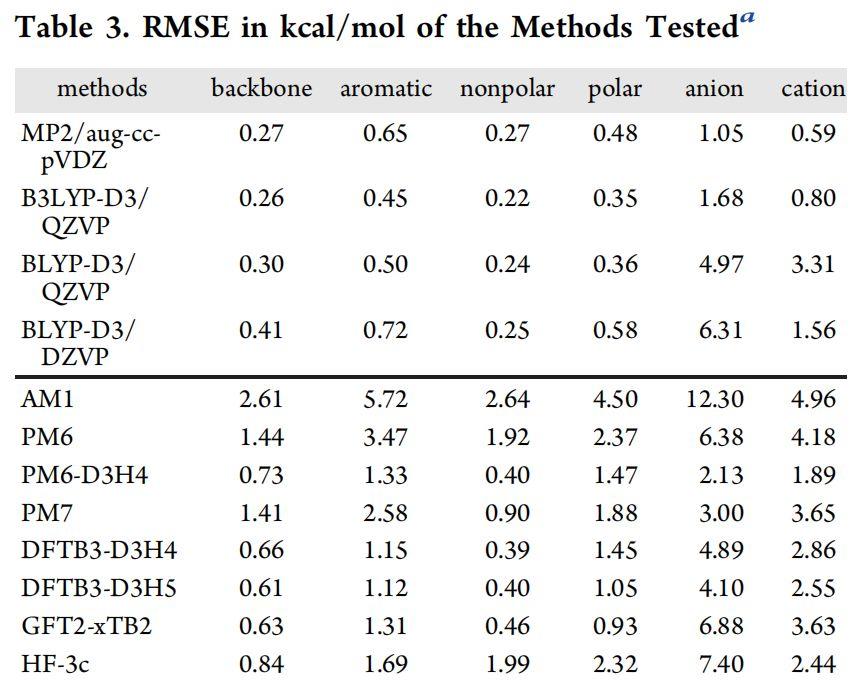
表3.按照蛋白质类型归类的测试方法的RMSE
图片来源JCIM
不同相互作用距离的准确性比较:
当根据相互作用片段的最近距离对PLF547配合物进行分类时,对于某些SQM方法,RMSE不会随距离增大而适当减小。相反,对于QM DFT和SQM DFTB3方法,范德华半径总和的130%以上的复合物的RMSE大于平衡距离附近的复合物。PM6-D3H4由于没有离域误差,因此在更长和更远的距离上比其他任何半经验方法都具有优势,并且在较短距离上的性能与DFTB3-D3H5相当。对于包括GFN2-xTB在内的DFTB3方法,很难在更长的距离上进行比较,因为它们也受到上述自交互误差的影响。PM7的精度不会受到自相互作用误差的影响,但随着分子间的分离,PM7的精度不会提高,就像PM6-D3H4一样,其色散校正在较大的间距时被高估了。
蛋白质部分类型的错误:
蛋白质片段按与配体相互作用的蛋白质种类进行分类。这些基团是主链(具有N-甲基甲酰胺模型),芳香族(His,Trp,Phe或Tyr的侧链),非极性(Ala,Leu,Ile,Val或Met的侧链),极性(N的侧链)。Ser,Thr,Asn,Gln,Cys或Pro),阴离子(Asp或Glu的侧链)和阳离子(Lys,Arg或质子化的His的侧链)。在非极性复合物中,所有对非共价相互作用进行了校正的SQM方法都表现良好。区别是,对于SQM方法,涉及芳环的配合物的描述更困难。则相互作用(包括被高估的硫)的强烈影响导致HF-3c对于非极性配合物有较高RMSE,对于一些配体中氯原子的相互作用描述也并不准确,在极性和离子基团中有较大的差异。对于HF-3c方法,最大误差来自对阴离子的描述,并且在大多数组中,该结果优于或等同于PM6-D3H4和DFTB3方法。但HF-3c方法对于阳离子-蛋白片段复合物的描述相对较好(见表3)。当蛋白质的骨架NH基团与配体(通常为氢键)形成紧密接触时,PM7往往会高估相互作用。在HB104氢键数据集中也观察到此错误。
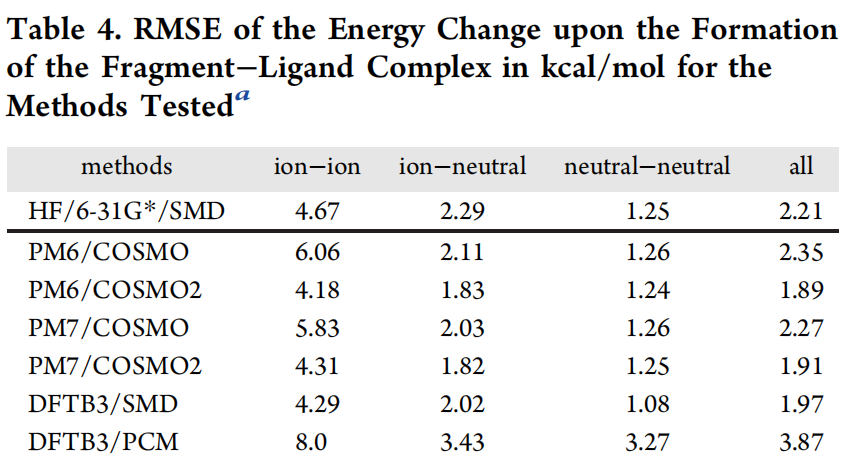
表4.对于测试方法,片段配体配合物形成时能量变化的RMSE
图片来源JCIM
03
基于PLF547溶剂化模型准确性测试结果
除DFTB3/PCM会产生较大的误差,尤其是对于中性系统,所有SQM方法的准确性均相当。相对于我们的基准,某些SQM方法甚至提供了比昂贵得多的HF/6-31G */SMD更精确的结果。COSMO-RS被认为是这类系统的最佳溶剂化方法之一,但仍未达到相互作用能计算(方法,基准计算)中基准方法达到的精度水平。因此,在这种情况下不能直接说明SQM优于QM方法。但具有COSMO2性能的PM6和PM7提供的精度最接近COSMO-RS。首先是DFTB3 / SMD,HF / 6-31G */SMD,然后是带有COSMO的PM6和PM7。DFTB3 / PCM的准确性最低。当分析单个片段复合物时,对于大多数弱相互作用系统,DFTB3 / PCM低估了相互作用(始终提供更高的正值)。这可能归因于排斥能的过高估计。对于较强的相互作用时,通常是在带电基团或离子对之间,会过高的估计相互作用能。在这种情况下排斥力大于吸引力,误差不能消除,因此PCM对于中性和带电系统都会产生相对较大的误差。对于弱相互作用的中性体系,根据系统不同误差不同。相对于各自的基准,通过这些模型得出的溶剂化相互作用能估计值比通过测试的SQM方法进行的相互作用能计算得出的绝对误差更大。这意味着在进一步开发药物设计方法时,最好集中精力于对溶剂作用的改进。大多数情况下是由于训练集中存在带点体系而导致电荷体系的描述有所改善。通过中性体系的RMSE对比,COSMO2相对于COSMO略微改善了对非极性物质的计算结果,对于中性极性物质的描述精度稍差。
04
PLA15数据集中的溶剂化测试结果
在为PLF547数据集选择的基组(B-P/def2-TZVPD,COSMO-RS)的合理水平下计算活性位点溶剂化相互作用能是不可行的。重要的是量化大型模型中溶剂化能变化的整体误差,以便将其与相互作用能的误差进行比较。因此,此处讨论的PLA15结果可通过各个片段中的溶剂化能差之和来近似。所有SQM模型都低估了能量,而HF/6-31G */SMD则高估了能量。DFTB3/SMD是性能最好的方法,而所有COSMO方法都具有相似的准确性。对比这些方法的RMSE,PM6/COSMO2和PM7/COSMO2是最准确的,但是除DFTB3/PCM和HF/6-31G */SMD以外,所有方法都具有较好的准确度。根据相对百分比和RMSE,PM6 / COSMO2随(参考)能量的绝对大小而变化,而DFTB3 / SMD以基准参考的一小部分表示时,会提供更好的结果。DFTB3 / SMD(与DFTB3 / PCM不同)为中性配体配合物提供了更准确的估计值,因为没有离子对相互作用,因此能量的绝对大小趋向于更小。从图3中也可以明显看出,DFTB3/PCM精度不足。在PM6和PM7中从COSMO过渡到COSMO2并不能仅在基准数据集中改善结果。对此之前的测试对碳酸酐酶II抑制剂进行评分,结果表明COSMO2系统地改善了得分与实验结合自由能的相关性。
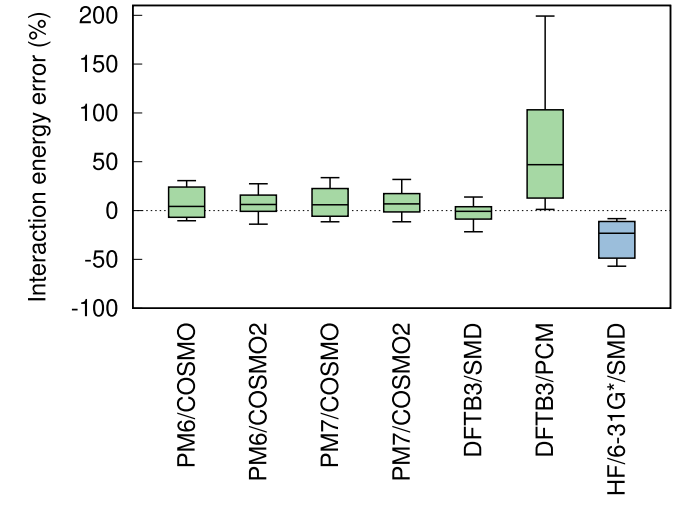
图3.蛋白质-配体复合的溶剂化能量变化的误差
图片来源JCIM
分析与总结
1. 这项工作引入了两个数据集PLF547,PLA15,用于测试半经验方法(SQM),重点是它们在计算机辅助药物设计中的应用。PLF547数据集包含与配体相互作用的547个蛋白片段(氨基酸侧链和主链片段),而PLA15集包含15个完整的活性位点AA5Q配体模型。相互作用能量基于显式相关的MP2-F12和DLPNO-CCSD(T)校正的组合进行计算,可解决基组的收敛和高阶相关效应带来的误差。
2. 使用这些数据集评估了该领域中使用较为广泛的几种半经验QM方法的准确性。基于数据集,PM6-D3H4方法提供了最好的结果,其次是DFTB3-D3H5。比起这些方法的实际应用,更重要的是它们在PLA15数据集的配体-活性位点相互作用的计算结果。其中DFTB3-D3H5是最成功的方法,其精度在较小的基组中接近DFT-D3计算的准确度,且误差分布远小于其他测试方法。
3. 对于结合时溶剂化能的计算,DFTB3/SMD,PM6/COSMO2和PM7/COSMO2溶剂化模型最准确。但溶剂模型的精确度仍然比气相相互作用能的精确度差得多。因此,溶剂化限制了在药物设计中的应用,应在以上方面做出改进。
参考文献
Benchmarking of Semiempirical Quantum-Mechanical Methods on Systems Relevant to Computer-Aided Drug Design. Kristian Kříž and Jan Řezáč. J. Chem. Inf. Model. 2020. DOI: 10.1021/acs.jcim.9b01171