先睹为快
靶点
病原菌,超级细菌
模型训练库
FDA数据库(1760个分子)
天然产物(800个分子)
筛选库
药物库:Drug Repurposing Hub(6111)
实体库:ZINC15(~1亿类抗生素分子)
计算方法
有向消息传递的深度神经网络(Directed message passing neural network)
计算软件
RDKit, Chemprop
计算流程
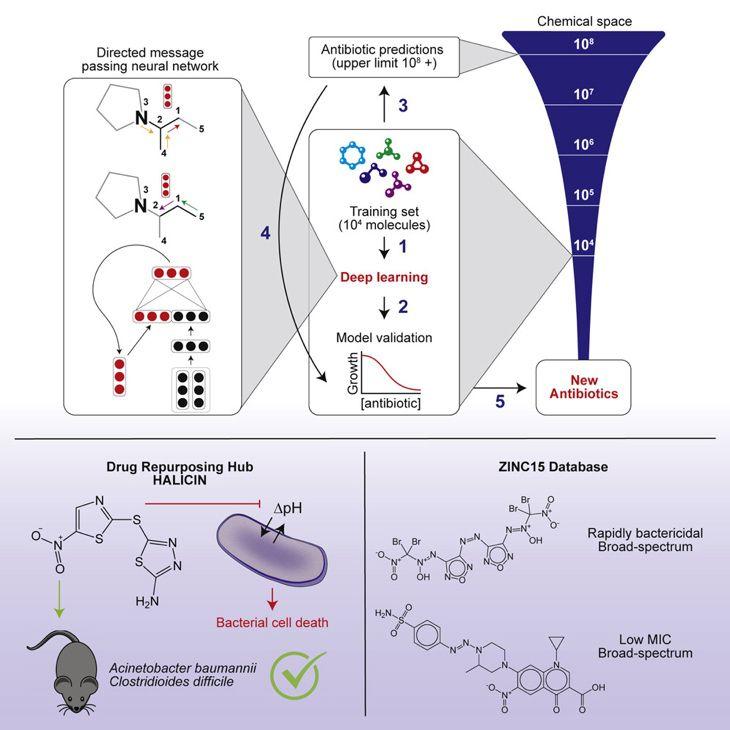
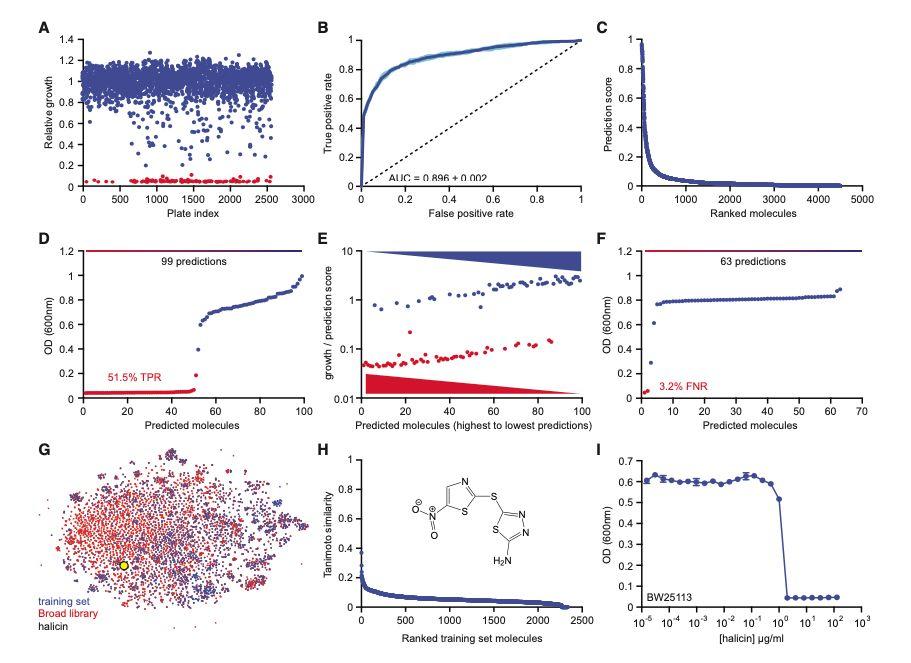
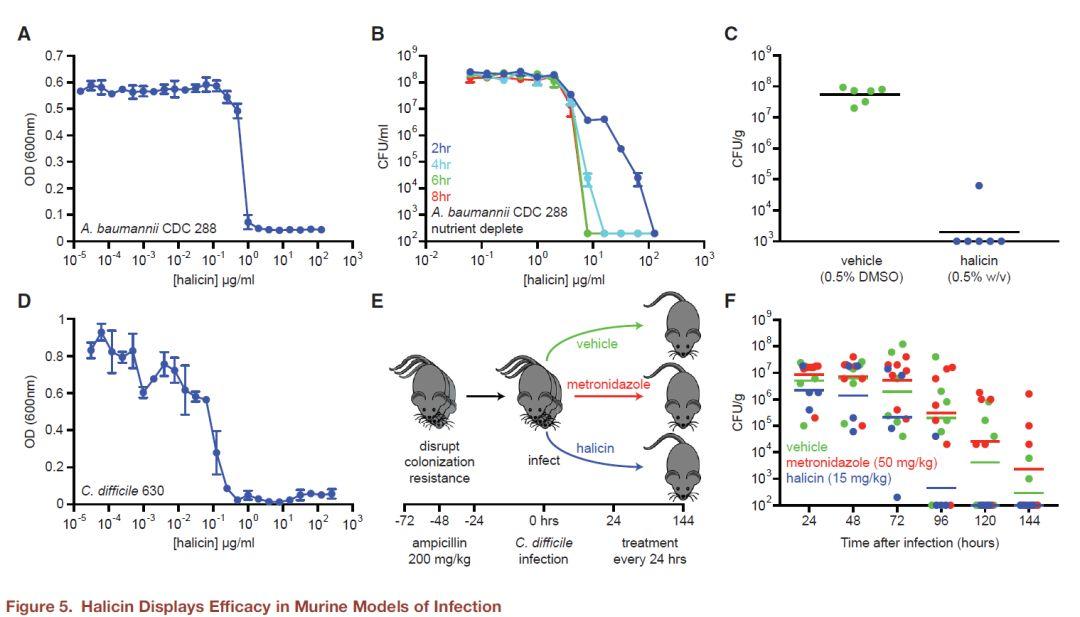
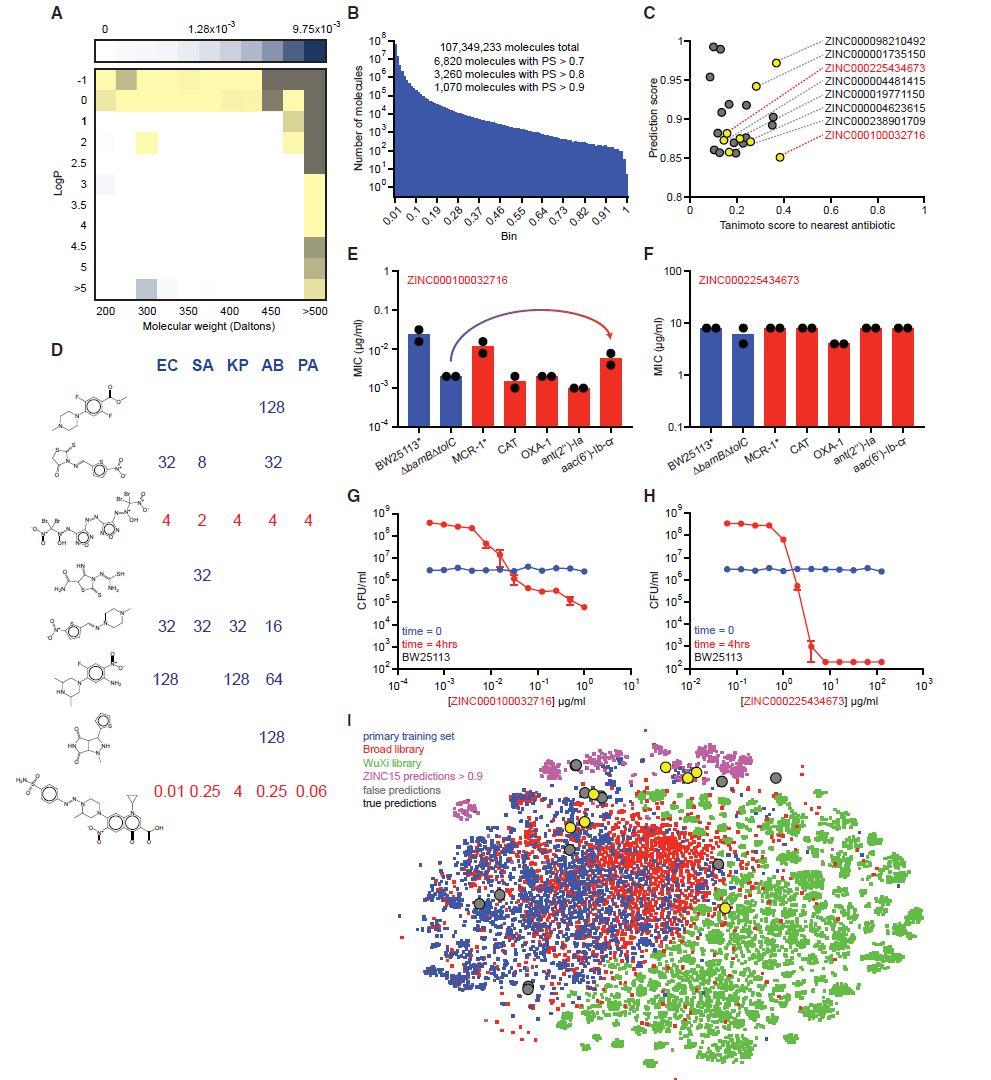
作者首先利用药物库和天然产物构建了一个训练集(2335个分子),并根据对大肠杆菌的抑制活性将化合物分为两类:hits或者non-hits。随后使用有向消息传递的深度神经网络训练模型,模型AUC值达到0.896。利用这个模型对药物库(Drug Repurposing Hub,6111)筛选发现具有广谱抗菌效应的新型抗生素halicin(糖尿病药物)。随后对ZINC15中约1亿类抗生素分子进行筛选,选择预测打分>0.8并且与已知抗生素相似度<0.4的23个化合物,测试发现8个至少对一种细菌有效,其中2个具有广谱抗菌效果。

有向消息传递深度神经网络
作者使用有向消息传递深度神经网络(directed-message passing neural network),直接从分子的图结构中预测分子特性,其中原子表示为节点,键表示为边界。作者重建了与每个化合物的SMILES字符串相对应的分子图。接下来,作者初始化了一个特征向量,基于单个原子和键的可计算特征:原子特征:原子序数,每个原子的键数,电荷,手性,氢键数,杂化,芳香性,原子量。键特征:键类型(单/双/三/芳香),共轭,环成员,立体化学。该模型应用了一系列消息传递步骤,在该步骤中,它将来自相邻原子的信息汇总并结合,以建立对局部化学的理解。经过固定数量的消息传递步骤后,对分子中学习到的单个特征进行求和,以生成整个分子的单个特征。这种特征化通过前馈神经网络进行馈送,输出预测值。作者使用分子是否抑制大肠杆菌生长作为分类标准,因此训练该模型输出0或者1来表示它对于输入分子的预测。
总结
作者使用深度学习构建了一个预测抗生素的筛选模型,特征向量使用有向消息传递深度神经网络构建,而后使用前馈神经网络进行训练,鉴别出Halicin具有潜在的抗菌活性,且与已知的抗生素结构差距较大,后期又进行了生物活性测试,在动物模型上,halicin疗效显著。后期又对ZINC数据库进行了预测,发现了8种与已知抗生素结构差别较大的分子。其中2中具有广谱抗菌效果。
图片来源:Cell
图片来源:Cell
图片来源:Cell
图片来源:Cell
参考文献
Stokes, J.M., et al., A Deep Learning Approach to Antibiotic Discovery. Cell, 2020. 180(4): p. 688-702 e13. DOI: 10.1016/j.cell.2020.01.021