背景介绍
近年来,研究人员已经确定了某些RNA可作为小分子治疗几种人类病理的药物靶点。然而,由于其固有的结构灵活性和较差的结构-功能关系,对这些RNA的调控是极具挑战性的目标。因此,研究人员对标准的药物发现方法进行了调整,以识别和优化RNA靶向小分子。结构化RNA是药物发现的一个突破,这些分子调节着许多重要的细胞功能,它们参与了从癌症到病毒和细菌感染等多种人类病理的发展。利用小分子药物可以靶向结构化RNA,从而有益地调节其生物学功能。RNA特异性计算工具的发展,有望帮助RNA靶向药物的合理设计,就像类似的工具可帮助蛋白质靶向小分子药物的发现一样。在此背景下,研究者在这篇综述中,概述了调控RNA的结构和功能特征,并通过代表性的案例研究,批判性地审视了分子建模、对接和模拟的威力和挑战,以及基于RNA靶向结构的药物设计中计算工具与实验方法的协同互联。相关的综述以“Computer-aided design of RNA-targeted small molecules: A growing need in drug discovery”为题发布在国际著名期刊Chem上。
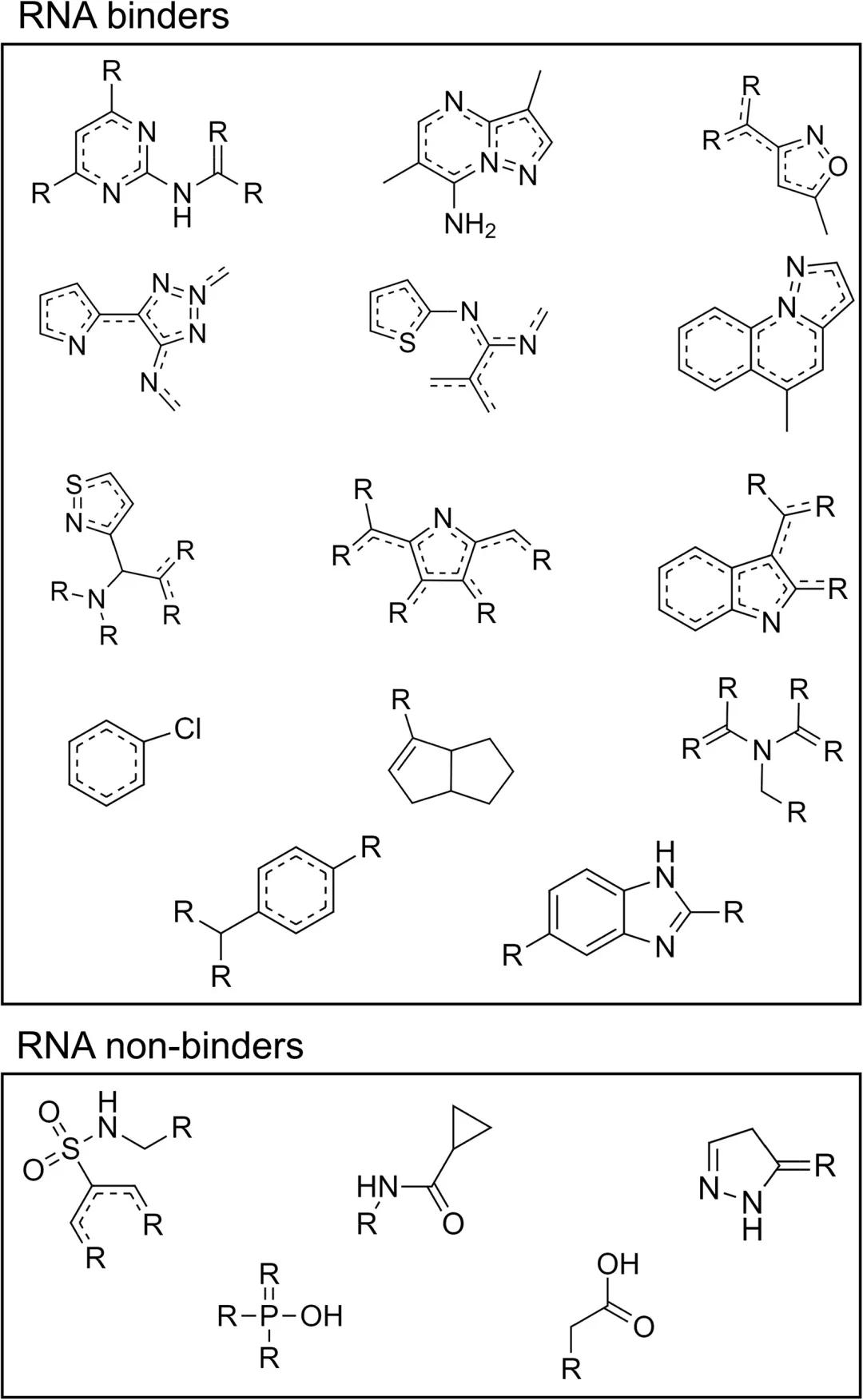
小分子可以靶向功能性重要的结构化RNA
RNA分子可以控制重要的细胞过程,如翻译、剪接、基因表达、免疫和代谢蛋白调控等。这些RNA分子,通常采用对其功能至关重要的复杂三级结构。而有机小分子化合物可以对RNA的3D结构进行精确靶向,尤其是靶向RNA的蛋白质识别位点。在其他情况下,结构化RNA在功能水平上也可以由有机小分子进行调节。RNA的3D结构,也可能是调节其长链非编码RNA(lncRNAs)功能的关键(长链非编码RNA是一种新发现的转录本,通过表观遗传机制参与基因表达调控和染色质结构)。种种研究表明,ncRNA结构,在调节关键细胞过程中发挥着非常重要的作用。同时,结构化RNA也是许多严重疾病治疗的潜在靶点,包括从癌症到病毒和细菌感染等等。
RNA结构的实验测定正在获得动力
并将促进计算研究
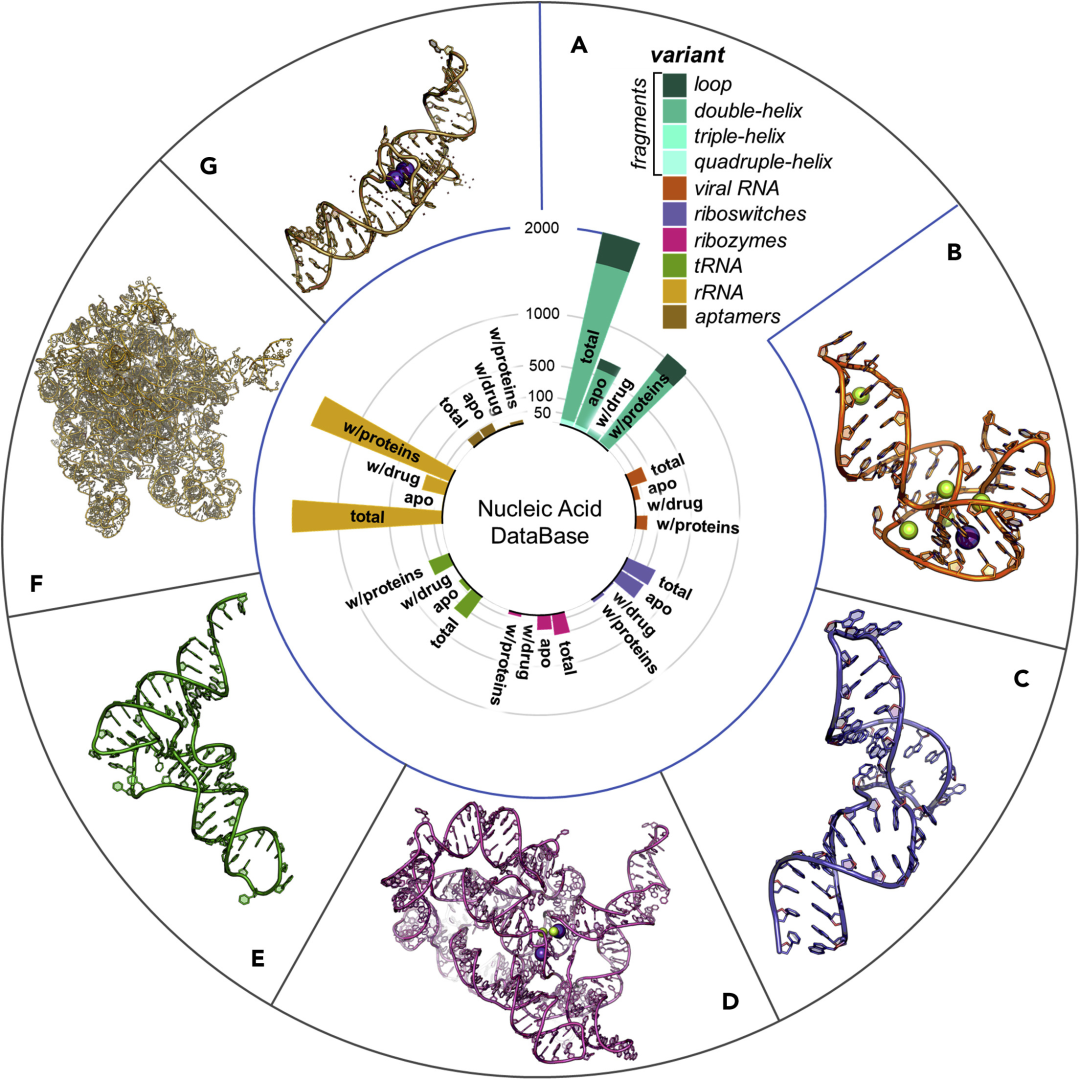
RNA结构,是影响RNA功能的关键因素。然而,RNA的结构测定具有挑战性,而且进展并不像其他生物大分子(如蛋白质)那样迅速。截至2021年4月1日,核酸数据库中只包含了906种RNA-小分子复合物晶体,这里面还包括了细菌rRNA和抗生素类小分子的复合物晶体,这些晶体结构,正帮助研究人员了解氨基糖苷类药物的作用模式,并可能促使发现克服细菌多药耐药的新药。
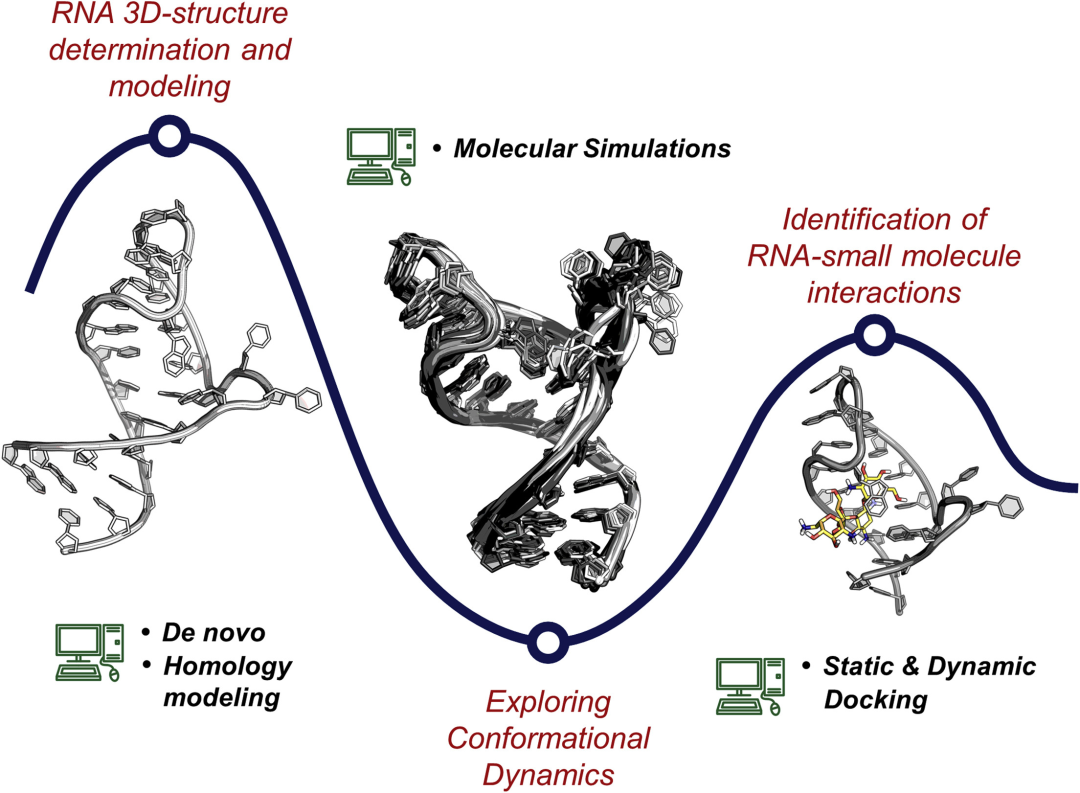
预测、建模和设计RNA结构的计算方法
(1) 基于RNA序列的从头RNA 3D结构预测
未知RNA三维结构的预测通常采用从头算(或者无模板)方法,可分为基于物理的方法和基于片段的方法。基于物理的方法,是利用分子动力学(MD)或蒙特卡罗(MC)框架中的参数化能量函数,来识别给定RNA序列的最低自由能构象。其中,有三种基于物理无模板的免费工具:iFoldRNA、NAST和SimRNA。无模板的基于片段的方法,使用了RNA结构模块化的概念,基于此,相似的RNA子序列可以折叠成与整体结构无关的相似的3D模块(即发夹环、假结、凸起)。其相关的工具包括:FARFAR、RNAComposer、Vfold3D等。
(2) 基于实验二级结构数据的RNA三维结构预测
值得注意的是,3D RNA模型的预测,也可以得益于RNA二级结构的化学制图(即通过诸如SHAPE或DMS探测等技术实现的)或RNA三级相互作用(即通过羟基自由基足迹或基于交联的技术获得的,如PARIS)所产生的结构数据。事实上,这些结构数据可以包含在靶向RNA的3D建模中,以支持先验已知结构基序(例如螺旋)的计算,而这些结构基序的折叠尚未计算。
(3) RNA 3D结构的同源建模
同源建模方法,需要一个或多个完整RNA模板的3D结构来生成查询序列的3D模型,目前主要包括两个计算工具:RNABuilder和ModeRNA。尽管存在诸多局限性,但同源建模已经成功地预测了复杂的RNA结构,如识别和正确塑造RNA配体的结合位点。
(4) 新型结构化RNA分子的计算设计
3D RNA结构的预测,还可用于识别和设计基于RNA治疗的具有特定结构特性的新型RNA分子(例如,合成RNA适配体用于生物标记物的选择性结合)。例如,Schlick实验室利用基于片段的RNA结构设计方法,就是建立在RNA-as-graph (RAG)粗粒化框架之上。这种方法允许从高抽象水平(即图形)开始设计新的RNA分子,这意味着不需要先验地知道RNA靶标的信息。
综上所述,这些方法和方式,展示了计算科学界的密集研究活动是如何推动了3D RNA建模的多种方法的持续和快速发展,以解决复杂RNA基序具有挑战性的结构预测。
移动中的RNA:利用分子动力学模拟来探索RNA的功能灵活性
(1) RNA力场:最新的改进和目前的限制
分子力学力场近似于粒子间的相互作用。力场可以分为两大类:全原子和粗粒化。直观地,根据全原子表示,系统的每个原子被定义为一个模型粒子。而粗粒化建模通过将特定的原子组分组成所谓的珠子,每个珠子代表一个给定的结构特征(例如,糖、碱基和主干),从而降低了模拟一个大系统的成本。粗粒化模型,虽然可以有效地再现大RNA分子的物理化学特性。然而,忽略原子细节,会导致计算机辅助药物设计的重大缺点,妨碍药物-靶标相互作用的准确识别。最近的一些研究,已经提高了全原子经典RNA力场的准确性,主要包括:
(a) AMBER99力场的一个改进是对RNA主链扭转角的改进;
(b) 基于量子力学/分子力学(QM/MM)计算,通过重新参数化AMBER-parmbsc0力场的α/γ扭转项,提高了RNA和DNA双螺旋的MD模拟精度;
(c) 在AMBERχ力场序列中重新参数化RNA糖苷的χ扭转角,纠正了高抗梯状RNA结构的形成;
(d) 在AMBER之外,CHARMM社区开发了一套新的RNA参数来提高MD模型的可靠性;
(e) CHARMM的一个进展是,在经典Drude振子模型的基础上发展了第一个可极化的核酸力场。
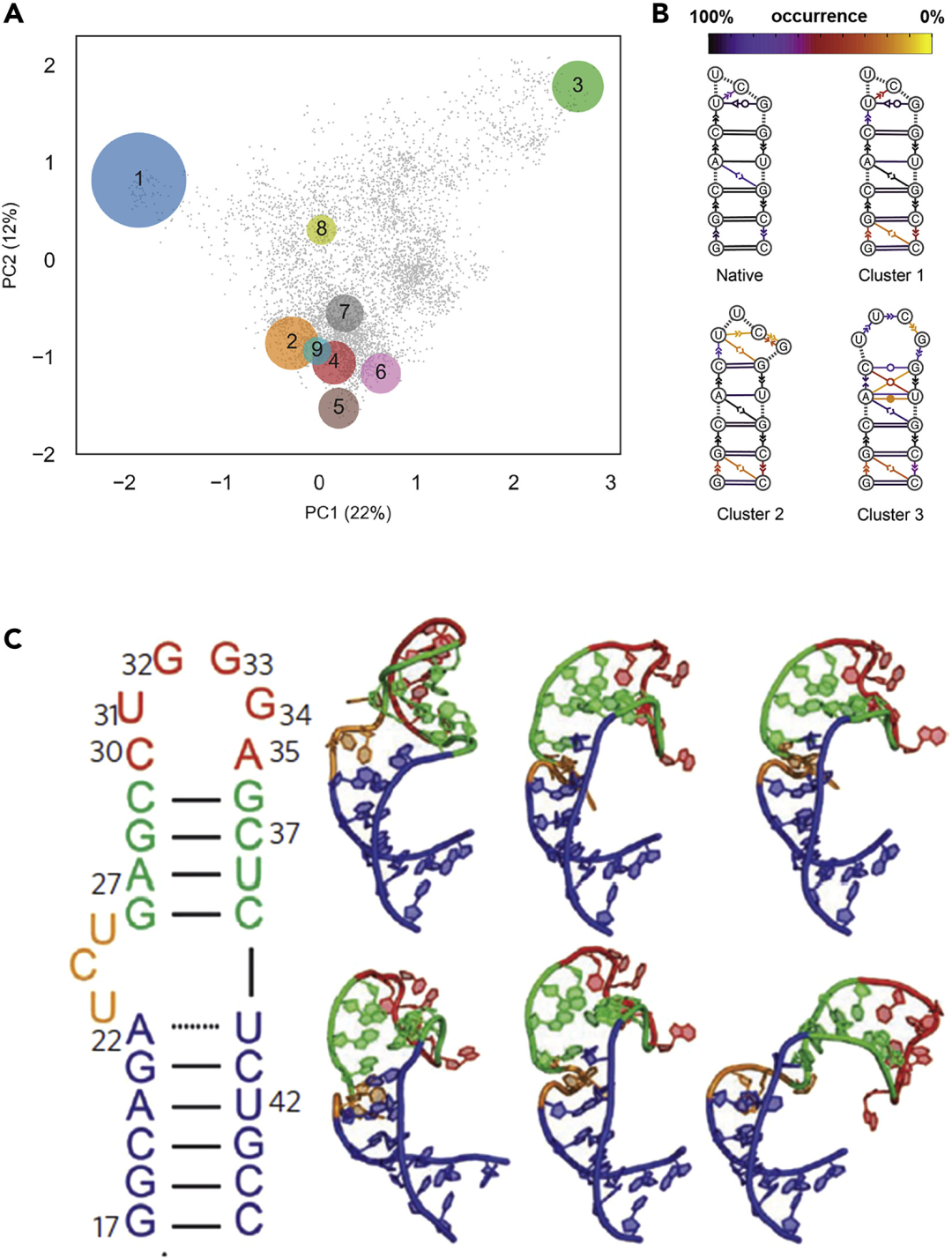
(2) 催化RNA的构象动力学
尽管MD目前存在局限性,但其可以提供详细了解RNA分子的功能动力学。实际上,MD已经表征了几种核酶的作用机制,这些核酶是RNA系统,可以在RNA/DNA主干上以顺式(即自裂解)或反式(即非自裂解)催化磷酸水解。此外,许多计算研究也已经阐明了这些小的反应性RNA的催化机制,包括HDV、hairpin和glmS核酶等。较大的II族内含子自剪接核酶,在核酶催化中碱基的稀有质子化态中发挥了功能作用。这些核酶是抗真菌药物的重要靶点。而对II族子结构动力学的深入了解,有助于提高研究人员对这些核酶作用机制的理解,促进了针对这些RNA系统的新药的合理设计。
(3) 对实验和模拟的集成获得了生物物理可靠性
对于那些需要详尽的组态采样(即长模拟时间)的限制,MD通常可以与实验数据协同集成,例如:
(a) 自20世纪90年代以来,MD模拟已经与核磁共振实验相结合,以表征HIV-TAR元素的结构动力学;
(b) 除NMR外,研究人员还利用小角度和广角X射线散射(分别为SAXS和WAXS)并结合MD模拟,获得了溶液中的RNA结构数据。
RNA-小分子相互作用的鉴定
(1) RNA的分子对接工具
最近3D RNA结构的解决方案,促使人们大力开发和应用分子对接技术,以加快基于RNA靶向结构的药物发现。例如,内部坐标力学(ICM, Molsoft)是一种分子建模/对接平台,已成功用于识别RNA结合物。为了克服分子对接中潜在的缺点,最近开发了几种RNA特异性对接/评分方法,主要包括:AutoDock、MORDOR、rDock以及RLDOCK等。
(2) RNA特异性的独立评分功能
除了RNA特异性的分子对接平台之外,还有许多独立的评分功能来提高一系列配体姿态的排序,主要包括:KScore、DrugScoreRNA、LigandRNA、SPA-LN112、ITScore-NL、RNAPoser以及AnnapuRNA等。
(3) RNA配体结合动力学与能量学的表征
MD可以成为探索配体结合和解结合的功能动力学和能量学以及进行动态对接的实用方法。MD也可解释RNA和配体在结合过程中的灵活性,克服静态对接协议的固有限制。相关的应用主要包括:
(a) MD已经成功地描述了嘌呤感应核糖体开关的配体识别和结合过程;
(b) 利用MD模拟研究了PreQ1核糖体开关的作用机理;
(c) 用交换偏置MD描述了SHAPE试剂和RNA分子之间的相互作用,揭示了RNA的构象动力学和糖的褶皱是如何增加有利于SHAPE反应的2-OH基团的可及性的关键;
(d) 自由能扰动(FEP)也可以评估特定突变对RNA配体结合自由能的影响,例如,鸟嘌呤核糖体开关;
(e) 监督MD (suMD)可以在短时间窗口内高效地复制RNA-配体结合模式,但它不能被盲目地用于预测药物结合的物理途径。
靶向RNA调控的计算机驱动基于结构的药物发现
Kang及其合作者证明了RNA靶向药物发现的实验和计算的协同作用,他们确定了miR-21的特定小分子抑制剂,可作为一种新的上皮抗癌药物。
RNA选择性抑制研究中,Baranger和合作者战略性地靶向了RNA分子中的保守结构基序。考虑到RNA和配体的灵活性,研究者利用短(5ns)平衡分子动力学(MD)模拟,选择了6个与目标形成稳定配合物的配体。这些MD模拟强调了氢键和电荷静电相互作用在稳定配体结合方面的重要性。
Al-Hashimi小组最近展示了MD在模拟、预测和开发RNA灵活性方面的效率。此外,Al-Hashimi小组还使用了HIV-1 TAR RNA广泛的MD模拟,从一个扩大的实验数据集(包括四组RDC)中生成另一个TAR结构集合。他们对10万个类药物小分子进行了高通量筛选(HTS),以识别TAR结合剂和非结合剂。
最后,基于结构的虚拟筛选,还指导了一种SARS-冠状病毒(SARS-CoV)复制小分子抑制剂的发现。
总之,这些研究表明,独立的计算工具和混合实验计算方法,可以用来识别和合理设计靶向调控RNA的新的小分子配体。
图表汇总
结论总结
本篇文章中,研究者阐述了靶向RNA在药物发现中最近的进展,重点是与RNA小分子相关的结构和计算模拟。此外,研究者还讨论了目前面临的主要挑战。调控RNA参与了从癌症到微生物感染等多种人类病理的发展,因此是治疗干预的一个有希望的目标,从而促使了对实验生化和生物物理特性的研究,证明了调节RNA是小分子的可用药靶点。一系列的前沿进展,激发了开发以RNA为靶点的常规药物的研究,制药公司正寻求开发以RNA为靶点的小分子疗法,其中一些已经进入临床开发的最后阶段。