背景介绍
高精度的蛋白质结构预测,有助于从分子水平上理解生物过程。在过去的几年中,由于深度卷积残差网络(ResNet)以及最近在AlphaFold2中实现的注意力网络的引入,蛋白质结构预测有了很大的进展。然而,相当大比例预测的蛋白质结构模型仍然偏离其真实结构,从而限制了它们在下游应用中的价值。为了进一步提高模型质量,人们在开发模型优化方法方面进行了大量的工作,主要目标是将初始模型进一步优化,生成更高质量的新模型。然而,这是一个非常具有挑战性的任务,因为较差模型的空间比较好模型的空间大得多。许多CASP参与者提交的优化后模型的质量反而比他们的初始模型要差。
典型的模型优化方法,包括侧链重装、能量最小化和约束结构抽样。由于能量函数通常很难优化,如果没有大规模的构象抽样,模型质量可能无法得到提升。目前,最成功的优化方法是通过分子动力学(MD)模拟或片段组装进行大规模构象采样,例如,Feig等人采用平底谐波约束迭代MD模拟采样构象,利用Rosetta评分函数选取样本模型的子集,取平均值,建立最终的优化模型;Baker组采用局部误差估计方法,通过片段组装的方法指导构象采样,并通过重新组合二次结构片段和替换扭转角的方法迭代优化模型,利用Rosetta评分函数确定最后一次迭代的最低能量模型,然后与构象邻域进行平均,建立最终的优化模型;由Seok组开发的GalaxyRefine2采用多种构象搜索策略,模型误差估计可以约束采样空间,防止稳定结构区域的退化;DeepAccNet使用3D和2D卷积网络来估计残差精度和残差间隔距离误差,然后将其转换为罗塞塔约束来指导构象采样。尽管这些方法在某些蛋白质上表现良好,但它们依赖于广泛的构象采样,因此,即使是单个蛋白质模型也需要大量的计算资源。
目前,优化蛋白质模型以获得更好的模型质量是极具挑战性的工作。而最成功的优化方法,依赖于广泛的构象采样,因此,优化一个蛋白质模型通常需要几个小时,几天,甚至更长的时间。为此,来自美国芝加哥丰田计算技术研究所的许锦波教授等研究者,提出了一种快速有效的方法,可以在非常有限的构象采样的情况下优化蛋白质模型。该方法采用GNN(图神经网络)从初始模型预测优化的原子间距离概率分布,然后以预测的距离为约束条件重建模型。相关的研究成果以“Fast and Effective Protein Model Refinement by Deep Graph Neural Networks”为题发布在国际著名期刊Nature Computational Science上。
方法概述
研究者提出了一种新的模型优化方法——GNNRefine,它可以快速提高模型质量,而不需要大量的构象采样。GNNRefine的流程图,主要包括以下三个步骤:
(1) 将初始模型表示为图,从初始模型中提取原子、残差和几何特征;
(2) 利用图神经网络(GNN)预测图中每条边的优化距离;
(3) 将预测的距离概率转化为距离势能函数,并将其输入到PyRosetta FastRelax中,通过侧链组装和能量最小化产生优化模型。
GNNRefine主要由三个模块组成:原子嵌入层、多个消息传递层和输出层。原子嵌入层用于学习一个残基的原子级结构信息,所得到的原子嵌入与其他残基特征相连接,形成残基的最终特征。蛋白质图建立在残基特征(节点)和残基对之间的键或接触特征(边)上。通过多个消息传递层迭代更新节点和边缘特征,获取全局结构信息。最后,利用一个线性层和一个softmax函数从边缘特征预测距离概率分布。(图1)
将预测的距离概率转换为距离势能,然后将距离势能输入到PyRosetta FastRelax中,建立优化模型。在CASP13数据集上测试,当使用10个cpu运行FastRelax(生成50个优化模型)时,研究者的方法平均只需要15分钟来优化一个蛋白质模型。相比之下,Baker组的DeepAccNet需要在50个cpu上工作超过10个小时,才能优化一个有120个残基的蛋白质模型。
优化目标性能
在此,研究者将上述方法与CASP13优化类别中的两个领先的人工组(FEIGLAB和BAKER)以及5个服务器组(Seok-server、Bhattacharya-Server、YASARA、MUFold_server和3DCNN)进行了比较。评估的第一个提交的模型的质量,如表1所示;图2为ΔGDT-HA分布的箱线图。即使仅为每个初始模型生成5个优化模型,GNNRefine也具有与这两个人工组相当的性能,并且在质量改进方面优于所有5个服务器组。此外,研究者的方法只生成4个较初始模型质量稍差的优化模型,但所有其他方法包括两个人工组,都产生了许多更差的优化模型。因此,使用GNNRefine来优化模型是非常安全的。(表1和图2)
同时,研究者也在37个CASP14优化目标上测试了该方法,并也将其与两个人工组(FEIG和BAKER)以及四个服务器组(FEIG-S、Seok-server、Bhattacharya-Server和MUFold_server)进行了比较。表2总结了相关性能,图3给出了ΔGDT-HA分布的箱线图。总的来说,在CASP14的优化目标上,GNNRefine比FEIG的方法略差,与BAKER的方法相当,但优于其他方法。同时GNNRefine产生了最少的更差的优化模型。(表2和图3)
GNNRefine成功地大幅度优化了5个CASP测试模型(3个CASP13模型和2个CASP14模型)的质量,把它们的GDT-HA分数提高最少10个点。图4显示了4个已经有公开实验结构的模型,表明该方法可以在不同的二次结构区域(螺旋、片和线圈)优化初始模型。(图4)
GNNRefine优于现有的独立软件
研究者还将GNNRefine方法与一些公开可用的软件,如GalaxyRefine和ModRefiner进行了比较。GalaxyRefine采用默认配置在本地运行。ModRefiner在[0,100]中有一个可配置的参数强度,来控制从初始模型中提取的约束的强度,强度0表示没有约束,而强度100表示初始模型的约束非常严格,研究者采用了三个不同的强度值(0, 50, 100)运行ModRefiner。作为对比,研究者也不使用GNNRefine预测的距离约束来运行PyRosetta FastRelax。表3显示了在CASP13测试模型上三种方法的性能和运行时间,从所有指标来看,GNNRefine优于其他方法。(表3)
GNNRefine距离预测改善的原因
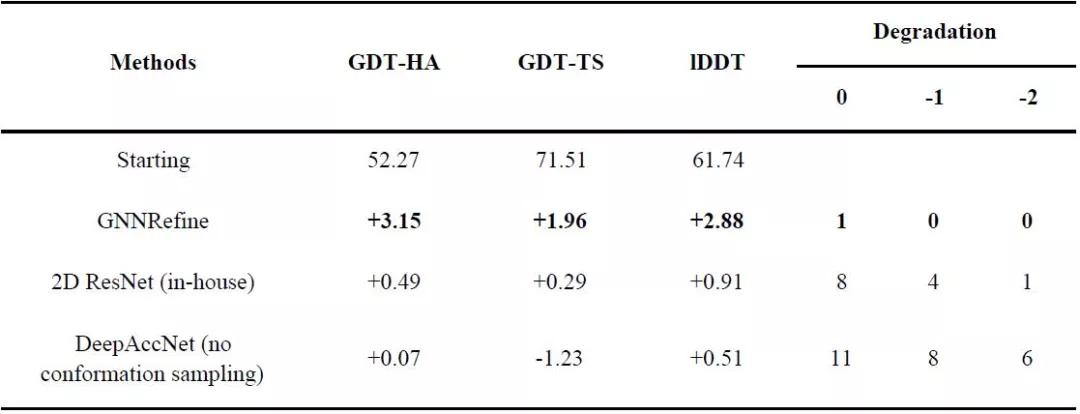
研究者从top L接触精度和lDDT两方面评估了GNNRefine预测的距离。对于每个残差对,将8Å以下距离的预测概率求和为预测接触概率。根据其各自递增的Cβ-Cβ欧几里德距离来选择初始模型中的top L接触残差对。为了计算GNNRefine预测的距离的lDDT,对于每个残差对,研究者使用预测概率最高的区间的中间点作为其距离的预测。与此同时,研究者只考虑预测距离小于20Å的Cβ-Cβ对。从表4可以看出,GNNRefine预测的距离在接触精度和lDDT方面都优于启动模型。(表4)
GNN在模型细化方面优于ResNet
卷积残差神经网络(ResNet),已经广泛应用于蛋白质接触和距离预测。Baker组开发了一种基于ResNet的DeepAccNet方法用于模型的优化。为了在有限的构象采样条件下测试DeepAccNet的性能,研究者将DeepAccNet生成的距离势能输入到PyRosetta FastRelax中,使用与GNNRefine完全相同的方法建立优化模型。此外,研究者还开发了一个内部的ResNet模型(包含41个2D卷积层)来预测与初始模型的距离,并测试其预测的距离是否可以用于优化模型。为公平地比较这三种方法,在实验中研究者只使用了一个GNNRefine模型来进行细化。
对于每种方法,研究者从每个初始模型中生成10个优化模型,并选择能量最低的模型作为最终的优化模型。表5显示,GNN方法大大优于研究者内部开发的ResNet方法,而ResNet又优于DeepAccNet。也就是说,DeepAccNet在不使用广泛的构象采样时无法优化模型,但GNN方法是有效的。(表5)
GNN比ResNet在模型优化方面工作得更好的根本原因是GNN比ResNet更容易建模多个残基的相关性。大多数蛋白质的半径与它们长度的立方根成正比,所以任何两个在一级序列上完全分开的残基都可以在蛋白质图中,通过比蛋白质长度立方根短的路径连接起来。因此,ResNet更适合于初步预测残基间关系,而GNN更适合于进一步优化残基间关系。
消融研究
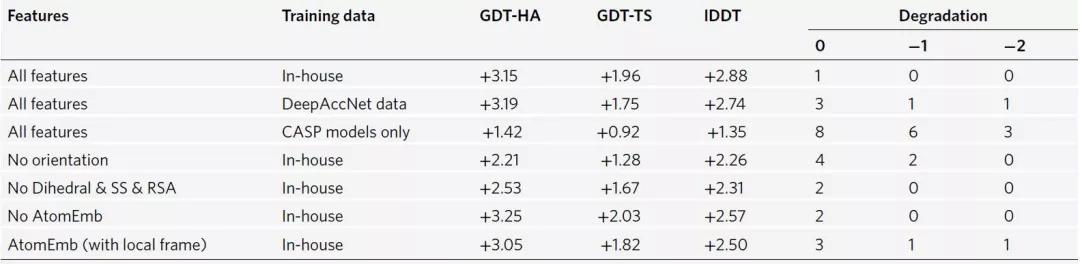
为评估单个因素对GNNRefine的贡献,研究者评估了如表6中不同数据和不同特征训练的GNNRefine模型。结果表明,大的训练数据、残差方向和DSSP衍生的特征是三个最重要的因素,而总体上原子嵌入并不能提供有用的信息。(表6)
图表汇总
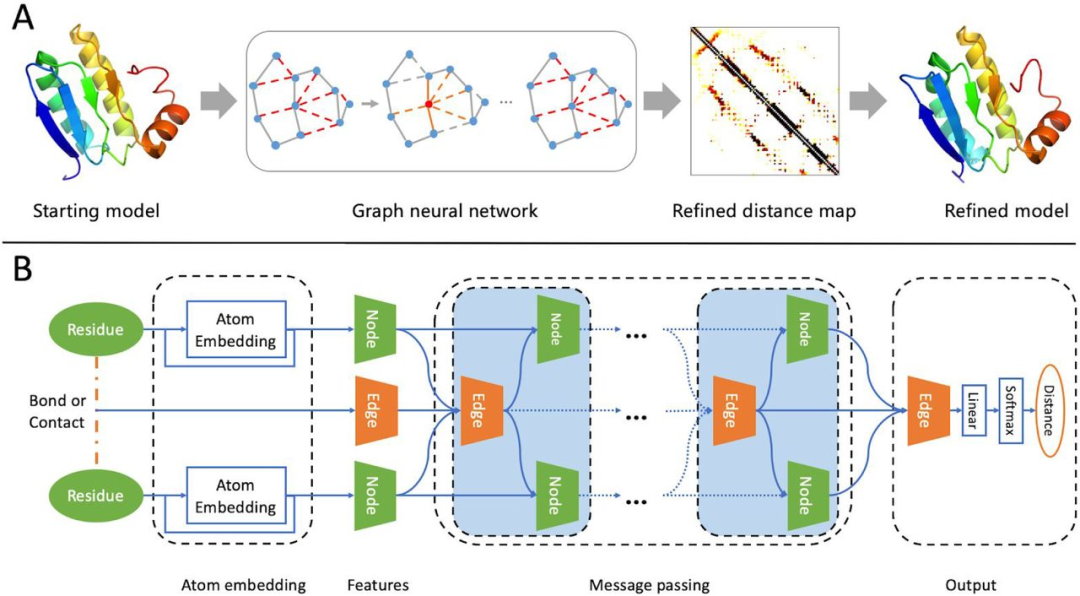
图 1. 用于蛋白质模型优化的GNNRefine方法。图片来源于Nat. Comput. Sci.
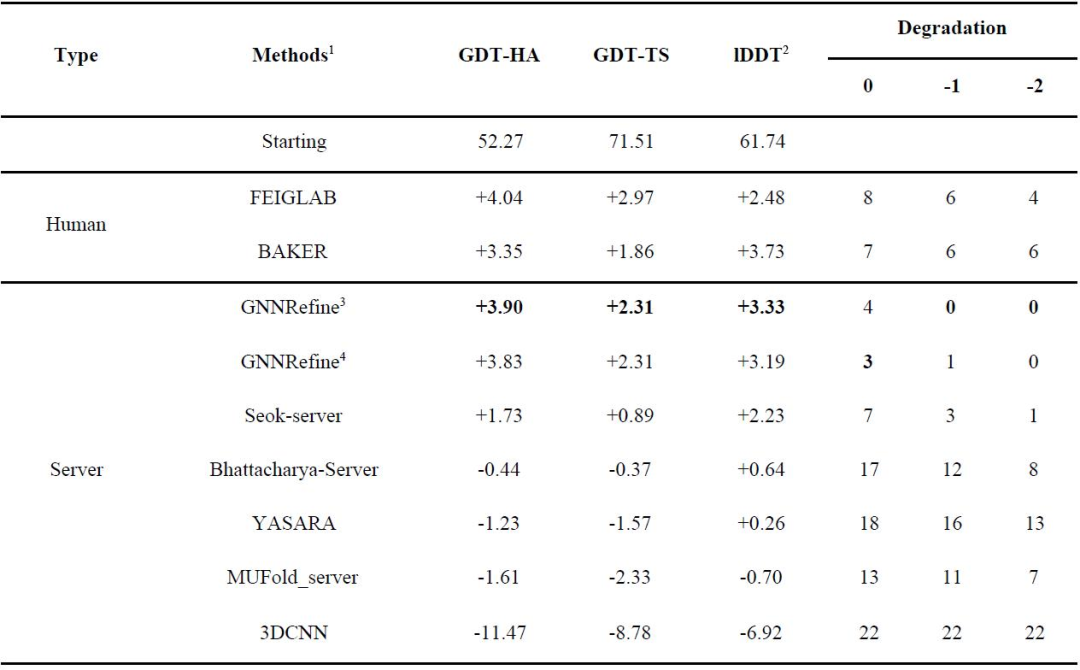
表1. CASP13优化目标上的性能。表格来源于Nat. Comput. Sci.
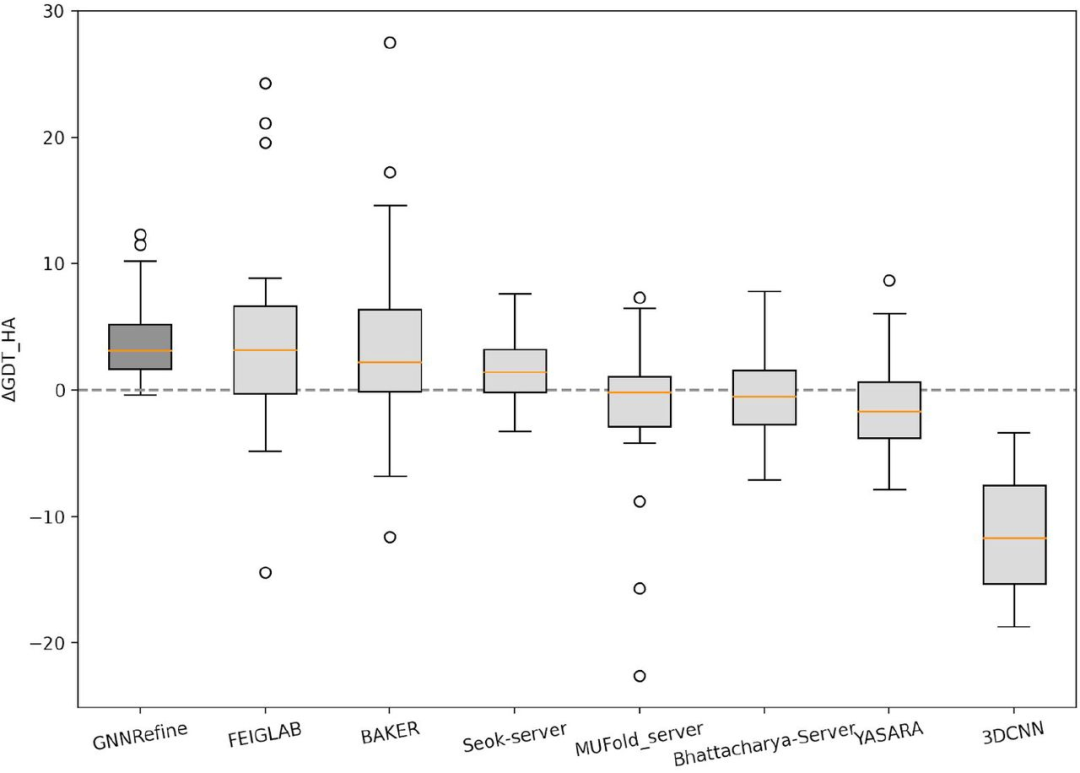
图2. ΔGDT-HA值在CASP13优化目标上分布的Box图。图片来源于Nat. Comput. Sci.
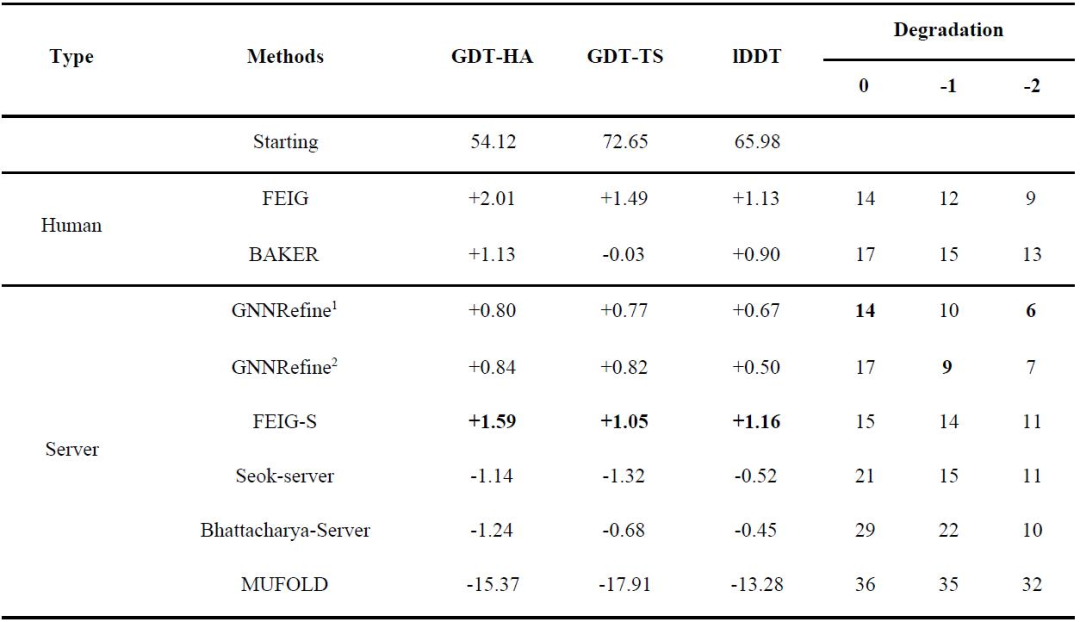
表2. 所有CASP14优化目标上的性能。表格来源于Nat. Comput. Sci.
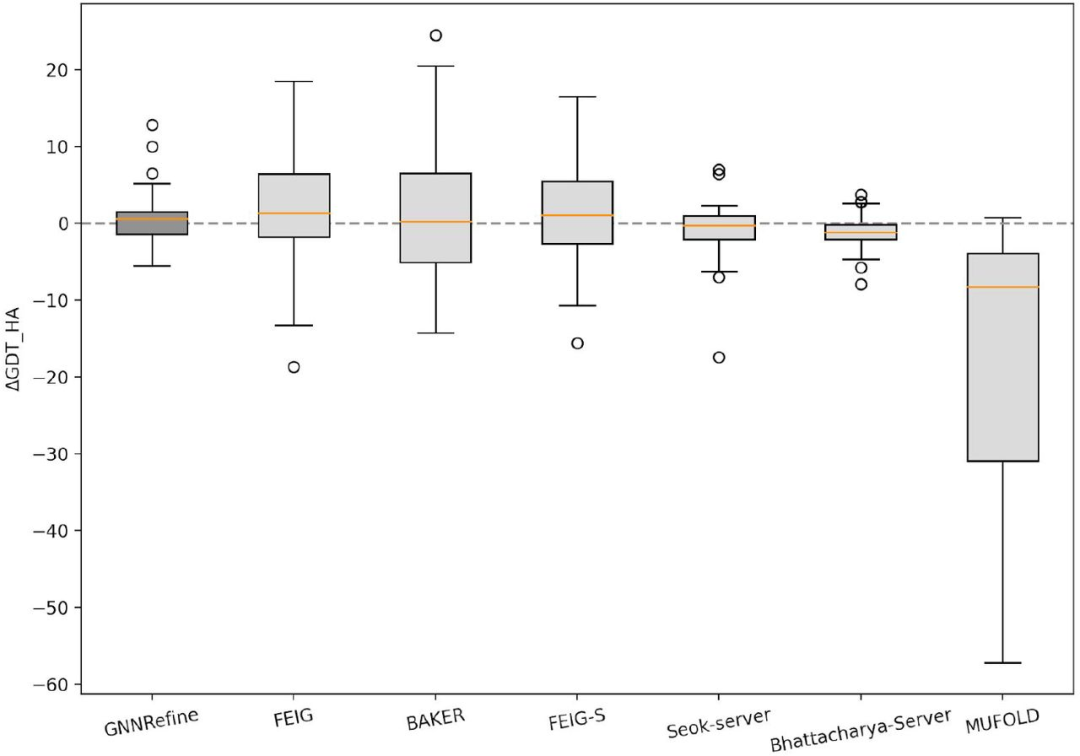
图3. ΔGDT-HA值在CASP14优化靶点上分布的Box图。图片来源于Nat. Comput. Sci.
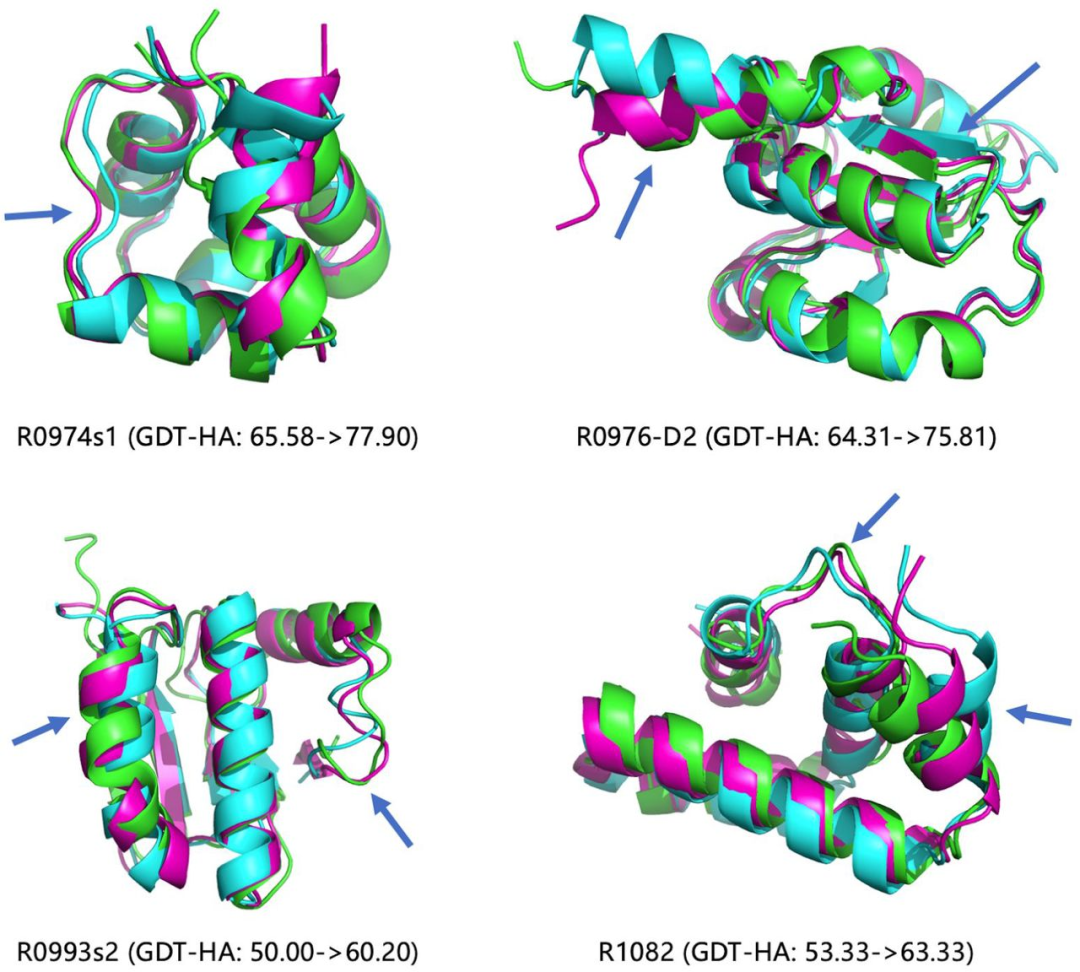
图4. GNNRefine对CASP13的R0974s1、R0976-D2和R0993s2以及CASP14的R1082目标的成功优化范例。图片来源于Nat. Comput. Sci.
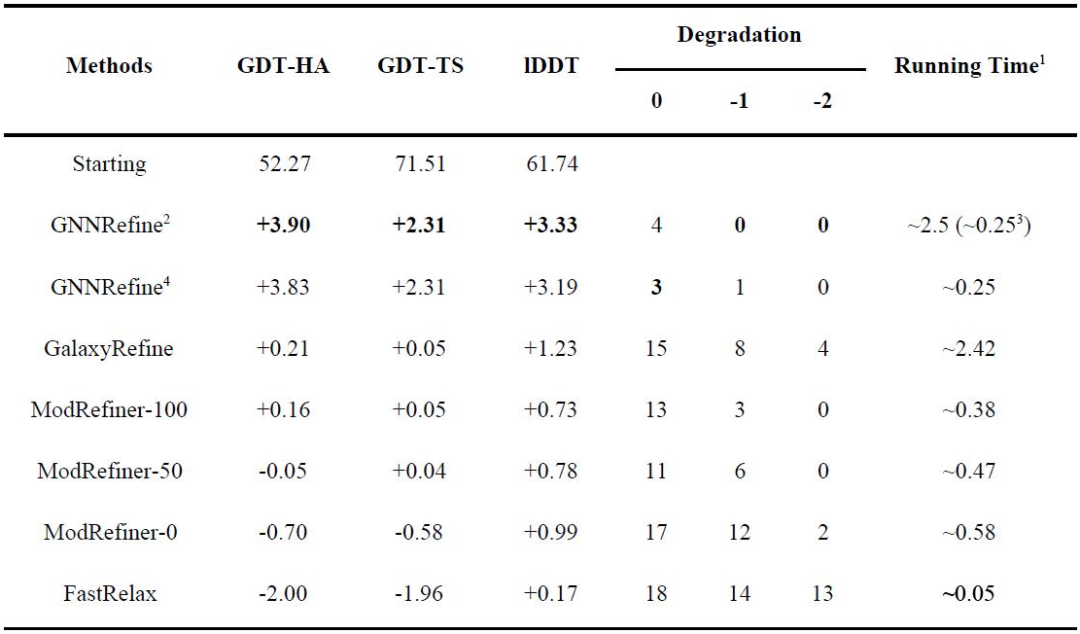
表3. 在CASP13优化目标上独立软件的性能。表格来源于Nat. Comput. Sci.
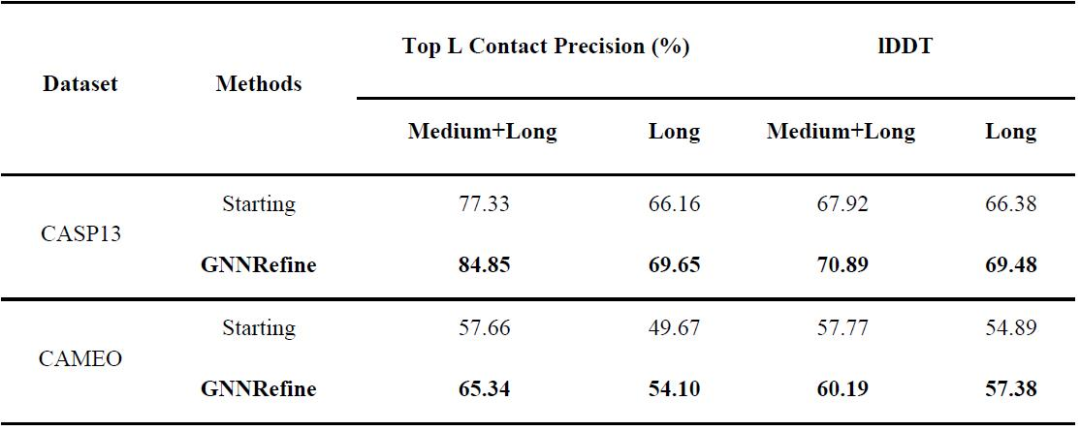
表4. 预测距离与初始模型中距离的比较。表格来源于Nat. Comput. Sci.
本文作者提出了一种新的蛋白质模型优化方法GNNRefine。GNNRefine利用图神经网络(GNN)从初始模型预测残差距离分布,然后将预测的距离信息输入PyRosetta FastRelax建立优化模型。由于只使用有限的构象抽样,GNNRefine可以非常快速地改进模型。研究表明,即使从一个初始模型中生成5个优化模型(约耗时15分钟),GNNRefine也可以提高模型质量,几乎和生成50个优化模型一样好,而且GNNRefine可以比一些使用广泛的构象抽样的方法更好。此外,当构象采样受限时,GNNRefine在蛋白质模型优化方面比ResNet好得多,因为GNN可以比ResNet更好地预测初始模型优化后的距离。
数据下载
In-house data: http://raptorx.uchicago.edu/download/
DeepAccNet data: https://github.com/hiranumn/DeepAccNet
CASP13 and CASP14 models for refinement: https://predictioncenter.org/
CAMEO models: https://www.cameo3d.org/modeling/
参考文献
Xiaoyang Jing, Jinbo Xu, Fast and Effective Protein Model Refinement Using Deep Graph Neural Networks, Nature Computational Science, 2021, ASAP. DOI: 10.1038/s43588-021-00098-9.