
DD-GUI 是英属哥伦比亚大学团队联合NVIDIA开发的深度学习和对接的图形用户界面,名为“深度对接”。DD-GUI 能直观且便捷地设置大规模虚拟筛选,并提供方便的工具,记录药物发现项目的进度和分析结果。
背景介绍
筛选数十亿种化合物的商业化合物库是发现新药的重要手段。然而,传统的虚拟筛选方法面临资源和时间的瓶颈。深度学习 (DL)可以用少量的分子对接成本评估数十亿种化合物,大大加速超大型化学库的虚拟筛选。
本文介绍的深度对接(DD)平台用迭代训练深度神经网络,丢弃预测对接分数较低的分子,将需要对接的分子数量减少到总数1%。DD 项目结果是一个分子子集,它比原始库小几十倍,但包含大多数得分最高的分子。因此,与传统方法相比,DD 的计算量可以在合理的时间内执行上十亿规模的大规模虚拟筛选,无需昂贵的高性能计算系统。例如,DD 用小于 400 个 CPU 和 40 个 GPU 在一周内针对 SARS-CoV-2 主要蛋白酶筛选整个 ZINC15 数据库(约 13.6 亿个分子),从中确定了新型强效的抑制剂。作者创建了一个图形用户界面 (DD-GUI),可以自动化地设置和运行 DD。DD-GUI 进一步降低了大规模虚筛进入门槛,药物发现研究人员只需单击几下即可对接数十亿个分子。
工具安装地址
https://github.com/jamesgleave/DeepDockingGUI
主要内容
安装 GUI
GUI 是本地托管的 Web 应用程序,用于实现与对接的集群通信。服务器通过 SSH 连接到集群,通过本地客户端服务器架构将信息传递给用户。要启动应用程序,用户需要使用 Node.js 启动服务器,然后在浏览器中打开它(类似于启动 Jupyter 笔记本)。
安装程序会设置本地 Conda 环境和运行服务器所需的所有 Node.js 模块。接着,它将请求访问具有 GPU 的 SLURM 调度集群,并安装所有必要的脚本和模块,以便在该集群上运行 DD 项目(除了必须预先安装的 AutoDock-GPU 对接程序)。具体的预准备和安装命令见github项目主页。
GUI的组成
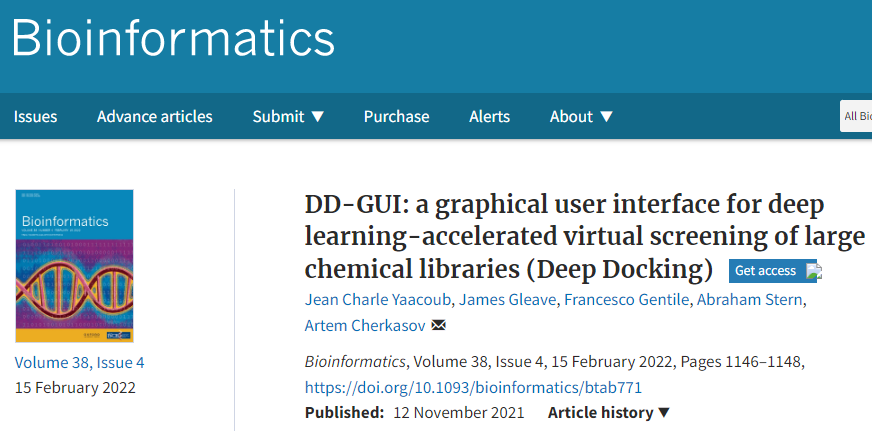
GUI 包含四个选项卡(图 1):Start a Run、Progress、Models和Top Scoring。
Start a Run 选项卡允许用户创建、修改、加载和运行项目。Progress选项卡预估当前迭代和整个运行的完成百分比,展示DD的进度,并提供当前阶段的时间估计。Progress选项卡还显示一个图表,报告估计的剩余分子数(根据模型的性能估计),以及每次迭代结束时剩余的实际分子数(库的子集),评估模型的普遍性。Models选项卡包含有关在每次迭代中训练的神经网络模型的信息。它显示在特定模型的训练期间获得的所有指标以及所有模型的平均值。它还有一个查看模型架构按钮,可在弹出窗口中显示所选模型的架构。最后,Top Scoring 选项卡显示了在模型预测的前一千个分子中最常见的骨架。用户还可以下载前一千个分子的 SMILES,分析模型优先考虑的化学结构,可视化每个分子的骨架。

图1. DD-GUI组成概览。图片来源:Bioinformatics
设置和运行筛选项目
DD-GUI需要预处理过的 SMILES 的化学库,并预先计算分子Morgan指纹以及受体对接口袋格点文件。软件提供了morgan_fp.py计算分子的指纹,以及 ZINC20 数据库。此外,还提供了 prepare_receptor.py 工具来计算 AutoDock 程序所需的受体文件。
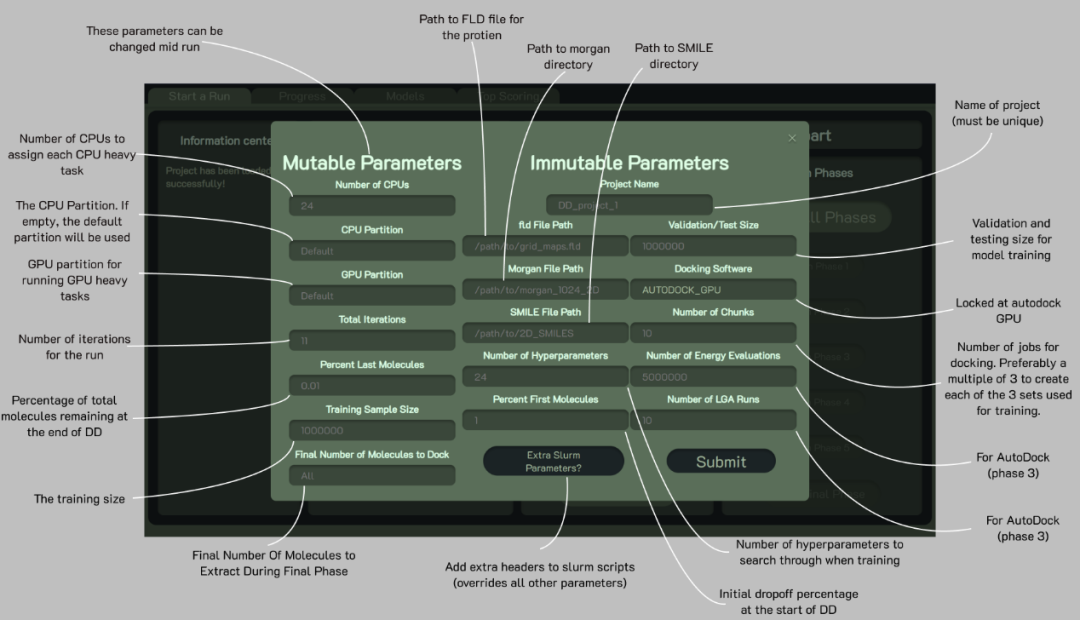
图2. 新建项目时的页面与参数解释。图片来源:Bioinformatics
GUI 会自动运行DD所有阶段并执行许多预定义的迭代,也允许定制特定的 SLURM 系统。随机采样开始后,该程序利用 OpenBabel 程序产生分子的低能构象,并保存为pdbqt格式。最后使用 AutoDock-GPU对接分子。训练结果随时受到监督并可视化到界面中。
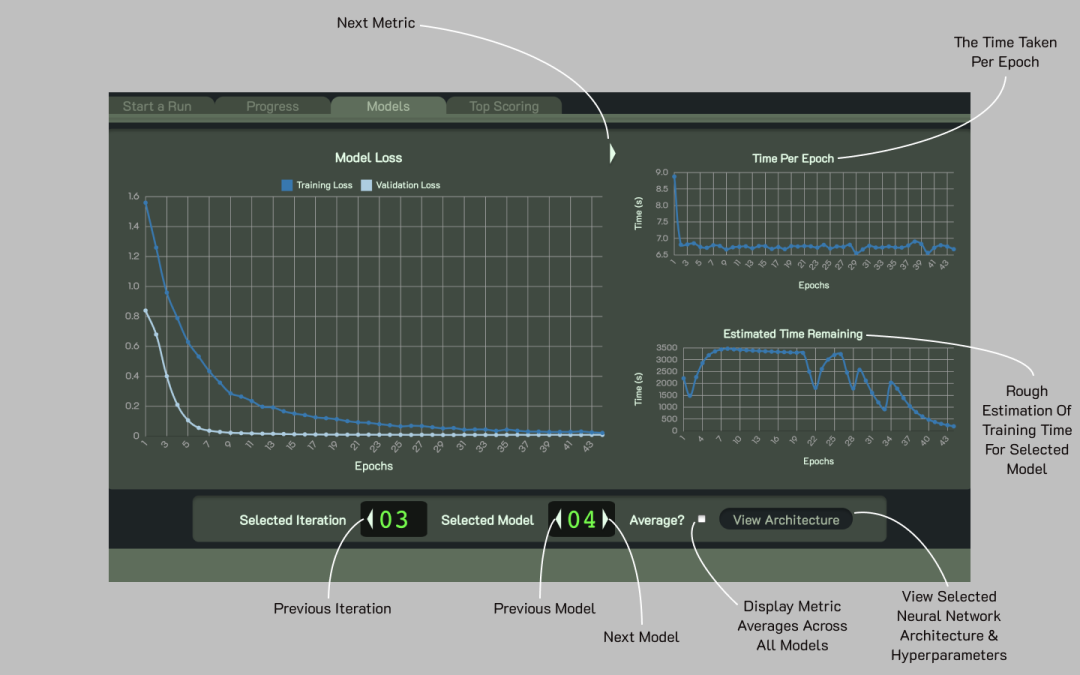
图3. 模型训练相关指标可视化页面。图片来源:Bioinformatics
计算结束后,软件会自动提取合格分子的 SMILES 结构,供进一步对接或后处理步骤。
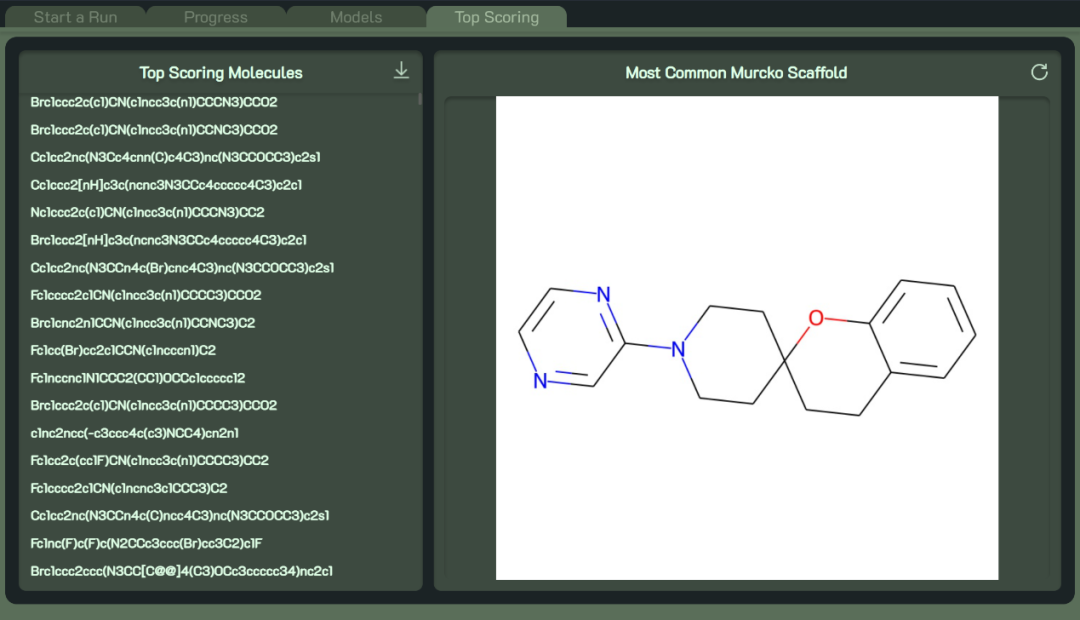
图4. 模型预测的Top1000分子的SMILES及结构。图片来源:Bioinformatics
结论总结
商业库的分子数量的空前增加,大规模虚拟筛选逐渐成为热点。DD-GUI 通过一系列开源程序来筛选大规模化合物库,且无需大量资源。该程序的用户友好性体现在为普通用户提供一个直观的平台,不需要用户了解底层机器学习模块知识。DD-GUI 将有助于计算机辅助药物发现的平民化,并鼓励更多学科的研究人员探索化学空间。
参考文献
Jean Charle Yaacoub, James Gleave, Francesco Gentile, Abraham Stern and Artem Cherkasov. DD-GUI: a graphical user interface for deep learning-accelerated virtual screening of large chemical
libraries (Deep Docking), Bioinformatics, 38(4), 2022, 1146–1148.