

通过关注肽和蛋白质的相互作用,首次从计算角度对肽结合位点进行的全面的形态学和能量学研究。
背景介绍
肽-蛋白相互作用与许多细胞和代谢系统的信号转导和调节网络有关,并与人类疾病如癌症和神经退行性疾病的发生密切相关。在过去的几十年里,人们对肽-蛋白界面的结构表征的兴趣对理解这些分子间的相互作用起到了关键作用,有助于合理设计出新的候选治疗调节剂。
然而,全面了解肽-蛋白质相互作用的分子细节仍然是一项艰巨的任务,这为计算方法的深入发展开辟了肥沃的土壤。目前,已有多个基于序列的肽-蛋白相互作用数据库(如ELM, DOMINO, PepBank)和从蛋白质数据库(如Pepx, PeptiDB,或PeptiSite)检索的肽蛋白复合物的三维晶体结构库。不同的开源预测工具,也可以在原子水平上研究发生在肽-蛋白界面上的相互作用。尽管如此,还需要进一步努力以获得基于能量的对肽与其靶标对应物之间发生的关键结合相互作用的了解。
网格分子相互作用场(GRID-MIFs)方法,已广泛应用于研究小分子和生物大分子上的能量有利结合位点。有趣的是,通过GRID力场产生的MIFs可以用来评估肽(靶标残基亦然)可建立的相互作用的类型、强度和方向。
主要内容
来自意大利巴里·阿尔多·莫罗大学的Orazio Nicolotti 和佩鲁贾大学的Gabriele Cruciani等人,通过聚焦多肽和蛋白质的立场,经由电脑模拟,得到了对肽结合位点的形态学和能量学的全面分析。研究者从PixelDB数据库(肽-蛋白复合物的高质量3D晶体结构的非冗余基准集合)开始,根据每个共结晶肽的性质对最具代表性的类别进行分类分析。为了揭示物理化学和结构上的因果关系,研究者从肽和靶蛋白两方面计算出了几种可解释的几何和能量描述符。最后,研究者分析了结合界面上最常见的肽-蛋白残基对,并基于GRID MIFs进行了广泛的能量分析。相关的研究成果以 “Getting Insights into Structural and Energetic Properties of Reciprocal Peptide–Protein Interactions” 为题发布在国际著名期刊Journal of Chemical information and Modeling上。
详细分析下载地址
https://github.com/KeatingLab/PixelDB
几何描述符
在尺寸和形状肽描述符中,考虑了长度、体积、粗糙度、球状度和灵活性等描述符,定义如下:
1. 用肽段长度来测定肽段的总残基数;
2. 体积表示水排开的体积(在Å3中),即被水可及表面包围的体积;
3. 凹凸度:表明肽表面存在分子褶皱,以体积/表面的比率表示;
4. 球形度来量化肽的球形度;
5. 弹性:分子的最大弹性。
考虑整个复杂的肽-蛋白,利用以下两个互反描述子来评价相互作用表面区域的埋深程度:
1. 溶剂暴露:表达为Å2的溶剂可接触到的靶标口袋的表面部分,不与蛋白质残基接触。
2. 埋藏体积:由蛋白腔内嵌套较多的原子的体积单位之和给出(即,距离低于预定阈值的原子等于9.0 Å)。
能量描述符
GRID以N1+和O-为探针,以对肽基产生的电荷、静电和极性相互作用的数量进行计算。用VolSurf+/BioGPS (FLAPsite tool)软件分别计算肽和靶蛋白的亲水性和疏水性区域(以及分子表面的相对比例)。疏水/亲水比率是疏水/亲水体积在整个分子表面的度量。
nr-PixelDB肽靶区多样性:形态和能量分布
首先,研究者对属于nr-PixelDB复合物的最具代表性的肽类进行了分类分析。根据先前所描述的关于肽的性质以及其已解决的晶体序列的百分比的标准,确定了四个主要类别。对于这四类肽,研究者分别计算了肽和靶点的二级结构以及几何和能量描述符的分布。四种肽类在所有分布(二级结构、几何和能量描述符)上均无显著差异。
肽-靶标界面的结构表征
首先,本文描述了在肽-蛋白质结合位点的二级结构基序的识别。如图1所示,热图量化了所有nr-PixelDB复合物在相互作用区域的不同二级结构基元的百分比,这些基元都是肽和相应的靶标对应物。每一行对应一个给定的nr-PixelDB复杂的交互位点,从黄色到蓝色的渐变颜色代码占特定结构图案的百分比不断增加。
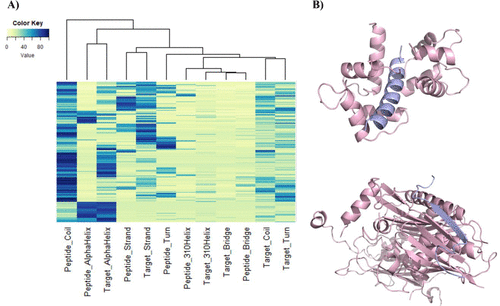
图1. nr-PixelDB复合物在相互作用区域的不同二级结构基元的百分比热图。图片来源:JCIM
从多肽的靶区角度看,α螺旋和β链是最常见的结构基元。对于大多数nr-PixelDB复合物,在相互作用位点发现 310 螺旋和桥式排列的百分比很低。
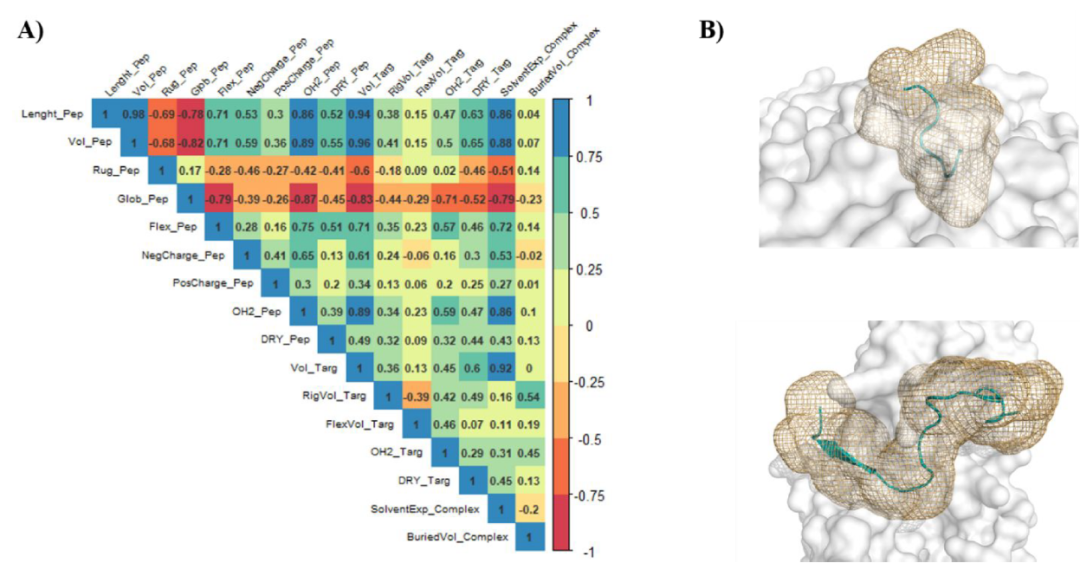
图2. 形态和能量描述符进行比较得到了肽-蛋白相互作用区域。图片来源:JCIM
将计算得到的形态和能量描述符进行比较以了解肽-蛋白相互作用区域(图2)。研究者构建了一个相关图来测量不同形态和能量特性之间的关系,这些特性包括肽、靶标和整个复合物。观察到肽的三维几何和能量特征与其形状之间的相关性为:高的球形度通常与较低的柔韧度(rGlob_Pep-Flex_Pep =−0.79)和较低的亲水性(rGlob_Pep-OH2_Pep =−0.87)肽相关。因此,具有扩展构象的多肽倾向于与蛋白质对应物上更宽的区域相互作用。在图2B中,为了完整起见,研究者报告了两个球形度高(PDB ID:2VDR)和低(PDB ID:2NUD)的多肽的例子。

图3. 肽靶三维分子描述符和二级基序的相关图。图片来源:JCIM
此外,研究者还分析了在相互作用位点上二级结构和三维描述符(几何和能量)之间的相互影响。图3显示了一个关联图,该关联图结合了二级结构图案的信息和从肽和靶标点导出的几何和能量3D-GRID描述符。具体而言,只有在肽或靶标中具有特定基元的nr-PixelDB复合物被用于推导相关性,其大小在括号中表示为考虑的nr-PixelDB复合物相对于nr-PixelDB数据库总大小的百分比(n = 423)。
首先,肽的圆度和粗糙度拓扑与其三维折叠成正比,如图3中的绿点所示。多肽以 310 个螺旋和桥基序的比例较高,呈球状排列。在线圈图案中占普遍比例的肽片段在其表面显示出更多的褶皱。
残差对统计分析与3D-GRID能量分布
第一步基于欧几里德原子间距离,研究者计算了发生在肽与蛋白质靶标之间的两组相互残基对的疏水性和极性相互作用。如图4左侧所示,根据其极性和疏水性质,考虑了用于评估疏水相互作用的减少数量的残基(Ala, Cys, Gly, Ile, Leu, Met, Phe, Pro, Trp, Val)。如图4右侧所示,研究者对所有20个氨基酸进行了研究,以检测极性对相互作用,主要是供体/受体的氢键。
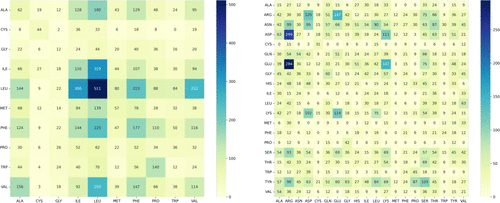
图4. 肽靶结合位点上的疏水(左手边)和极性(右手边)相互作用的相互残基矩阵。图片来源:JCIM
肽-靶界面上电荷相对的残基之间的静电相互作用,在极性对相互作用中起着特别重要的作用。事实上,得分最高的肽-靶对是GluTarget-ArgPeptide、AspTarget-ArgPeptide、ArgTarget-GluPeptide和GluTarget-LysPeptide,其相互作用对数分别为294、249、147和147。值得注意的是,除了侧链外,还包括主氢键的供体和受体,从而建立了极性相互作用,如图5所示。
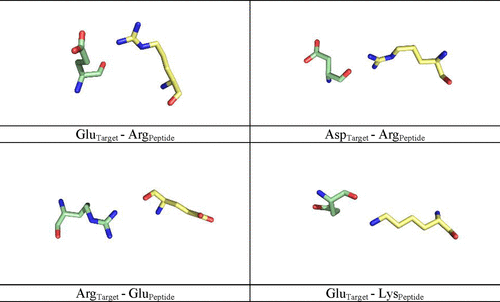
图5. 最常见的靶标-肽极性对的典型例子,通过招募氢键的主供体和受体建立。图片来源:JCIM
研究者利用CRY、N1和O三种GRID探针,在GRID力场中生成MIFs,利用BioGPS算法分别计算了亲脂性、氢键受体和供体相互作用。给定MIF的宽度取决于所涉及的相互作用基团的能量。MIF越大,在该区域找到互补的概率越高,如果残基是疏水的,则为疏水基团;如果残基是含氢键供体或受体基团的,则为供体部分。
图6中,研究者总结了多肽和相互作用结合位点靶点上的所有残基MIFs的能量分布(以kcal/mol表示)。
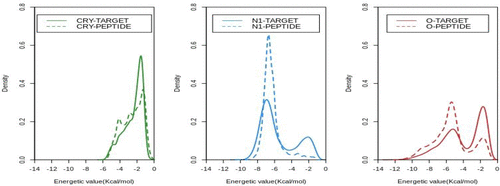
图 6. 分别用于研究亲脂性、氢键受体和供体相互作用的CRY、N1和O探针获得的多肽和相互作用结合位点上的所有残基的3D-GRID能量分布。图片来源:JCIM
值得注意的是,由CRY探针产生的两条曲线(绿线)的重叠表明,在疏水能量贡献方面,属于肽和靶标区域的残基具有相似的行为。相反,在极性相互作用中观察到不同的趋势。N1探针在能量水平上的峰值约为-7.0kcal/mol,且几乎完全没有弱相互作用(蓝虚线),表明肽残基在相互作用位点上具有很强的作为氢键受体的能力。相反,蛋白质靶点残基,既作为强H键受体又作为弱H键受体(连续的蓝线)。O探针对这两种残基(虚线和实线)都产生了强相互作用和弱相互作用(即在−2.0和−6.0 kcal/mol处出现峰值)。两个倒峰也被观察到,这表明与相应的靶标相比,肽残基作为氢键供体的能力更强。
蛋白质-蛋白质相互作用残基对的比较研究
接下来,研究者比较了nr-PixelDB与nr-PP-DB的配合物在相互作用位点上出现的给定残基对。考虑到肽(图7A和C)和靶(图7B和D)的观点,与蛋白质-蛋白质复合物相比,研究者计算出了疏水和极性对的百分比差异。
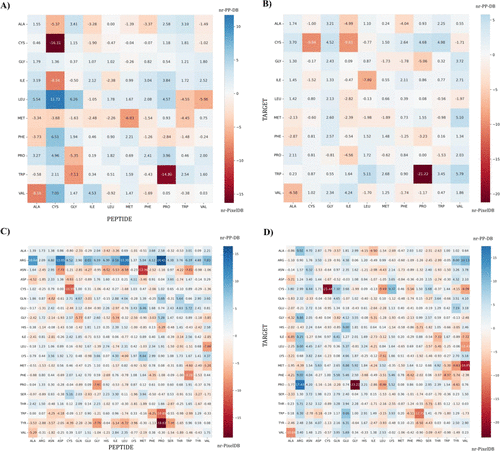
图 7. 属于nr-PixelDB的配合物与属于nr-PP-DB的配合物。图片来源:JCIM
如图所示,在nr-PixelD结合位点,ProPeptide与TrpTarget相互作用和CysPeptide与CysTarget相互作用时,疏水对接触的比例较高,分别为14.80%和16.31%(图7A,红色方块)。另一方面,胱氨酸和亮氨酸进行疏水相互作用,这种情况在蛋白质-蛋白质复合物中更为常见,其比例差异为11.72%(图7A,蓝色方框)。值得注意的是,TrpTarget被证实在肽-蛋白结合位点与ProPeptide特异性相互作用,其值达到21.22%。
有趣的是,脯氨酸的极性贡献对于属于nr-PixelDB的配合物来说是不同的,而对于肽的立场是nr-PP-DB(图7C)。结果表明,如果脯氨酸属于肽-蛋白复合物中的多肽,则更倾向于与靶标残基酪氨酸(18.63%)相互作用;此外,脯氨酸更倾向于与精氨酸在蛋白-蛋白极性对中相互作用(16.41%)。
如图7D所示,将属于nr-PixelDB的复合物的相互作用位点上的极性对与属于nr-PP-DB的复合物的相互作用位点上的极性对进行了比较,得出了相关结果。
结论总结
本文在基于肽和靶标蛋白的立场上,对肽腔进行了详细的拓扑和能量描述。这一分析证实了3D-GRID策略是一种有用的替代方法,可来表征肽设计中的氨基酸结构,以及与伴侣蛋白进行增强亲和力的相互作用。
参考文献
Daniela Trisciuzzi, Lydia Siragusa, Massimo Baroni, Ida Autiero, Orazio Nicolotti, and Gabriele Cruciani, Getting Insights into Structural and Energetic Properties of Reciprocal Peptide–Protein Interactions, Journal of Chemical Information and Modeling Article ASAP. DOI: 10.1021/acs.jcim.1c01343.