文章简介
作者设计了一种用于从头设计具有所需特性的分子的新型计算策略,称为ReLeaSE(结构演化的增强学习)。ReLeaSE在深度学习(DL)和强化学习(RL)方法的基础上,集成了两个深度神经网络(生成式和预测性),它们经过训练可用于生成新颖的指定属性分子库。ReLeaSE使用SMILES字符串作为分子的表示形式。生成模型通过stack-augmented RNN进行训练,以生成化学结构合理的SMILES字符串,并使用预测模型来预测生成的化合物相关属性。作者使用ReLeaSE方法来设计化合物库,这些化合物库均具有相关的特征,或偏向于结构复杂性,或偏向于特定物理特性,或者偏向于特定的生物活性。本文提出的方法可用于设计具有特定属性的化合物库。
计算方法
Deep RL(深度强化学习)
作者
Alexander Tropsha(JCIM副主编)
单位
Laboratory for Molecular Modeling, Division of Chemical Biology and Medicinal Chemistry, UNC Eshelman School of Pharmacy, University of North Carolina, Chapel Hill, NC 27599, USA
计算过程简述
作者使用Deep RL的方式来进行化学分子从头设计,使用ChEMBL数据库中的150万个分子进行模型训练,SMILES作为分子表现形式,以此训练G模型,使其学习生成SMILES字符串的规则,以便生成合理的SMILES字符串,后续又使用P模型来对化合物特性进行预测,并使用P模型来对G模型进行微调,以便产生符合特定属性的分子库。

图1.文章发表
图片来源 SCIENCE ADVANCES
ReLeaSE 工作流
ReLeaSE 由深度神经网络[generative (G) and predictive (P)]组成。训练过程分为两个阶段,第一阶段,使用监督学习算法对两个模型进行训练;第二阶段,使用RL方法对模型进行联合训练,RL可以优化目标属性。整个系统中,G模型用于产生符合具有化学可及性的新型分子,而P模型(用于预测新化合物的性质)则扮演评判的角色,对生成的分子打分,给与奖励或者惩罚。P模型会对G模型进行微调,目的是将奖励打分提到最高。
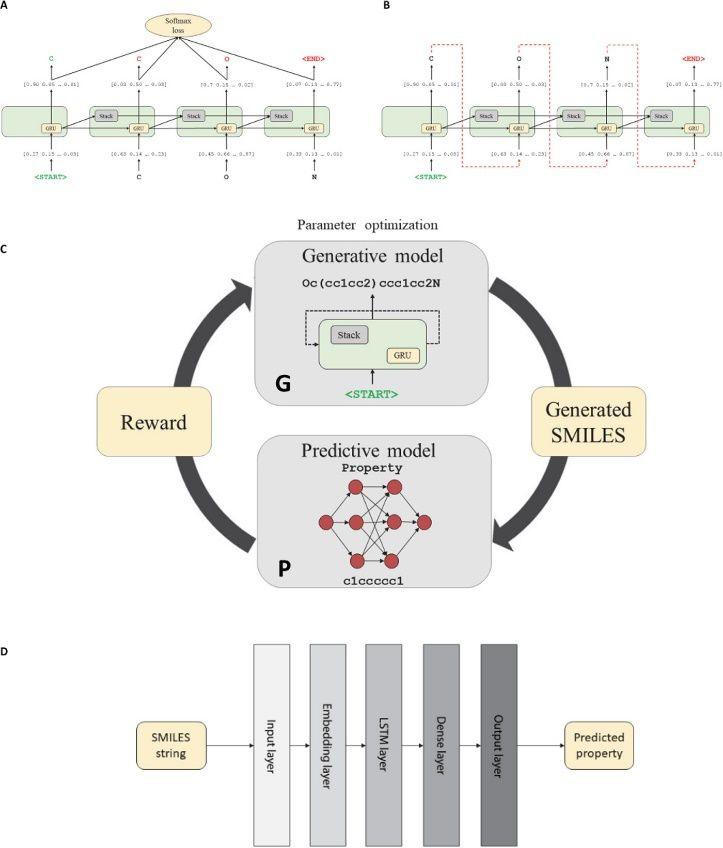
图2. Deep RL算法的工作流程
图片来源 SCIENCE ADVANCES
神经网络架构
G模型和P模型都组合到RL系统中。标准SMILES格式(字母和符号的集合)被用于编码。例如,阿司匹林被编码为[CC(= O)OC1 = CC = CC = C1C(= O)O]。模型G为generative RNN,作者这里使用一种特殊的stack-augmented RNN(Stack-RNN)。Stack-RNN的目标是学习如何生成标准的SMILES字符串,学习其中隐藏的规则。模型P被用于估算生成分子的物理,化学或生物学特性。该预测模型是一个深层神经网络,它由一个嵌入层,一个LSTM层和两个密集层组成。该网络旨在使用SMILES字符串作为输入向量来计算分子的相关属性。
生成具有新颖结构的化学分子
用ChEMBL数据库中的约150万个化学分子结构训练模型;训练的目的是为了学习生成SMILES字符串的规则。为了证明Stack-RNN的无偏性,作者生成了超过1M的化合物。从头开始分子设计方法一直以来存在的缺陷就是生成的化学分子无法合成,不具有化学可及性。为了解决这个问题,作者使用ChemAxon来对生成的化学结构进行有效性查验。结构证明,生成的结构中95%的结构都是有效的化学分子。随后,作者将1M个从头生成的分子与ChEMBL数据库中分子进行了比较,发现模型产生的分子数据比训练数据集的数据少了0.1%。
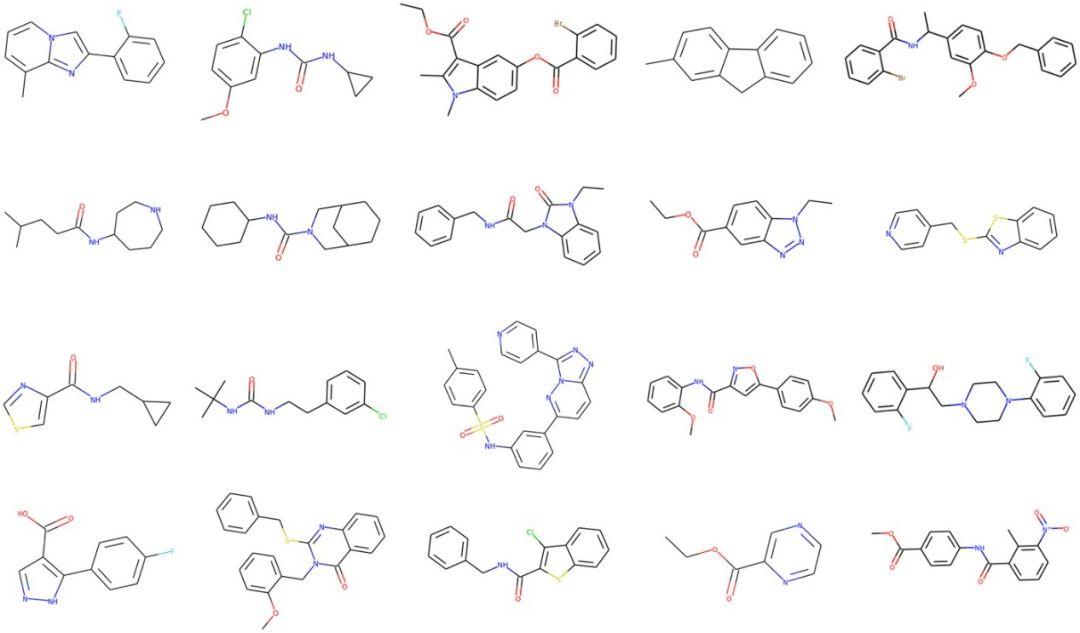
图3. 模型G产生的一部分分子
图片来源 SCIENCE ADVANCES
使用强化学习(RL)生成指定属性的分子库
为了探索强化学习(RL)在药物设计领域中的实用性,作者设计了案例用于研究,此案例用于设计具有指定属性的化合物库,属性如下:(i)重要的分子类药性物理特征,(ii)特定的生物活性,和( iii)化学复杂性。物理性质方面,作者选择了Tm和logP;生物活性方面,作者设计了新型化学表型的Janus蛋白激酶2(JAK2)的假想抑制剂;苯环的数量和取代基的数量(例如-OH,-NH2,-CH3-CN等)被用作奖励得分,以便用于设计复杂的化合物。作者使用基线(无RL)生成器和RL优化生成模型采样10000个分子,然后使用相应的预测模型计算了它们的性质,RL模型表现良好。
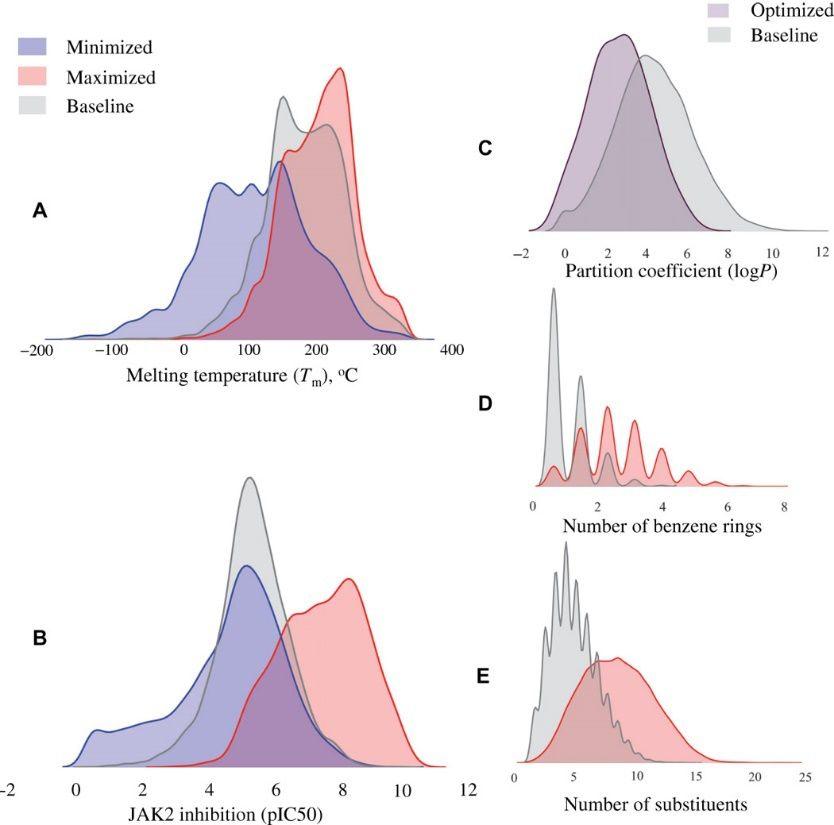
图4. RL优化与基线模型生成分子的属性分布
图片来源 SCIENCE ADVANCES
化学分子库可视化
为了了解G模型产生的分子在化学空间中的分布,作者使用t-SNE进行了降维处理,选择Tm,logP和JAK2的相关数据集,此类数据集是通过相应的优化模型生成。每个点对应一个分子,并根据其属性值进行着色。总的来说,这种方法提供了一种快速的方法,可以根据化学多样性和相应物化属性的变化来直观地观察化合物在化学空间中的分布。从结果中可以明显的观察到相似logP值的分子明显的聚集在一起。但对于Tm来说,没有明显的区分,高和低Tm分子混合在一起。而JAK2数据集中,可以观察到两个比较大的不重叠区域,大致对应于无效化合物(pIC50 <6)和有效化合物(pIC50≥6)的聚类。
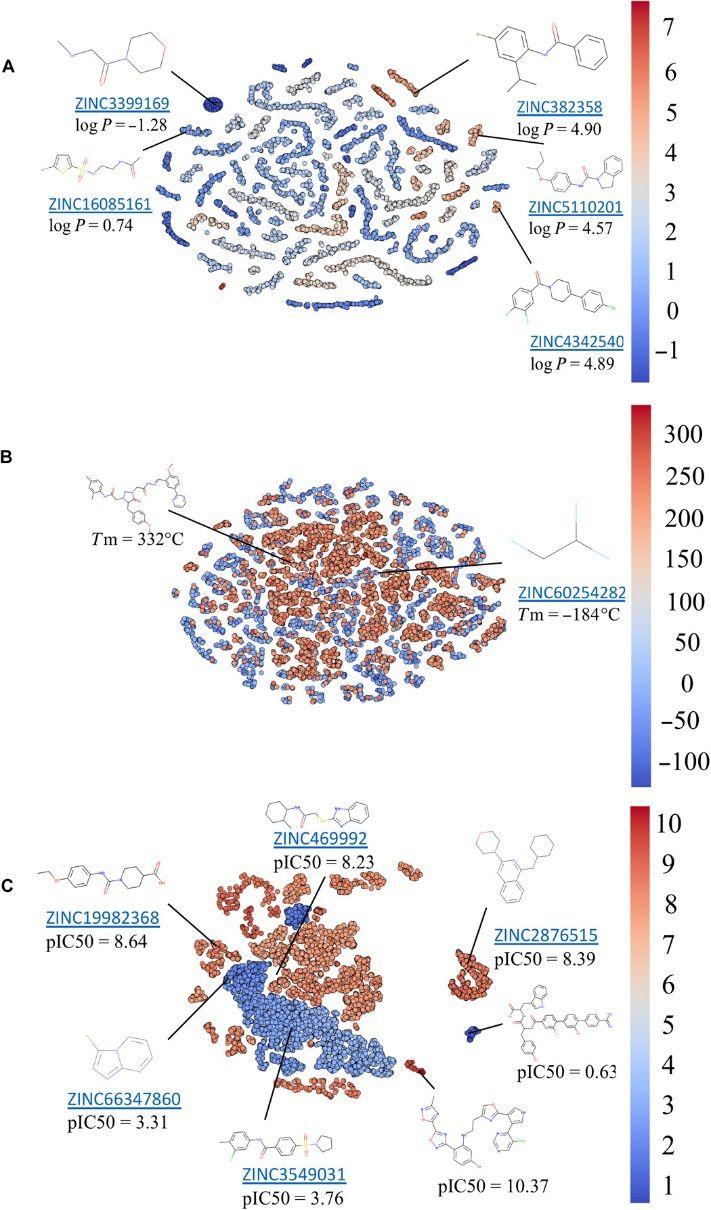
图5. 依据t-SNE对生成的分子进行聚类
图片来源 SCIENCE ADVANCES
总结讨论
本文作者设计出了ReLeaSE策略,此策略依赖于DL和RL方法,可用于生成指定属性的化合物库。作者进行了计算实验,验证了ReLeaSE策略在单任务方案中的效率。同时作者也在扩展该系统以便同时对多个目标多种属性进行优化。在药物研发领域,在功效,特异性,选择性,溶解性等方面对药物分子进行优化是必不可少的,作者未来将会着力于解决这一挑战。
参考文献:
Popova, M., O. Isayev, and A. Tropsha, Deep reinforcement learning for de novo drug design. Sci Adv, 2018. 4(7): p. eaap7885.