Align
align首先执行序列比对,然后进行结构叠加,进行多次迭代以便进行微调,在蛋白序列相似性大于30%的时候可以达到良好的效果。
用途
Align常常在结构生物学以及虚拟筛选中使用,当对不同的蛋白结构并对其进行比较时,我们就可以使用align比较蛋白结构,查看两者之间的差异,这个结构上的差异有一个量化的指标就是RMSD。它的概念和计算方式,都会在下面列出。目前,pymo是一个很流行的三维蛋白结构显示工具。本次的目的是,使用pymol对蛋白结构进行align,结果可以通过肉眼观测或者RMSD进行量化。
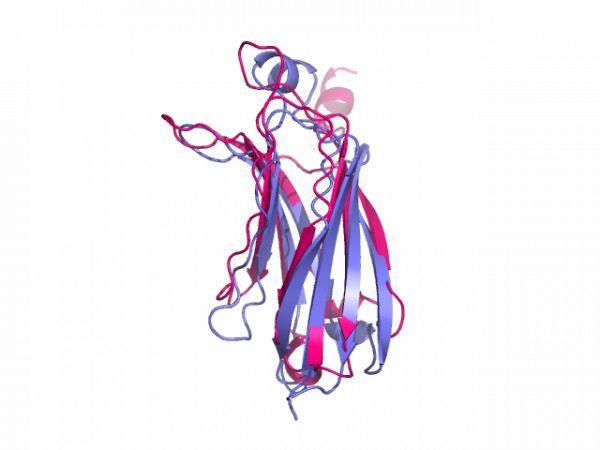
用法
align mobile, target [, cutoff [, cycles [, gap [, extend [, max_gap [, object [, matrix [, mobile_state [, target_state [, quiet [, max_skip [, transform [, reset ]]]]]]]]]]]]]
解释:
mobile =字符串:需要移动的对象名 target =字符串:目标的对象名 cutoff = 浮点数:截断值,默认2.0 cycles =整数:最大循环数,默认5 gap, extend, max_gap: 序列对比参数 object = 字符串:创建的一个比较对象名,默认无 matrix = 字符串: 序列比对的替换矩阵的文件名,默认BLOSUM62 mobile_state =整数: 移动选择的对象状态,默认全状态 target_state = 整数:目标选择的对象状态, 默认全状态 transform = 0/1: 是否做叠加,默认1
Alignment Objects
可以使用object = somename参数创建align对象。align对象可以:
(1)对比序列查看器
(2)对比原子对结果展示3D查看器中的线条
(3)可以保存到clustalw序列比对文件
RMSD
单位是埃 RMSD,root-mean-square deviation,也就是均方根偏差。原子位置的均方根偏差是叠加蛋白质的原子(通常是骨架原子)之间的平均距离的量度。注意,RMSD计算可以应用于其他非蛋白质分子,如小的有机分子。在球状蛋白质构象的研究中,通常在刚体进行完叠加后通过计算Cα原子坐标之间的RMSD来表征三维结构的相似性。
等式:
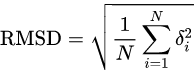
其中δi是原子i与参考原子之间的距离。计算时通常只计算为骨架重原子C,N,O和Cα或有时仅计算Cα原子。例如给出两套原子坐标,v和w,计算他们的RMSD就是,如下:
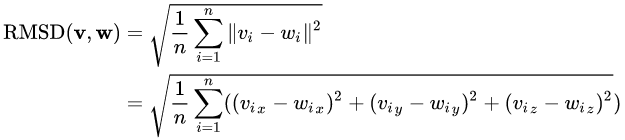
通常,RMSD用作两种或更多种蛋白质结构之间相似性的定量测量,通常越低越好。
Examples
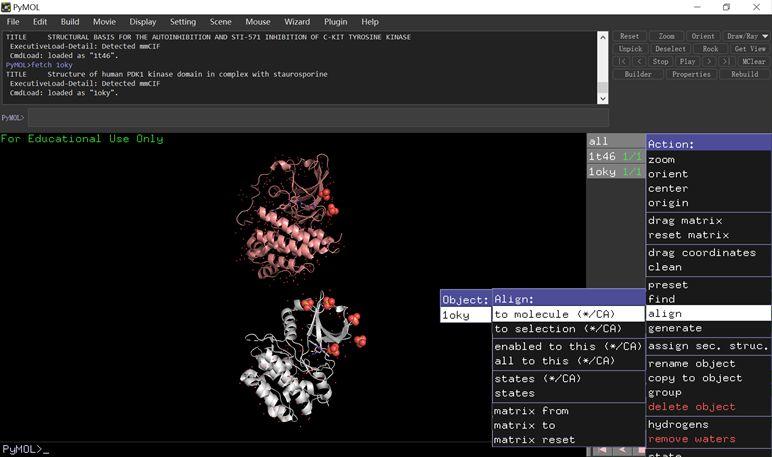
#获取蛋白
fetch 1oky 1t46
#比较这两个结构
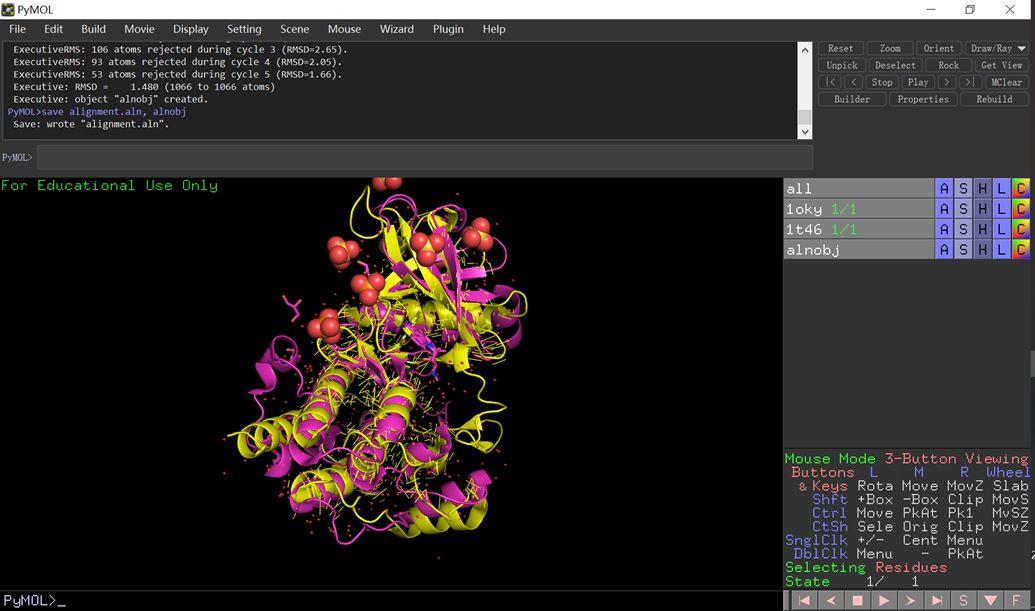
align 1oky, 1t46
# 比较这两个结构,比较比较对象命名为:alnobj,并且将alnobj保存为clustalw 文件,文件名为alignment.aln
align 1oky, 1t46, object=alnobj
save alignment.aln, alnobj
结果
alignment.aln文本内容

PS
附加鼠标操作流程:1t46右侧的A按钮–>align–>to molecule–>1oky
参考网页:https://pymolwiki.org/index.php/Align