前言

埃博拉病毒(EBOV)复制周期中的关键步骤会涉及到病毒糖蛋白2(GP2)的构象改变,从而促进宿主 – 病毒膜融合,释放病毒基因组的过程。这个蛋白的作用和HIV的GP41的作用机制类似。
这篇文章针对EBOV的GP2蛋白为靶点进行虚拟筛选,所选用的分子库包含大约170万个分子,使用传统的DOCK软件进行对接,从排名前列的化合物中,选出165种购买并进行生物活性检测,发现了4个良好的候选化合物,其IC50在3-26uM之间。
在随后的分子动力学模拟中,发现了结合相关的作用大多涉及到了7个高度保守的氨基酸残基,这个发现对未来EBOV的抑制剂研发有重要的指导作用。
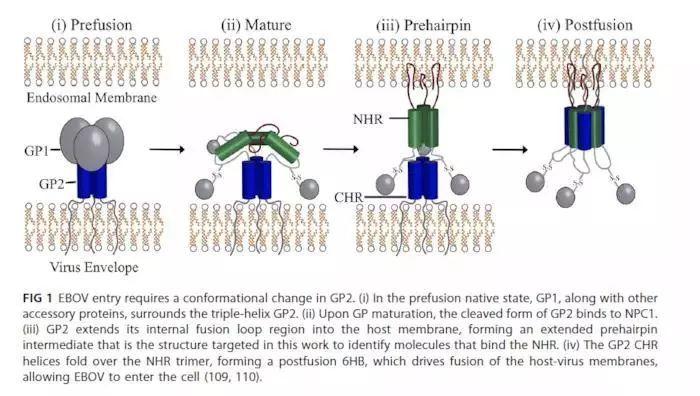
主要思路
大体思路很经典,第一步分子对接,然后选择得分靠前的化合物进行生物学实验验证,然后进行分子模拟解释实验现象。现在就来对这篇文章进行解析。

细节展示
蛋白靶标区域选择
蛋白:2EBO
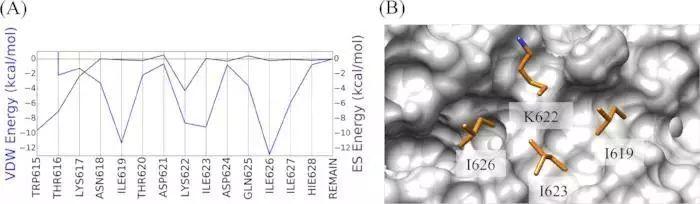
对GP2的结构进行分析,在CHR(599-632)区域分析出范德华力以及静电力贡献最大的残基,作为参考配体。
删除2EBO的C链残基A609到F630,以便使指定的对接靶点区域暴露,根据分子足迹分析,C链残基I619-I626与五螺旋区域有着最为紧密的联系,并将I619-I626作为参考配体。
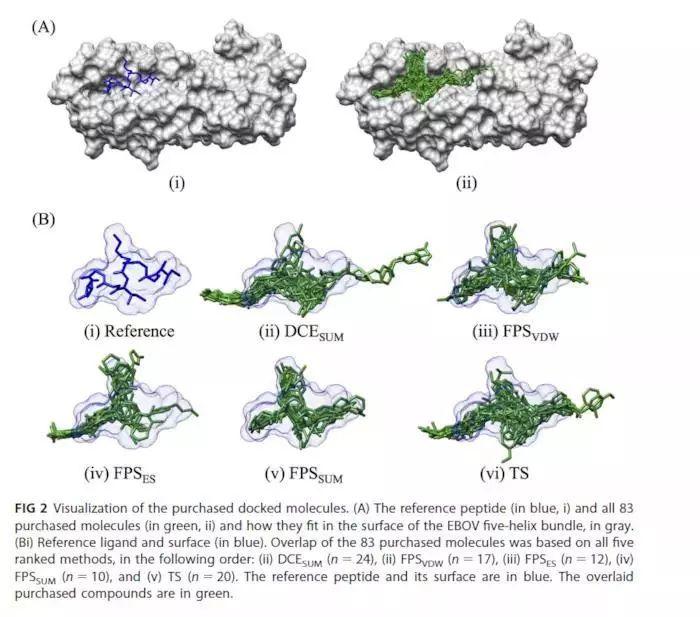
A图:GP41的CHR 多肽区域与GP2 五螺旋束之间的静电与范德华力的分子足迹(footprint profiles)分析
B图:靶点蛋白的关键性作用残基
02
分子库准备
来自于ZINC上面的170万个的一个类药化合物库。作者根据分子的旋转键将这个化合物库分为了42个小类,每个最多50000个分子。
03
对接
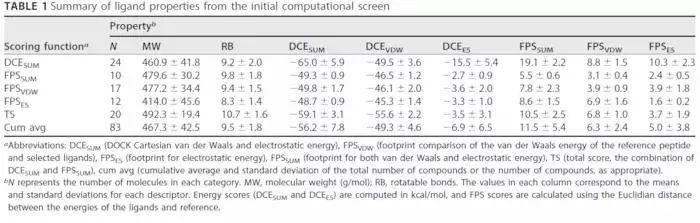
对接程序使用的是MPI版本的DOCK6.6,最好的结合模式被保存,然后使用DOCK Cartesian energy (DCE)进行微调 。排名前100000的化合物使用MOE计算其描述符,包括类药五原则,手性中心,以及logP。使用MACCS指纹对分子按照结构进行分类,每个类选中DCE得分最高的作为clusterhead。为了更加多样性的选择分子,clusterheads按照5种打分函数进行排序:
DCESUM ,FPSVDW, FPSES,FPSSUM, TS
04
生物学实验验证
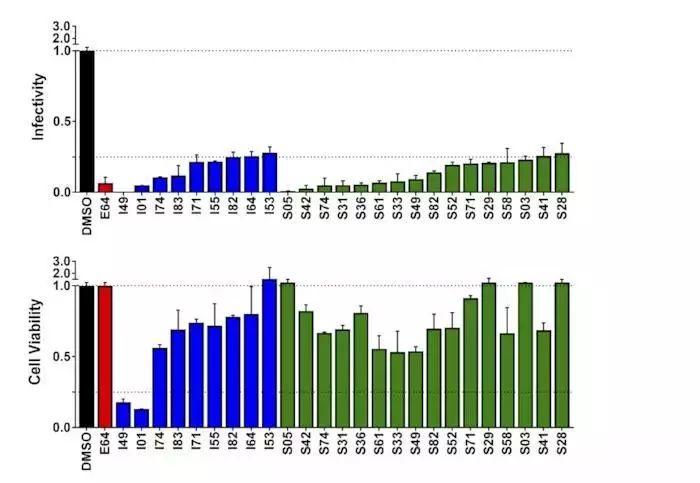
检测病毒感染率:295T细胞检测荧光素酶活检测细胞死亡率。从中选出9种化合物作为Hits。
05
分子动力学模拟
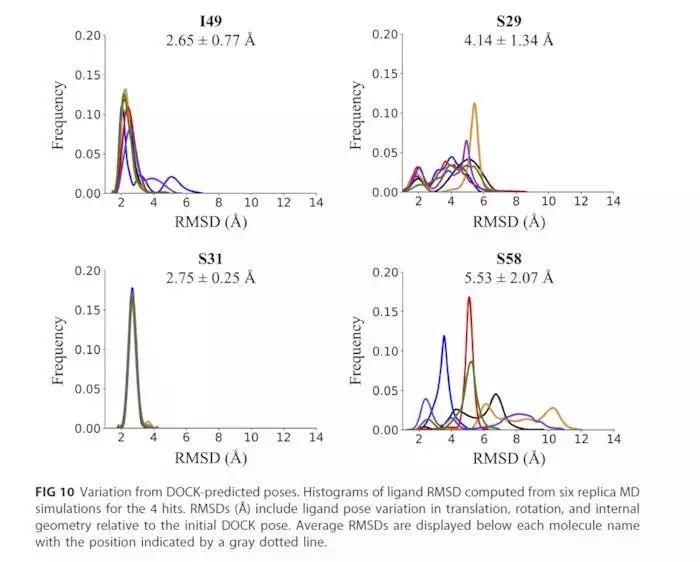
使用Amber程序进行分子动力学模拟,蛋白力场:ff14sb,水分子力场:TIP3P,配体力场:GAFF。进行20ns的分子动力学模拟,重复6次。使用RMSD(均方根偏差)对配体运动进行量化,RMSD考虑了相对于初始姿势的平移,旋转和几何构象的差异。
总结
一篇很经典的虚拟筛选文章,分子对接使用DOCK,对接后处理手法的目的是要尽可能的拿到多样性的分子结构,这其中涉及到一些化学信息学相关的计算,随后进行生物实验验证,得到好的Hits之后,进行动力学模拟,其结果被用于分析残基与配体之间的相互作用力。
亮点
在靶点区域的确认、打分函数以及动力学模拟结果分析都用到了Molecular Footprint这个技术。
