

TP-DB是从PDB中提取170万个螺旋结构并编译而成的一个治疗肽设计数据库,允许查询化合物复合物模式以促进识别螺旋结构的序列基序。
背景介绍
蛋白质的二级结构赋予了其一种特殊的机械性能,特别是α-螺旋,可以介导各种类型的分子功能,包括细胞膜穿透、亚细胞定位、分子内/分子间变构,特殊螺旋DNA框架、以及蛋白质-蛋白质相互作用等。
功能肽的系统设计具有良好的治疗应用前景。现有的数据库如蛋白质二级结构数据库(PSS)不再服务于社区,而蛋白质二级结构字典(DSSP)则在提供蛋白质三级结构时才对二级结构进行注释。因此,作者从PDB中提取了约170万个螺旋,并将其编译成一个数据库(治疗肽设计数据库:TP-DB)。本文展示了该数据库的三个应用:识别已知的纯化标签特异性抗体、设计一种新的抗菌肽(AMP),以及提示螺旋肽阻滞剂的点突变。
TP-DB链接
http://dyn.life.nthu.edu.tw/design/
TP-DB界面
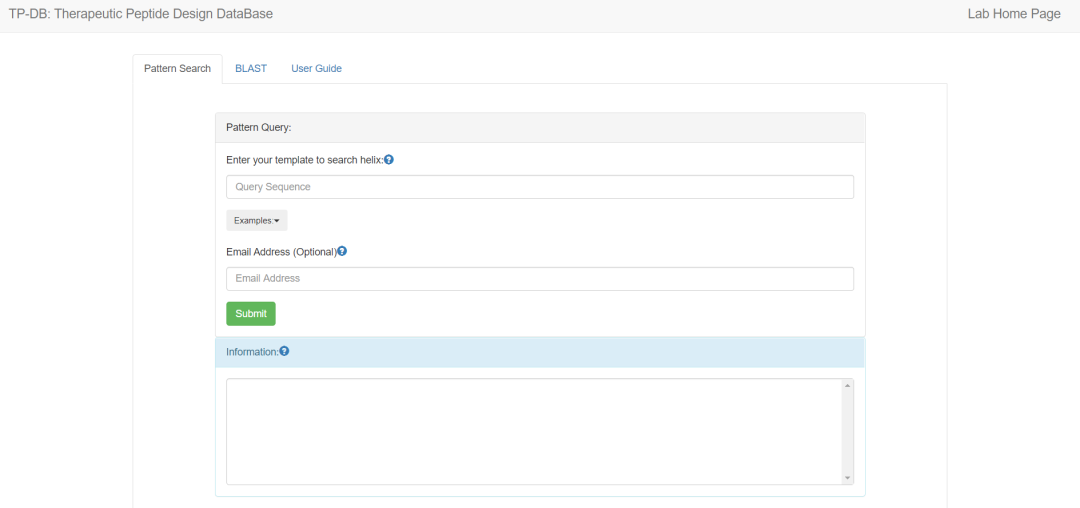
数据库特性和统计数据
TP目前由1,676,117个螺旋肽组成,从130,000个以上的实验解析的蛋白质结构中提取。作者设计了两个指标来评估和优选多肽——HP和接触分数。更高的HP和/或更低的接触分数表明肽的单独形式具有很好的螺旋性。标准化的HP,HPNA定义为HPa.a./NAa.a.,其中HPa.a.为给定的氨基酸类型的HP,NAa.a.是氨基酸的天然丰度(表1)。HPa.a.和NAa.a.为概率,HPa.a.定义为TP-DB中某种氨基酸类型的分数,NAa.a.则是ExPASy工具“ProtScale”工具中同种类型氨基酸的分数。相关肽的上述数据的对数值总结于表1。
第二个度量是平均接触分数,定义为氨基酸的总数,由它们的Cα原子表示。在其亲本蛋白中,一个螺旋的接触数较低,这表明它在独立地保持螺旋形式的可能性较高。只有高度接触的螺旋在如此高的接触稳定时才能形成。
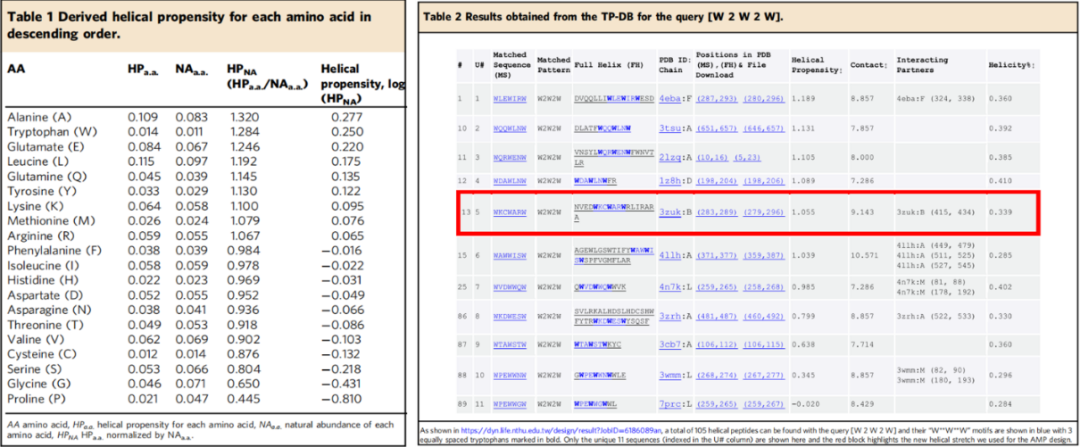
表1. HPa.a.及NAa.a相关数据结果。表格来源:Nat. Commun.
TP-DB搜索引擎的构建
作者建立了一个不考虑序列同源性的基于模式的搜索引擎,并针对TP-DB的数据结构进行了定制。匹配特定模式的螺旋肽首先在数据库中进行索引。一种模式是“Y***G**K”的形式,其中“*”属于20种氨基酸中的任何一种,酪氨酸(Y)、甘氨酸(G)和赖氨酸(K)被称为该模式的“锚定残基”。模式的位置由PDB ID、链ID和模式在该链中开始的位置进行索引。组成模式为[A1mA2nA3],其中A1、A2和A3是氨基酸的单字母编码,两者之间由整数m和n表示的间隔, m和n的范围是从0到4。“Y3G2K”或其搜索模式[Y 3G 2K]表示“Y”是G上游的(3+1)残基,K上游的(2+1)。一个键与标识符(值)配对,指的是在所有存储的多肽段中都可以找到这个键的位置(图1c,e)。这些值存储在一个数组中,是肽标识符和每个肽中某个键的所有起始位置(图1c)。这些键-值对组成了TP-DB数据库(图1),可以通过简单的模式[A1mA2nA3]或由这些简单模式组合的模式来搜索(图1f)。
要查询数据库,用户需要指定至少三个锚定残基和分隔它们的间隙,例如“ADE”或“K***VA”可以分别用 [A 0 D 0 E]和[K 4 V 0A]搜索(图1d)。对于更复杂的查询,如“A/Y 3 G 2 K 3 H 4 K”(组氨酸:H),它首先被分解为两个子查询“A 3 G 2 K 3 H 4 K”和“Y 3 G 2 K 3 H 4 K”的组合(图1f)。每个子查询中的五个锚定残基被视为两个连接的三锚定键。在返回结果之前,进一步解析肽序列中共定位的匹配模式以考虑重叠区域。TP-DB目前有两个搜索引擎来搜索索引的肽段,一个是通过上面描述的模式搜索,另一个是通过BLAST。

图1. TP-DB的肽段索引和查询。图片来源:Nat. Commun.
Anti-FLAG抗体被重新用于病原体诊断
为了解释搜索引擎在没有考虑残基进化相似性时发现模式的效用,作者需要确定匹配查询模块的序列是否只能通过其TP-DB工具查询到。作者演示了一个纯化标签(FLAG-tag)及其抗体(M2抗体)可被重新用于检测人类病原体的诊断试剂盒的案例。已知FLAG-tag有一个序列DYKDDDDK,其模式D-Y-K-x-x-[DE]被实验证实为主要的亲和决定基序。
当对TP-DB查询“D0Y0K2D/E”模式时,可以识别出完整的93个序列和唯一的19个序列。同时使用PHI-BLAST搜索NCBI的非冗余蛋白序列(nr),不能查到任何E-value<100的序列。另外,在19个TP-DB鉴定的序列中,在幽门螺杆菌菌株26695中发现了含有序列“DYKYLE”的HP-NAP。如果anti-FLAG M2抗体能够识别这个随机选择的致病蛋白,则可以使用相同的概念,将任何已知的抗体和抗原识别基序的原始用途作为其替代用途。如预期一样,通过酶联免疫吸附试验(ELISA;图2a)和免疫印迹法(WB,图2b)分析,发现anti-FLAG M2抗体确实能够识别HP-NAP。进一步分析显示,M2抗体与HP-NAP上含有Arg77到Glu116残基的多肽片段结合,该多肽片段中存在抗原识别基序DYKYLE。
因此,纯化试剂盒的抗体(以下简称M2抗体)可以重新用于检测人类病原体的诊断试剂盒,文中为幽门螺杆菌检测,通过ELISA识别天然HP-NAP或通过WB识别17 kDa条带处的NAP单体。
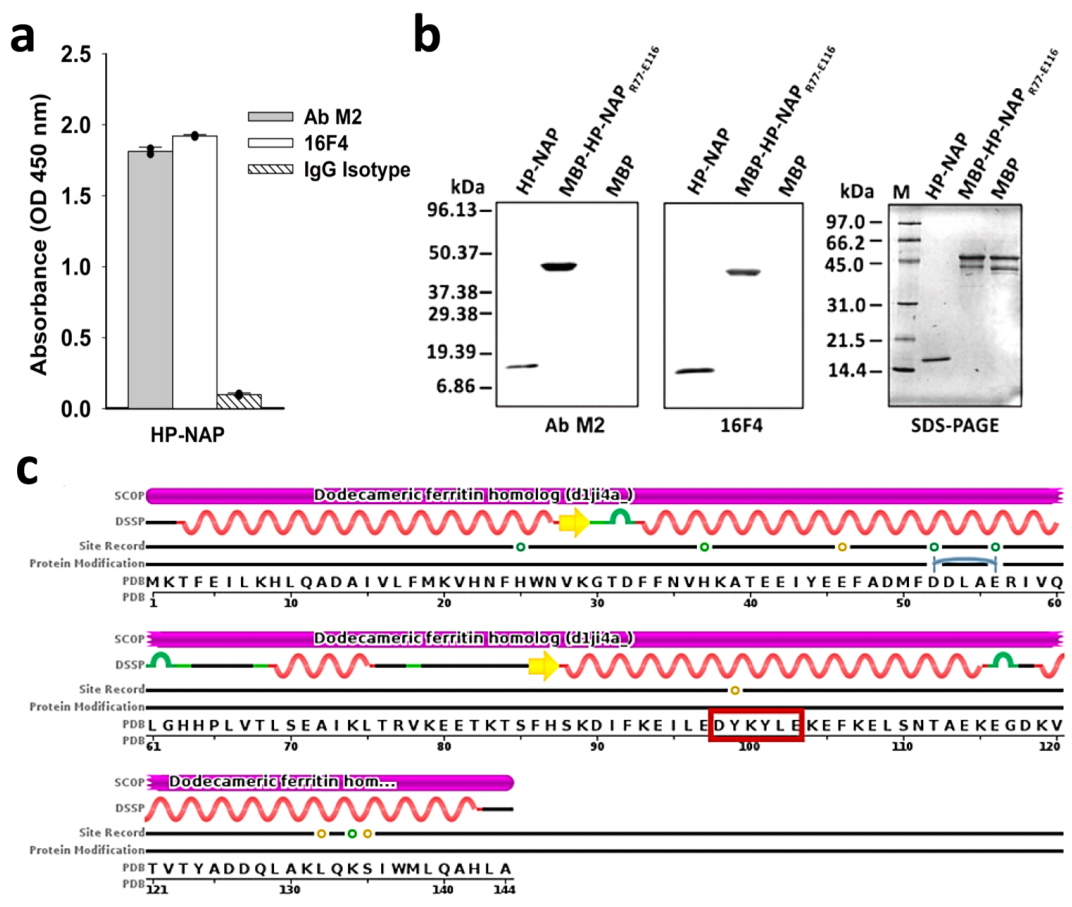
图2. 用抗FLAG M2抗体检测HP-NAP。图片来源:Nat. Commun.
α-螺旋AMP设计
泛耐药细菌对人类健康有重大威胁。以细菌脂质膜为目标的AMP的进化速度比细菌蛋白受体要慢得多,似乎是一种很有前途的抗生素替代品。作者使用TP-DB进行了相关的研究。首先通过MD模拟详细研究了之前报道的两个AMP螺旋(W3_p1和W3_p2)的插入过程,以了解每个残基在细菌膜插入过程中的物理化学作用,肽穿透脂膜越深,它所能具有的杀菌能力就越高。以W3_p1为例,将结果展示在图3。通过微秒模拟作者发现,这些AMP插入细菌膜,严重依赖于与两个氨基酸等量间隔的三个色氨酸锚点,是插入的必要条件。
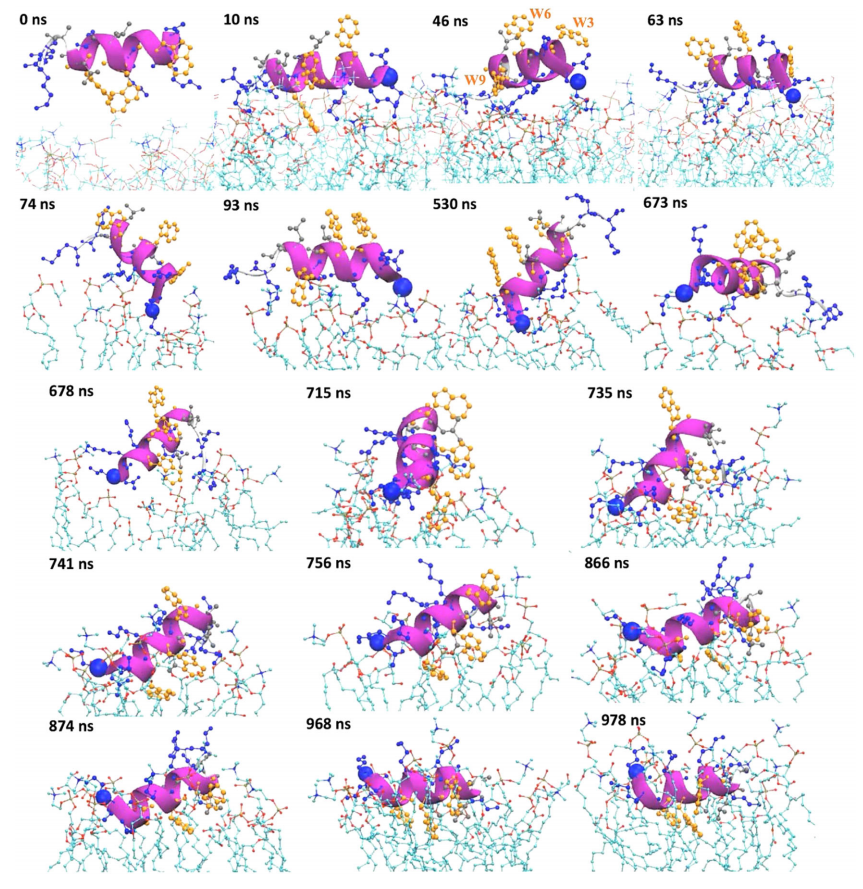
图3. 1μs MD模拟揭示了W3_p1插入细菌膜的机制细节。图片来源:Nat. Commun.
作者从已知的螺旋AFP/AMP作为设计模板开始进行AMP设计。基于现有的模板和关键的锚定残基,使用TP-DB取代了其中三个等距色氨酸(W3、W6和W9)两侧的残基,以找到功能有利的取代基,并确保具有所需的二级结构。使用TP-DB中的输入查询模式“W**W**W**W”或[W2W2W]作为搜索查询。
肽“WKCWARWRL”(C:半胱氨酸)具有最多的正电荷残基和相对较高的HP,被列为首选。该肽来自结核分枝杆菌锌依赖金属蛋白酶-1,是一种不与膜结合的可溶性蛋白。添加了两侧的双赖氨酸残基以扩展这个基序成为一个成熟的AMP候选。新设计的 AMP命名为 W3_db5,然后由MD模拟进行分析并合成用于后续杀菌实验。
对比模板的MD模拟结果发现,在1 μs 模拟结束时,W3_db5 只有一个 Trp (W3) 插入细菌膜。随后,作者检测了其在能抑制90%的细菌生长时的最小抑菌浓度(MIC90)。新设计的AMP W3_db5的抗真菌MIC90为3.75 μg/mL,比W3_p1 和W3_p2的效果好两倍以上,但在抑制细菌的能力上,MIC90弱于W3_p1 和W3_p2,为30 μg/mL,在细菌抑制能力上的结果与MD模拟结果一致。
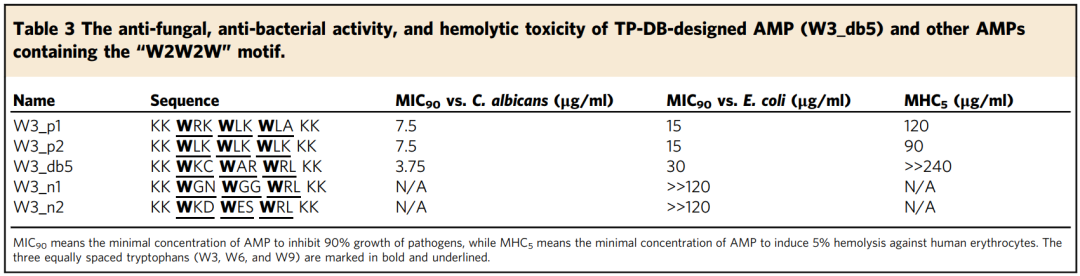
表2. AMP体外药效结果。表格来源:Nat. Commun.
尽管新设计的W3_db5与其母本的抗菌活性相似,但是其毒性更低。W3_db5的溶血性至少比W3_p1和W3_p2低20倍。表2中的AMP的最小溶血浓度MHC5测试也展现出了类似的结果。
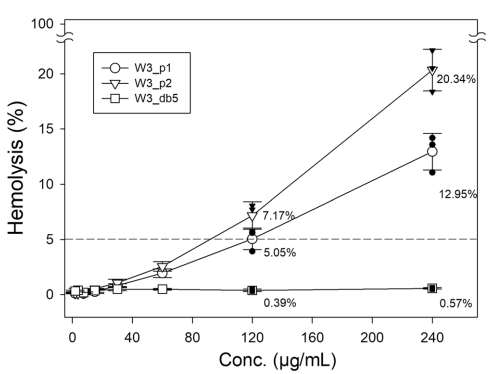
图4. W3_p1、W3_p2和W3_db5的溶血百分比与AMP浓度的关系。图片来源:Nat. Commun.
含有W3_db5中这种强效拉伸“WKC WAR WRL”的原蛋白(结核分枝杆菌锌依赖金属蛋白酶-1)不是跨膜或膜结合蛋白;螺旋拉伸的位置接近但不完全暴露在水-蛋白界面。因此,TP-DB能将结构解析的元素组合在一起,匹配所需的模式,为了治疗方案的理性设计,这里则展示了一种距离其起源有6个点突变新的AMP。从设计的角度来看,这 6 个点突变似乎并未明显改变所需的插入热力学,W3_db5仍然可以穿透肽聚糖,到达脂质-水界面,保持两亲性和螺旋状,并最终插入细胞膜。这很难通过突变实验或 MD 模拟在没有TP-DB的指导下实现。
随后作者发现从TP-DB中提取的序列在单独存在时比其他方法(比如PHI-BLAST)获得的相似序列更具螺旋性。圆二色谱数据显示(图5),搜索获得的三个AMP中,W3_db5螺旋度最高,W3_n1最低,且W3_n1的螺旋度最接近Tat(一种流行的细胞穿透肽,具有随机螺旋结构)。
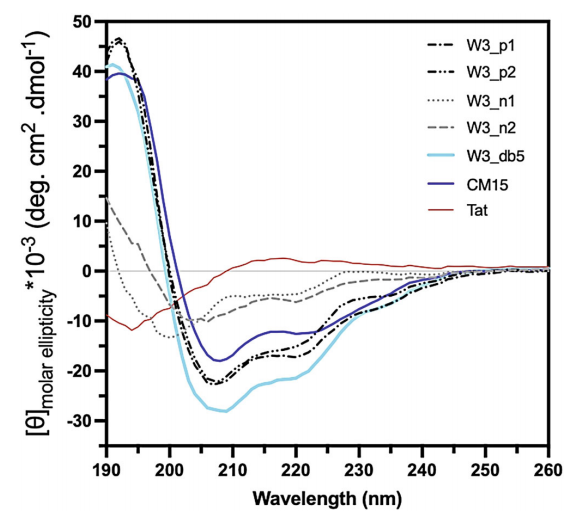
图5. 变化范围为190~260nm的CD谱。图片来源:Nat. Commun.
设计针对致瘤性PPI的肽抑制剂
表1中的统计数据显示,丙氨酸仍是点突变的安全选择,同时仍然保持螺旋的完整性。然而,使用基于模式的搜索引擎在TP-DB中的可及性,可以通过选择TP-DB建议的残基,在螺旋中进行点突变,以消除或者增强其余确定药物、多肽或者蛋白的相互作用,并且仍然保持螺旋的螺旋性,以及能在结构生物学家对一个酶及其同源底物进行共结晶的情况下,取消α-螺旋上残基的催化功能,同时保持其与某些底物或配体的结合亲和力。
总结
TP-DB搜索引擎首次让在引入突变以获得功能的同时确保所需的二级结构成为可能。本文演示了如何通过TP-DB设计一个新的螺旋AMP,且随后验证了其能抗真菌和细菌,并通过圆二色谱验证了其高螺旋度。除此之外,TP-DB还可用于设计螺旋-肽阻断剂,以防止致瘤性蛋白-蛋白相互作用。TP-DB的出现加速了功能肽设计的过程。
参考文献
Tsai CY, Salawu EO, Li H, Lin GY, Kuo TY, Voon L, Sharma A, Hu KD, Cheng YY, Sahoo S, Stuart L, Chen CW, Chang YY, Lu YL, Ke S, Ortiz CLD, Fang BS, Wu CC, Lan CY, Fu HW, Yang LW. Helical structure motifs made searchable for functional peptide design. Nat Commun. 2022 Jan 10;13(1):102. doi: 10.1038/s41467-021-27655-0. PMID: 35013238; PMCID: PMC8748493.