

一种基于深度学习的多肽-蛋白质相互作用(PepPIs)预测框架(CAMP),包括了多肽-蛋白质相互作用预测和相应的多肽结合残基识别。
背景介绍
多肽,通过与多种蛋白质相互作用参与了许多细胞过程,如程序性细胞死亡、基因表达调控和信号转导,在人类生理中发挥着重要作用。通过实验确定PepPIs通常是费时且昂贵的。为了解决这个问题,研究人员开发了一系列计算方法,以促进肽药物的发现。
基于序列和基于结构的方法,是预测蛋白质与配体相互作用的两种主流方法。基于序列的方法,主要利用原始序列信息对交互进行建模。但这种方法,通常需要已知的蛋白质-配体相互作用作为监督标记,蛋白质(或配体)的成对相似度评分作为输入特征,由于相似度计算的复杂性,这对于大规模数据通常是不切实际的。基于结构的方法,如分子对接,通过在原子水平上建模结构构象和预测结合亲和力,成功地解决了这个问题。目前,确定PepPIs有许多成熟的对接策略,根据输入结构信息的程度,大致可分为局部 (DynaRock和rossetta FlexPepDock)和全局(如PIPER-FlexPepDock和HPEPDOCK)对接方法。不幸的是,求解这样的三维结构,通常是耗时且昂贵的,更不用说由于能量函数的高计算复杂性而消耗的大量计算资源。
最近,蓬勃发展的深度学习技术,为蛋白质配体或蛋白质相互作用(PPI)的建模提供了可行的解决方案,用更少的计算资源就可以实现更高的准确性。尽管肽药物越来越被重视,最近几十年获批的肽疗法的数量也呈上升趋势,但只有少数工作提出了利用机器学习或深度学习方法来建模PepPIs。此外,对于解读PepPIs的潜在机制,现有的方法主要集中在蛋白质表面的肽结合残基的识别上,如基于序列的PepBind方法和基于结构的InterPep方法。遗憾的是,这些方法都有自身的局限性。
此外,现有大多数用于建模PepPIs的计算方法,都未能回答药理学家经常提出的一个重要问题——如何确定每个单独的肽残基对结合活性的贡献?因此,显然需要解决以下挑战:(1)准确有效地识别PepPIs,同时考虑多肽和蛋白质的信息;(2)对大数据集具有很强的泛化能力;(3)检测关键的肽结合残基,可提供有用的线索。
主要内容
为此,来自清华大学的赵诞&曾坚阳等研究者提出了CAMP,这是一个可同时预测PepPIs和识别结合残基以及肽序列的深度学习框架。综合评价表明,CAMP可以成功捕获多肽与蛋白质之间的二元相互作用,并识别出参与相互作用的多肽的结合残基。此外,CAMP在预测双肽-蛋白相互作用方面,优于其他主流方法。CAMP可以作为预测肽-蛋白相互作用和识别肽中重要结合残基的有用工具,有助于肽药物的发现过程。相关的研究成果以“A deep-learning framework for multi-level peptide-protein interaction prediction”为题发布在国际著名期刊Nature Communications上。
CAMO概述
首先,CAMP应用了以下5个步骤的多源数据管理和多级标签构建(图1a):
(1) 从RCSB PDB中提取肽蛋白复合物结构,从DrugBank中提取已知药物-靶点对;
(2) 利用蛋白质配体相互作用预测因子(protein ligand interaction predictor, PLIP),识别每个PDB复合物中肽与蛋白质之间的非共价相互作用,仅保留具有非共价相互作用的肽蛋白对作为阳性样本;
(3) 从PepBDB获得肽的结合残基标签(PepBDB是由RCSB PDB衍生的肽蛋白复合物的结构数据库);
(4) 基于肽和蛋白质的一级序列,生成肽和蛋白质的残基级结构和理化性质、内在无序倾向以及蛋白质的进化信息;
(5) 整合多级标签,即肽-蛋白对的二元相互作用标签和肽结合残基标签进行训练过程。
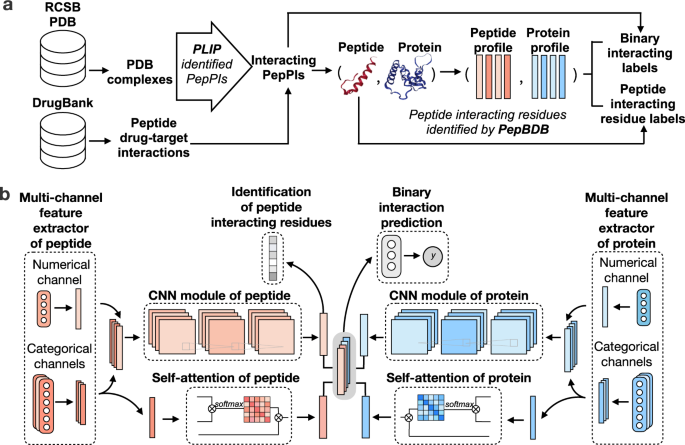
图 1. CAMP的工作流程和体系结构。图片来源于Nat. Commun.
CAMP的整体网络架构,如图1b所示。给定输入肽-蛋白对的特征轮廓,CAMP利用两个多通道特征提取器分别对它们进行处理。该数值通道,用于提取预定义的密集特征(即蛋白定位特异性评分矩阵(PSSM)和蛋白和肽序列中每个残基的内在无序趋势)。每个分类通道都包含一个自学习词嵌入层,该层采用输入肽或蛋白质的分类特征之一(即原始氨基酸、二级结构、极性和亲水性)。
接下来,CAMP利用两个卷积神经网络(CNN)模块分别提取多肽和蛋白质的隐藏语境特征。此外,CAMP采用自注意力机制来了解残基之间的长期依赖关系,以及蛋白质和多肽个体残基对最终相互作用预测的贡献。然后,CAMP结合所有提取的特征,使用三个完全连接层来预测给定的肽-蛋白对之间是否存在相互作用。CAMP取每个位置具有sigmoid激活功能的肽CNN模块的输出,预测每个肽残基是否与伴侣蛋白结合。
在二元相互作用预测中,CAMP优于基线方法
在这里,研究者比较了CAMP与其他最先进的基线方法的分类性能,包括基于相似度的矩阵分解方法(NRLMF)、基于深度学习的PPI预测模型(PIPR)和基于深度学习的CPI预测模型(DeepDTA)。
图2显示,CAMP的表现始终优于最先进的基线方法,在AUC和AUPR方面分别增加了10%和15%。此外还注意到,在“新肽设置”下的模型表现,似乎比在其他设置下的模型表现更好。这些测试结果表明,在所有交叉验证设置下,CAMP都能比基线方法获得更好、更健壮的性能。图2还显示了CAMP在某些聚类设置下生成的预测结果相对不同。
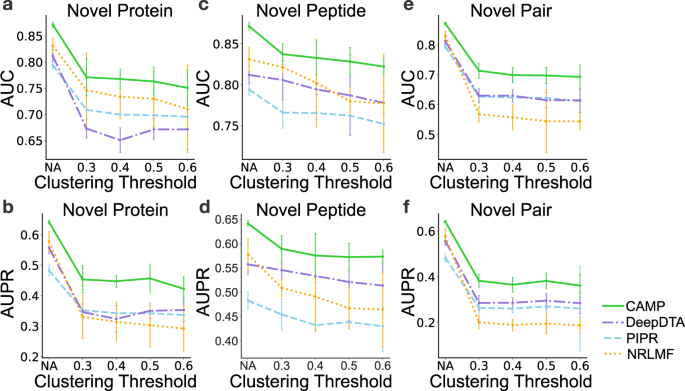
图2. 交叉验证三种设置下CAMP与基线模型的AUC和AUPR。图片来源于Nat. Commun.
通过表征肽结合残基的新见解
在CAMP中,研究者设计了一个监督预测模块,来识别肽序列中的结合残基。首先,研究者利用来自PepBDB的相互作用信息,构建了一组肽结合残基的合格标签;这是一个全面的结构数据库,包含了从RCSB PDB已知的相互作用肽蛋白复合物,以及有关氢键和疏水接触中肽结合残基的信息。
在这些监督信息的支持下,CAMP在使用随机分裂设置的五倍交叉验证程序进行肽结合残留鉴定时,获得的平均AUC为0.806,马修斯相关系数(MCC)为0.514(图3a, b)。
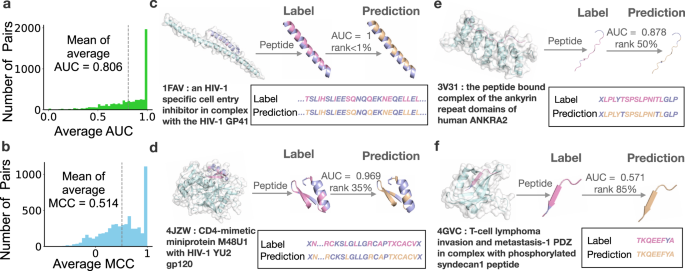
图3. 通过5倍交叉验证,评价CAMP在基准数据集上肽结合残基识别的性能。图片来源于Nat. Commun.
为了进一步证明CAMP在结合残基预测中的性能,研究者还选择了4个具有代表性的案例(预测肽结合残基的平均AUC评分分别为约1%、35%、50%和85%),并将预测残基与真实相互作用的残基进行了比较。
图3c显示了第一个案例,一个HIV-1特异性细胞进入抑制剂和HIV-1 GP41三聚体核心的复合物(PDB ID: 1FAV)。肽抑制剂有33个氨基酸,其中12个是结合残基。CAMP识别了所有这些结合残基,没有任何假阳性。
图3d显示了第二个案例,HIV-1 gp120包膜糖蛋白和CD4受体的复合物(PDB ID: 4JZW),在平均AUC方面排名前35%左右。该肽有28个氨基酸,其中13个是结合残基。研究者预测的结合残基覆盖了沿着肽序列的11个真结合残基,遗漏了2个真结合残基。
图3e显示了第三个案例,组蛋白去乙酰化酶和锚蛋白重复家族A蛋白的一个肽复合物(PDB ID: 3V31)。在研究者的预测中,这一对在AUC方面排名中位数,CAMP成功识别了11/13的真结合残基,其中有一个假阳性。
图3f显示了最后一个案例,T淋巴瘤侵袭和转移诱导蛋白和8个残基磷酸化的syndecan-1肽复合物(PDB ID: 4GVC),在研究者的预测中排名约85%,平均AUC为0.571。所有8个残基(包括1个假阳性)都被CAMP预测为结合残基。
总体来说,研究者的测试结果表明,CAMP能够准确预测结合残基,从而为进一步理解肽与伴侣蛋白的相互作用机制提供了可靠的证据。
CAMP在附加基准数据集上的通用性
为了证明CAMP对二元交互预测的鲁棒性,研究者评估了CAMP和基线模型对上述测试数据集的几个变化的正-负比率的性能。图4a和b显示CAMP在所有场景下取得了最好的结果,表明CAMP优于基线方法,具有相对稳健的性能。研究者还观察到,随着正负比从1:1下降到1:10,所有方法的AUC均略有增加。
研究者还评估了CAMP对肽结合残基识别的预测结果。研究者从PepBDB中获得了注释的肽序列结合残基。从测试数据集来看,总共有208种PepPIs具有这样的肽结合残基标签。图4c和d显示了CAMP能够维持其对上述附加数据集的预测能力。
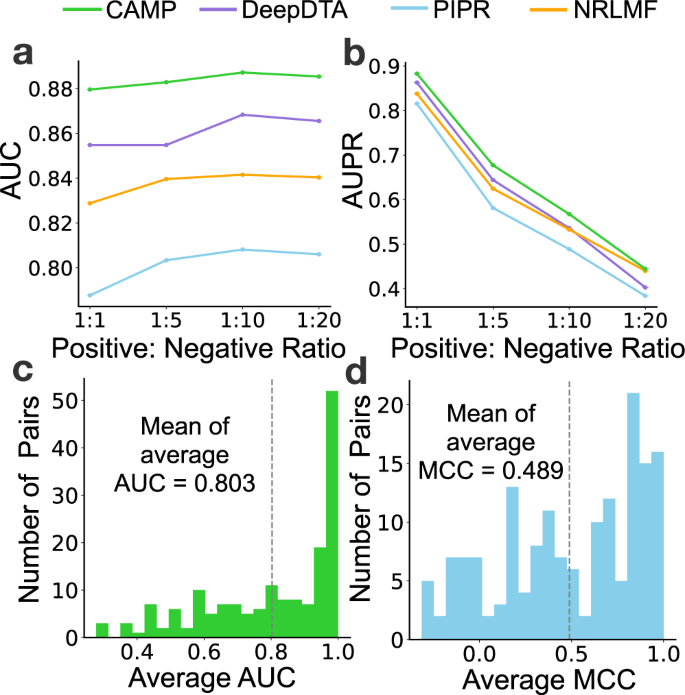
图4. CAMP具有稳健的性能,并在独立测试集上优于基线模型。图片来源于Nat. Commun.
CAMP在三个相关任务中的扩展应用
研究者进一步研究了CAMP在预测肽-PBD (protein binding domain)相互作用、结合亲和评价和肽虚拟筛选三个相关任务中的应用潜力。图5显示了CAMP在除PDZ外的所有家族中,都显著优于HSM-ID和HSM-D。
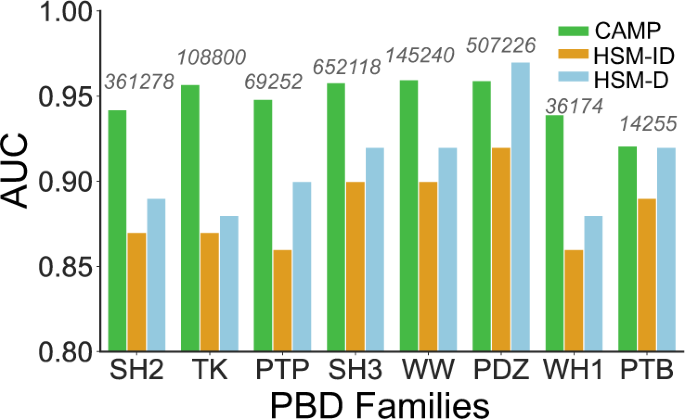
图5. CAMP、HSM-ID和HSM-D在8个系列中的模型表现。图片来源于Nat. Commun.
结论总结
本篇文章提出了CAMP,一个用于预测多肽-蛋白多水平相互作用的深度学习框架,包括二元相互作用预测和多肽结合残基预测。所有结果表明CAMP可以提供准确的肽-蛋白相互作用预测,并有助于理解肽结合机制。
未来,研究者计划加入更多的数据,如结合域信息,以进一步改进预测蛋白结合残基的结果。
参考文献
Lei, Y., Li, S., Liu, Z. et al. A deep-learning framework for multi-level peptide–protein interaction prediction. Nat Commun 12, 5465 (2021). https://doi.org/10.1038/s41467-021-25772-4