
引言
针对具有大的、无特征的、高度亲脂性的或高度极性的和高柔性等特点的结合位点的靶标的先导化合物的生成是非常具有挑战性的。本文作者描述了大环天然产物的分子核心如何充当高质量的计算机筛选库,从而为难以药物靶向的靶标提供起始的先导化合物。针对仔细选择的一组天然产物衍生的核心进行了两轮循环的分子对接,结果发现了Keap1-Nrf2蛋白与蛋白相互作用的不带电荷的大环分子抑制剂,Keap1-Nrf2由于其高极性的结合位点一直以来都是一个特别具有挑战性的靶标。该大环分子抑制剂显示出细胞活性,并且基于其与Keap1的复合物的结构和合成途径,该大环分子能够较好的得到进一步的分子设计与优化。作者相信本文工作将激发人们对使用大环核心用于基于计算机的潜在先导化合物的生成的兴趣,并且还有益于未来的大环筛选库的收集与设计。
背景介绍
在所有已被发现的与人类疾病有关的靶标中,有超过一半的靶标被认为很难依靠符合Lipinski的“类药五规则”(Ro5)的传统小分子药物来调节,蛋白质-蛋白质相互作用(PPI)、蛋白酶、某些激酶以及转移酶和异构酶就是其中重要的例子。这些难以药物靶向的靶标通常具有大的、无特征的、高度亲脂性或高度极性和高柔性的结合位点。寻找细胞内难以药物靶向的具有口服生物利用度的药物是一项艰巨的任务,通常需要在Ro5以外(bRo5)的未知化学空间中发现相应配体。大环化合物能够在bRo5空间中的口服药物中富集,因为与传统的小分子相比,它们可以与具有大而无特征的结合位点的靶标更好的结合。此外,大环化可以改善血浆稳定性、细胞通透性和口服吸收。但是,大环化合物在筛选分子收集库中的代表性通常不足,限制了其在先导化合物生成中的使用。例如,阿斯利康(AstraZeneca)分子收集库中总共有380万种化合物,但包含不到17000个大环化合物。
尽管有越来越多的bRo5化合物进入了临床试验,但难以系统地研究难以药物靶向靶标的先导化合物的生成,并且随着化学空间随着分子体积大小的增加而急剧扩大,这是一个很大的挑战。天然产物是重要的药物来源,我们假设大环的核心可能是自然界的优势子结构,并且在难以药物靶向靶标的先导化合物的生成中可以发挥作用,就像传统药物的分子片段一样。类先导化合物的大环核的集合随后将可用于对靶标(例如PPI)进行计算机筛选,随后的优化可以提供新颖的先导化合物。在这里,我们报告了通过对大环天然产物化学空间的全面研究,如何生成两组可用于发现难以药物靶向靶标的先导化合物的大环核心。通过将更小和更像先导化合物的化合物对接到Keap1上Nrf2的高电荷结合位点中,确定了环噻啶的核心是Keap1的潜在抑制剂;使用核的结构简化类似物进行第二轮对接研究,然后进行合成,确定了Keap1-Nrf2 PPI的弱抑制剂。基于该hit抑制剂合成了另外九种优化的化合物进而得到了活性在低μM范围内并显示出细胞活性的抑制剂14。Keap1和14的复合物的晶体结构揭示了不带电的大环分子14如何与Keap1的带电结合位点结合,并为其进一步优化提供了基础。
结果1: Dictionary of Natural Products的挖掘
Dictionary of Natural Products (DNP) 包含超过15万种化合物,涵盖了广阔的化学空间,其中包括已经批准上市的口服和非肠道药物(图1a)。因此,我们认为DNP是用于鉴定大环核心的有吸引力的来源。首先,使用在Pipeline Pilot中实施的HTS过滤器从DNP中除去含有已知毒理基团和非常活泼的基团的天然产物,该过滤器除去了包括酰基卤、酸酐、重氮基团和酰肼在内的几种官能团。然后,将剩余的化合物进一步过滤,以包含至少一个大环,不超过十个较小的环,并且分子量(MW)小于2500 Da(图1b),这一步删除了所有非大环和大型大环,例如多糖或多肽性质的大环,最终获得了3657个大环的数据集。使用先前报道的片段生成算法的前两个步骤,将该数据集侧链修剪到由至少两个与大环连接的重原子或官能团组成的连接点。过滤得到的扩展的Murcko骨架(本文中称为大环核),使其至少包含总共三个氧和氮原子并且小于15个可旋转键,从而去除不太可能发展为药物的高亲脂性和柔性核。整个大环收集流程下来,得到了将近2200个结构不同的核心,这些核心被聚集成217个代表性核心的集合。通过视觉检查去除了含有少量剩余反应性基团(如二硫化物、过氧化物和烷基卤化物)和具有高度合成复杂性的核以及寡聚体(寡肽和寡糖)。这提供了较小的41个更加类似先导化合物的核心的数据集,这些核心在很大程度上遵守了Lipinski的5规则。这组核心来自已经报道其绝对立体化学的35种天然产物以及仅了解相对立体化学的三种天然产物。这两组核心构成了独特的计算机筛选分子库,且两组核心之间的结构复杂性各不相同,其中一些很容易合成,而简化的类似物可能是其他大环化合物更有吸引力的起点。
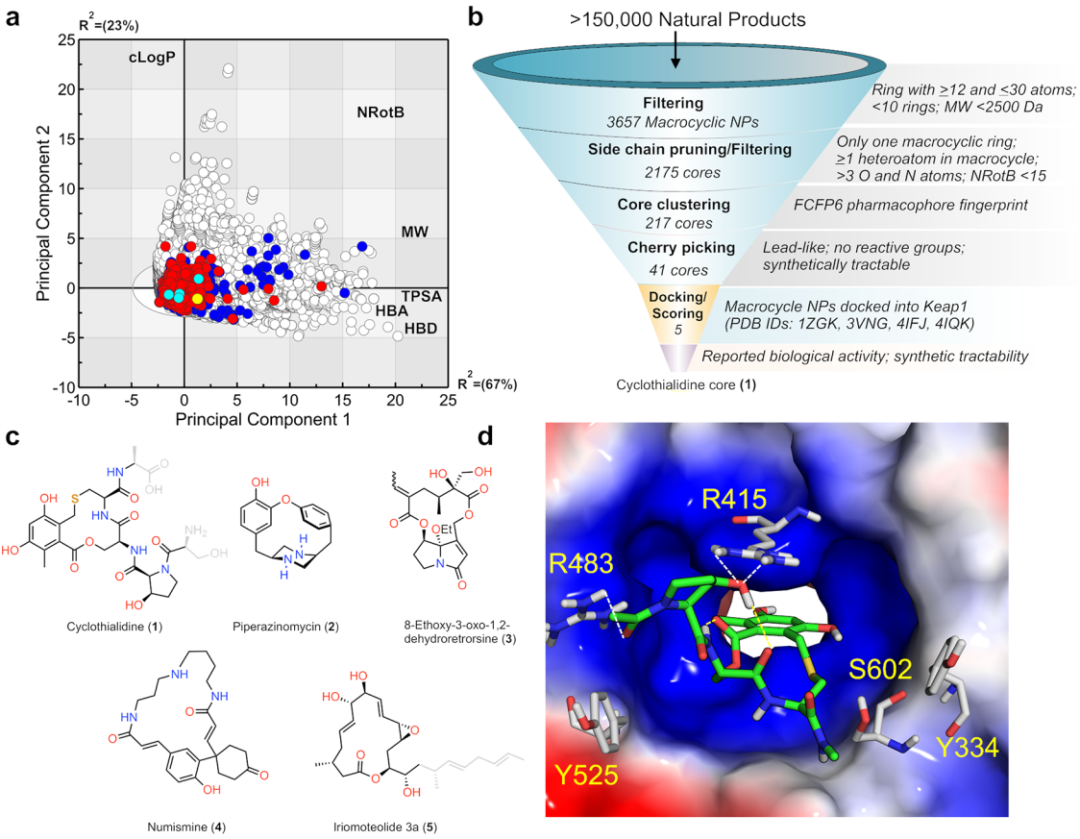
图1. 从DNP中挖掘的大环核心
图片源自JMC
结果2:Keap1-Nrf2 PPI的新型抑制剂
我们选择了Kelch样ECH相关蛋白1(Keap1)和核转录因子E2相关因子2(Nrf2)之间的PPI来评估我们的大环核心数据集。Keap1-Nrf2系统是氧化应激的重要细胞应答机制,氧化应激涉及许多慢性、年龄相关或炎症性疾病。该PPI的抑制剂是药物开发的主要兴趣,因为Nrf2控制着几种细胞保护性基因的表达,但通过与Keap1的复合作用而受到负调控,从而导致其泛素化和降解。Keap1-Nrf2 PPI由Keap1上的一个高度极性口袋组成,该口袋以高亲和力结合Nrf2上的肽段序列DEETGE和其他带负电荷的配体。我们假设大环由于其构象限制,可能在Keap1的极性结合位点提供最佳结合。与目前大多数为酸性的Keap1抑制剂相比,大环对于发现新型不带电抑制剂十分理想,并且大环具有改善的细胞通透性和口服吸收性等性质。为了避免在对接大环核时偏向特定的结合位点构象,我们选择了Keap1的四个高分辨率晶体结构,这些结构在结合位点上表现出差异:两个结合小分子配体(PDB ID:4IQK和3VNG)和两个空蛋白结构(PDB ID:4IFJ和1ZGK)。通过GlideScores确定对接Keap1结构最好的十个核心。然后,从合并的前十列表中找出已经对接到Keap1结构中的三个或四个的核心,并选择它们作为潜在hit。有趣的是,由此产生的前五个大环表现出很大的结构多样性(图1c)。分析对接pose表明,环噻啶的核心很好地对接在所有四个Keap1结构中,能够在结合位点深处结合并到达Kelch通道(图1d)。诱导契合对接表明,环噻啶核的简化类似物,如立体异构体6-9,其中亚苯基的羟基和甲基以及脯氨酸的羟基已被去除(图2a),也能够与Keap1结合。特别地,在溶液分析(ISA)中基于表面等离子共振(SPR)的抑制实验中,有9种能够抑制Keap1与Nrf2衍生肽的结合(KD = 237μM)(图2a)。将I-脯氨酸部分的立体化学转化为10导致结合亲和力降低,这表明其对抑制Keap1的重要性,而I-脯氨酸的羟基化(11)则保持KD(图2a)。重要的是,用不同的酰胺(12–15)取代甲酯可提供二甲酰胺14,其活性比9强2个数量级。开环16的活性大大低于14;同样,大环分别扩展一个和三个原子(17和18),也失去了结合亲和力,证实了大环对于抑制Keap1的关键作用。ISA中14的活性通过直接SPR结合测定法和等温滴定热法(ITC)证实,这为Keap1提供了几乎相同的亲和力(图2b,c)。大环14通过抑制与Keap1的结合而诱导Nrf2易位进入细胞核,显示出细胞活性(图2d)。此外,14具有较高的水溶性、跨Caco-2细胞单层的中低渗透性以及中等的体外微粒体清除率。因此,14是一种有前途的先导化合物,可以进一步优化为不带电的非酸性的靶向Keap1的高级先导化合物。
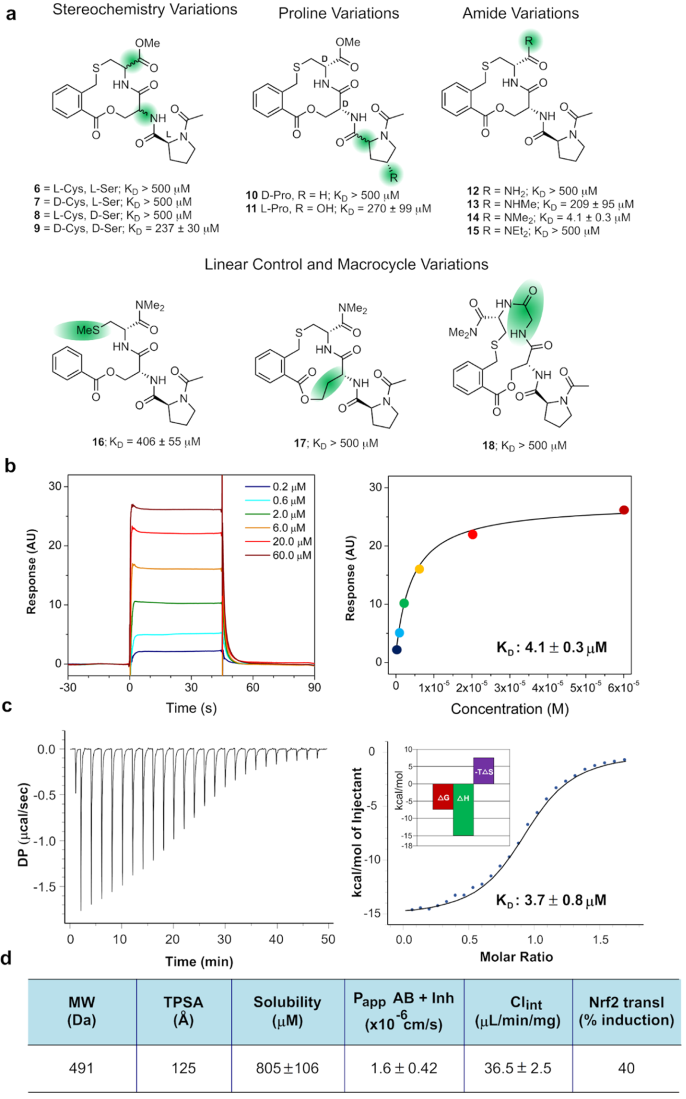
图2. 环噻啶类似物的表征
图片源自JMC
结果3: 抑制剂14与Keap1的复合物结构
我们确定了大环分子14和Keap1在2.4 Å分辨率下的复合物结构,以了解不带电荷的14如何与Keap1的带电和极性结合位点结合。晶体结构证实14与Nrf2一样结合在Keap1的同一个极性结合位点(图3a)。对结构的分析表明,亚苯基和大环的一部分被楔入R415和A556之间的相当疏水的口袋中,除了R415和14之间的阳离子-π相互作用外,复合物仅通过少数极性相互作用得以稳定。此外,有个氯离子桥接了14中的Ser的NH和Keap1中的三个残基,与通过基于片段的药物发现鉴定的具有高效抑制剂的复合物相似。由于在SPR和ITC分析以及细胞Nrf2易位分析中使用了生理盐条件,因此我们认为这种复合物的形成也发生在生物学相关的环境中。大环化合物14在结合位点采用紧凑且稳定的结合构象的能力解释了为什么开环和扩环类似物16-18对Keap1具有显著较低的亲和力。Pro的一部分及其N-乙酰基在溶剂中的暴露符合以下事实:用羟脯氨酸(化合物11)替代不会影响抑制活性。使用Prime MM-GBSA进行配体结合亲和力计算,从而提供了对活性差异的潜在原因以及14和Keap1之间复合物稳定作用力的解释(图3b)。与实验解离常数相符,大环14被预测对Keap1具有最高亲和力,其中9和13被预测为中等,而12和15被预测为相对较弱。我们发现非极性范德华和亲脂性相互作用是14的结合亲和力的主要贡献者,并且它比抑制剂9和13具有更高的亲和力。根据计算,非活性化合物12通过更强的极性(库仑)相互作用而得以稳定,但同时被较大的去溶剂化作用抵消;而非活性化合物15形成了所研究的五个化合物之中最弱的极性(库仑)和范德华相互作用。有趣的是,两个14的N-甲基与Pro和N-乙酰基的羰基之间的距离表明在这些残基之间形成了两个分子内非经典氢键(图3a),这一发现得到了量子力学计算的支持。非经典氢键比氢键弱,但是两个分子内非经典氢键可能会提供额外的构象限制来稳定14与Keap1的结合。
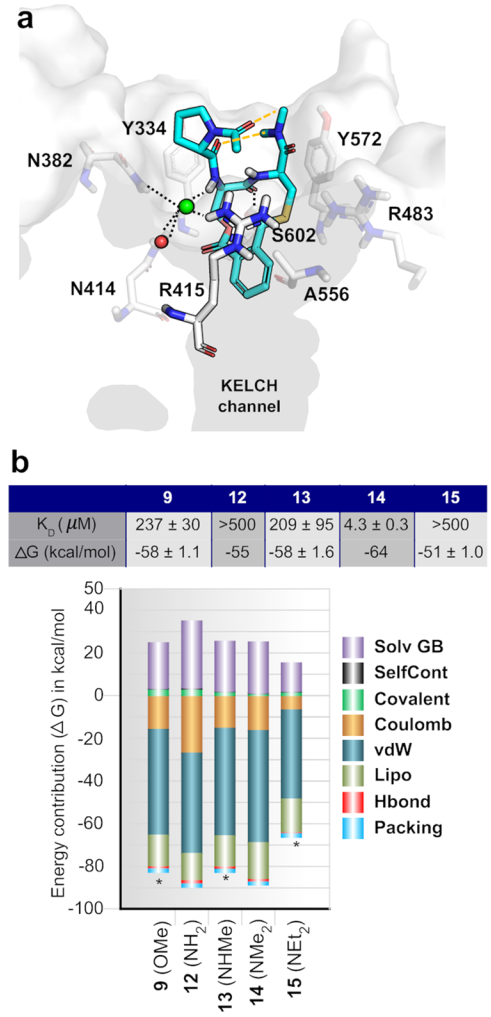
图3. Keap1-14复合物的表征
图片源自JMC
结果4: 抑制剂14与其他Keap1抑制剂的比较
从七个指纹中计算出用来描述结构多样性的Tanimoto系数,结果显示抑制剂14与PubChem和验证数据集中的化合物相似性较低(图4a)。Keap1抑制剂空间的多样性是通过类似网络的相似性图进行研究的,该相似性图来自抑制剂14、组合的验证组和PubChem组以及单独的验证组计算得出的子结构片段指纹。组合数据集的相似性图与验证组的一样,均证实了大环化合物14在化学空间中占据了唯一的没有相似结构的位置(图4b,c)。组合数据集还显示,Keap1抑制剂的空间相当多样,大多数抑制剂几乎没有结构上的相似化合物,只有大簇位于图的左中心。该簇包含一系列已验证的抑制剂V1代表的邻苯二甲酰亚胺(图4c)。1,4-二氨基萘V3、环磺酰胺V7和二唑V8(图4c)是经过药物化学优化后获得的最有效的Keap1抑制剂。但与14相反,它们都含有羧酸。总而言之,Tanimoto系数和类似网络的相似图都凸显了使用天然产物核心作为结构独特的Keap1抑制剂来源的潜力,该Keap1抑制剂属于与先前报道的抑制剂完全不同的另类化合物。
图4. 抑制剂14与已经报道的Keap1-Nrf2 PPI抑制剂的结构相似性
图片源自JMC
总结
近来,天然产物也已被用作三维(3D)片段的来源,并启发了通过面向多样性的合成(DOS)制备化合物集合的设计灵感。此外,还有通过将天然产物衍生的片段组合和融合来设计假天然产物。但是,天然产物及其衍生物通常具有复杂的结构。我们展示了如何通过两轮对接较小的一组类先导化合物的大环核心来识别出环噻啶核,然后通过化合物6–9的对接使其针对靶标Keap1量身定制,同时降低结构复杂性。通过这种方式,减轻了天然产物的主要缺点之一,即它们通常需要通过较长的合成路线来制备。总之,本文报道的大环核心说明了如何利用自然界的优势子结构及其多样性作为药物发现的高质量起点的丰富来源。
参考文献
F. Begnini, V. Poongavanam, B. Over, M. Castaldo, S. Geschwindner, P. Johansson, M. Tyagi, C. Tyrchan, L. Wissler, P. Sjö, S. Schiesser, and J. Kihlberg, Mining Natural Products for Macrocycles to Drug Difficult Targets, J. Med. Chem., 2021, 64, 2, 1054-1072. DOI: 10.1021/acs.jmedchem.0c01569.