
引言
对未知病原体迅速作出反应,如新型冠状病毒,于制止导致流行病的疾病不受控制的传播至关重要,可将保护措施维持在造成尽可能少的社会和经济危害的水平。而以上,可通过显著加快药物发现的计算方法来实现。一个强有力的方法是,通过现有药物的再利用,来减少全新药物的研究,这可以大大加快通常漫长的审批过程。在这里,研究者展示了,一套目前用于识别COVID-19可重复使用药物的代表性计算方法及其基础数据资源。
先睹为快
来自德国慕尼黑工业大学的Jan Baumbach & Josch Konstantin Pauling等研究者,比较了计算方法预测的候选药物和临床试验评估的药物,并从回顾的研究工作中获得的经验教训,包括如何成功地将计算方法与实验研究联系起来,提出了一个统一的药物再利用战略,以便在未来爆发疫情时,提前做好准备。
最近的研究,越来越多地使用计算方法,来系统性地预测新的药物靶点或药物再利用候选药物。与实验性的高通量筛选相比,计算机模拟的方法更快,成本更低,并且可以作为评估数千种化合物的初始过滤步骤。因此,它们对于确定需要进一步评估和实验验证的药物的优先级是有用的。这需要应用适当的算法方法,来确定与疾病相关或特定的机制。
本文所涵盖的研究,采用了标准的药物再利用工作流程和数据驱动算法。本文,将重点讨论用于药物再利用的计算方法,它们在一系列研究(同行评议和预印本)中的应用、可用性和可行性,这些研究涵盖了各种不同的方法。值得注意的是,大多数的研究在临床上并不被认为是成功的。然而,重要的是,要适当地评价和提高利用现有药物,以及宿主和病毒生物学信息的计算机模拟方法的预测能力,即便当前新出现病原体的数据十分有限。这有助于对感染作出迅速和实际的反应,从而在今后的大流行中取得更大的成功,特别是在大流行的早期阶段,或在疫苗开发之前应对感染病例的上升。
数据资源
数据共享加速了研究,因为计算方法依赖于高质量的数据集,而实验室并不需要自己收集信息。用于COVID-19药物再利用研究的大量资源表明,通过社区的大力努力,可以迅速生成和收集数据。本节介绍了在回顾的研究中,使用的数据资源的选择,以描述加速计算药物再利用方法的资源类型:其中大多数是在大流行之前就已建立的一般数据资源,但已被COVID-19或SARS-CoV-2特异性数据所扩展。
1.1 分子数据资源
评审出版物中使用的所有分子数据,均取自已建立的一般数据资源,并迅速扩展为SARS-CoV-2特异性数据。GenBank、GISAID倡议或UniProt等资源,提供了宿主和SARS-CoV-2的基因组/蛋白质组学信息。收集蛋白质信息的结构资源,如蛋白质数据库(Protein Data Bank, PDB),被扩展为不同的SARS-CoV-2特异性蛋白。最后,收集基因表达数据的转录组资源,被用于几种COVID-19药物再利用方法。例如,基因型-组织表达(GTEx)程序,提供了对组织特异性基因表达的理解。肺组织表达是COVID-19药物再利用研究的重点,并且经常被整合到计算模型或研究中。其他资源,如LINCS L1000数据库,描述特定药物治疗条件下的基因表达变化,并用于识别与SARS-CoV-2感染样本反向表达谱的药物。
1.2 网络与相互作用资源
蛋白质-蛋白质相互作用(PPI)网络,使宿主蛋白或病毒蛋白与其他宿主蛋白相互作用的可视化和分析成为可能。此外,PPI网络允许特定的适应和搜索策略(例如,边缘过滤),并可以连接到药物资源。Gordon等人,鉴定了332种高度可信的SARS-CoV-2与人类蛋白之间的病毒宿主相互作用。这是该综述中,唯一一个新创建的、专门与SARS-CoV-2相关的资源。VirHostNet,其是2019/2020年SARS爆发前已经存在的病毒宿主PPI资源,它也扩展了167个新的SARS-CoV-2相互作用。除了分子网络,知识图谱,如全球生物医学关系网络(GNBR),已经证明了它们在药物再利用方面的效用。这些网络包括从文献中收集的各种类型的生物关系,并被整合到COVID-19药物再利用方法中。
1.3 药物和试验资源
在大流行之前就已经存在的药物数据库,并随着新开发药物的不断扩大,用于将不同方法的结果与潜在药物联系起来。一个广泛使用的药物数据库是DrugBank,它拥有超过13000个已批准和正在试验的药物,包括药物靶点。另一方面,ChEMBL和ZINC15含有数以百万计的具有药物性质的化合物。药物再利用方法也得益于试验数据库,因为它们可以用来验证,预测的药物是否已经在试验中或尚未被评估。这些资源的范例,有欧盟临床试验注册(https://www.clinicaltrialsregister.eu/)和ClinicalTrials.gov (https://clinicaltrials.gov/)。后者包含来自219个国家的超过35万份研究报告。
药物再利用研究
过去的时间内,全世界的人类迅速调动了各种临床、实验和计算药物再利用工作,优先确定化合物,以确定有希望用于SARS-CoV-2大流行的候选药物。在本节中,研究者考察了一些代表不同计算方法的研究,以确定COVID-19的潜在新靶点和可重复使用的药物。
2.1 病毒靶向方法
病毒靶向方法大多依赖于基于结构的药物筛选方法,其利用目标蛋白的三维结构,来预测已知化合物与蛋白质的亲和力或相互作用能(图1)。这些方法主要用于识别靶向病毒蛋白的候选药物,所以称它们为病毒靶向方法。该方法采用了两种主要的方法工作流程,即基于结构和基于深度学习(DL)的药物筛选。在这里,研究者描述了这些方法,并比较了23项COVID-19药物再利用研究。
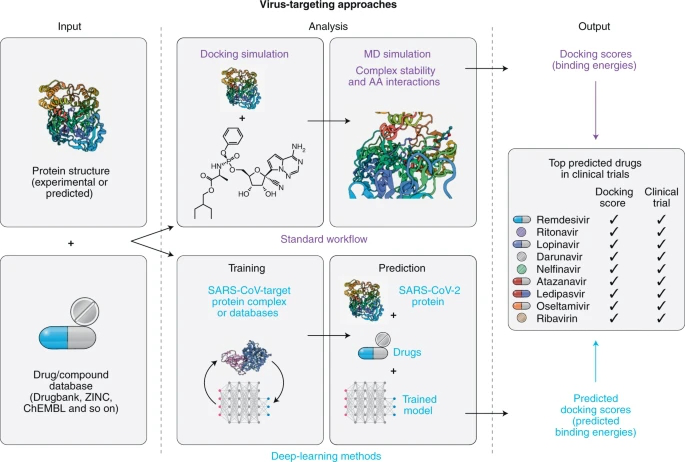
图1. 病毒靶向计算药物再利用方法的工作流程
图片来源于Nature Computational Science
2.1.1 基于结构的药物筛选
基于结构筛选的第一步是药物库和目标蛋白的选择。对于COVID-19,靶向病毒蛋白的直观候选药物是抗病毒药物。因此,许多研究将他们的研究局限在这些方面。筛选到的抗病毒药物,从3种到123种不等。更广泛的研究,如Chen等人的研究,结合来自KEGG(京都基因和基因组百科全书)和DrugBank数据库的化合物筛选了7173种药物。另一个关键步骤是选择目标蛋白及其相应的三维结构(实验或预测)。Wu等人对病毒的19个编码蛋白进行了筛选。相比之下,其他大多数研究集中在3CLpro、包膜(E)、spike、RNA聚合酶和甲基转移酶蛋白。利用已开发的软件,如Autodock和Glide,对药物库进行虚拟筛选。采用相应的评分方法筛选候选药物,然后用分子动力学模拟进行验证。大多数药物预测用于3CLpro,这也是大多数研究的重点(17项研究),其次是RdRp和PLPro。对于3CLpro,每个研究的预测范围在2到27种药物之间。最常预测的5种药物是利托那韦(8项研究)、洛匹那韦(6项研究)、奈非那韦、瑞德西韦和沙奎那韦(各5项研究)。然而,有99种候选药物仅在一项研究中得到了预测,显示了结果候选集的高可变性。有趣的是,对整个数据库进行筛选的研究后,所预测的抗病毒药物,也是得分最高的药物。
2.1.2 基于DL(深度学习)再利用策略
DL模型可以预测结合亲和力或对接分数,并显示出了传统对接协议的优势。虽然标准的对接协议仅限于数百万种,但DL方法可以分析数十亿种化合物。这使得它们可以应用于整个数据库,从而增加了被测试化合物的多样性和发现非常规化合物的可能性。此外,它们能够处理更多(物理)化学特征,并能找到与不利对接相关的特征。然而,这些方法大多需要训练数据集,这些数据集通常来自真实的对接模拟;因此,许多基于DL的方法的性能仍然依赖于用于训练的对接软件的准确性。Ton等人,开发了DeepDocking,利用经过训练的定量结构活性关系模型,来预测靶向SARS-CoV-2 3CLpro蛋白的化合物的对接分数。它需要更少的对接管道,因为它只对化合物的子集进行对接,并且可以产生更少的化合物,这也丰富了潜在的顶级碰撞。Nguyen等人,开发了MathDL方法,该方法利用药物靶标蛋白复合物结构的低维数学表示,然后将其输入DL算法来预测药物蛋白复合物的结合能。对于SARS-CoV-2,研究者使用来自PDBbind和SARS-CoV蛋白酶抑制剂的SARS-CoV配体3CLpro复合物的实验结合亲和力数据作为训练数据,预测了SARS-CoV-2 3CLpro的药物库化合物的结合能量,且不依赖于对接软件。Beck等人,开发了一种基于DL的药物-靶标相互作用预测模型,命名为分子-药物靶标相互作用。它利用简化的分子-输入线-输入系统(SMILES)表示的药物和蛋白质序列作为输入训练和预测亲和力。对于SARS-CoV-2,该模型接受了商用抗病毒药物和病毒靶蛋白的训练。目前,已经用于对抗SARS-CoV-2的抗病毒药物,发现其在鉴定的候选药物中。
2.2 宿主靶向方法
宿主靶向方法,涉及到识别可能干扰导致病毒发病机制的宿主机制的药物,这也使它们不太容易产生耐药性。此外,SARS-CoV-2感染可触发过度反应性免疫反应,其特征是过度释放促炎细胞因子和趋化因子。因此,调节宿主免疫应答的药物,可针对特定的失调通路,使COVID-19重症患者受益。
2.2.1 基于特征方法
基于特征的方法,主要利用SARS-CoV-2或密切相关的人类冠状病毒感染样本的转录组数据集,通过连接映射来识别候选药物(图2),这是一种成熟的方法,它依赖于寻找药物诱导的表达特征,显示出与疾病特征相反的特征。几项研究将此作为识别COVID-19新疗法的主要方法。Loganathan等人,对病毒感染的细胞进行了差异表达分析,并在感染条件下提取了一致的失调基因。它们可用来查询连接图数据库的药物扰动概况,显示抗相关的表达特征。Jia等人,实施了一种改进的方法,其中来自感染和健康个体的表达数据,用作途径引导的药物再利用框架的输入。在反向特征匹配之前,它们鉴定了疾病共表达簇并进行了富集分析。
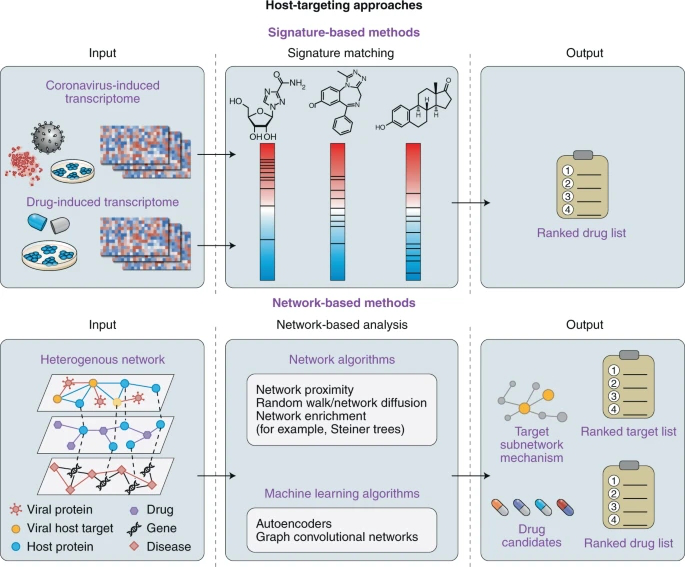
图2. 宿主靶向计算药物再利用方法的工作流程
图片来源于Nature Computational Science
2.2.2 基于网络的方法
用于COVID-19药物再利用研究的基于网络通用方法,整合了多个数据源,包括病毒-宿主相互作用、PPIs、共表达网络、功能关联或药物靶标相互作用(图2)。基于网络的算法或拓扑测量被应用到组装的网络中,以识别相关的宿主蛋白质目标或宿主相互作用组的区域。多项研究,以基于随机游动的算法作为识别新的推定药物靶点的主要方法。Law等人,在病毒-宿主相互作用组上实现了几种算法,以识别其他SARS-CoV-2相互作用体。冠状病毒刺突蛋白的主要功能是介导病毒进入宿主细胞。类似地,但侧重于特定背景,Messina等人利用三种密切相关的冠状病毒的数据,探索了spike蛋白触发的致病机制。他们以刺突蛋白为种子,在组装的分子网络上实现了随机行走算法,以识别COVID-19的相关目标。此外,CoVex实现了TrustRank (PageRank算法的一种变体),将用户自定义的种子分数传播到其他宿主蛋白质,并对宿主药物靶标进行排序。网络邻近性依赖于这样一种原理:如果一种药物的目标是相互作用组中疾病相关蛋白附近的蛋白质,那么它就可以有效。Zhou等人,利用这一概念计算了药物靶点和人类相互作用组中冠状病毒相关蛋白之间的网络邻近性。他们还使用了“互补接触”模式(根据网络邻近度预测的两种药物靶点之间的最短距离),以确定治疗COVID-19患者的潜在药物组合。一些研究结合了多种基于网络的策略来预测候选药物。Gysi等人,利用实验确定的SARS-CoV-2相互作用物,表征并提取COVID-19疾病模块。他们进行了基于网络的分析,分析了组织特异性和潜在的疾病并发症。他们对病毒-宿主相互作用组采用了一种多模态方法,将网络邻近性、扩散状态距离和图形卷积网络(GCNs)结合起来,以识别可干扰与COVID-19疾病模块相关的宿主蛋白活性的药物。对不同的药物管道进行排序聚合,得到了最终的药物清单。CoVex,是一个研究SARS-CoV和SARS-CoV-2病毒宿主药物相互作用组的网络平台。用户可以预测药物目标和候选药物使用多种图表分析方法,允许定制种子蛋白作为输入。例如,KeyPathwayMiner是一个网络充实工具,它通过从宿主交互组(从种子开始)提取最大连接的子网来识别特定条件的子网。CoVex还实现了一种加权多斯坦纳树方法,它将几个非唯一的斯坦纳树近似聚合成一个单一的子网络,这些子网络是连接种子集的最小代价子网络。其他研究还利用机器学习预测抗SARS-CoV-2的候选药物。Belyaeva等人,在特征匹配和基于网络的方法之间实现了一种混合方法。通过使用自编码器,他们学习了药物的特征嵌入,使用药物诱导的表达谱识别出与SARS-CoV-2感染特征相反的药物。采用斯坦纳树和因果网络发现算法,可提取SARS-CoV-2和衰老介导的机制。Ge等人采用GCN算法,构建了病毒相关知识图。候选药物的列表,通过文本挖掘进一步过滤现有的抗病毒活性证据。同样,Zeng等人从PubMed文章中收集了一个大规模的知识图谱。然后,应用GCN模型学习了节点和边缘的低维嵌入。
经验教训
接下来,研究者将对所审查的数据资源和计算方法的质量和潜力进行研究,以改进未来大流行的应对措施。
3.1 数据资源
分子数据集的可用性是快速开发药物再利用方法的前提。除此之外,基于网络资源是药物再利用的一个巨大推动力。然而,大部分出版物仅基于少数主要资源,这往往会导致偏差或测量误差的风险。此外,唯一使用的分子相互作用网络类型是PPI。然而,由于没有一种方法包括结构数据,因此需要高可信度的PPIs。未来,还应考虑其他网络类型,如基因调控网络。其他数据资源,如药物的超说明书数据,也应该整合到药物再利用研究中。最后,现有的药物和试验资源,可广泛用于药物再利用管道的开发。然而,研究者发现试验资源没有标准化,这使得由于不同的名称、不同的拼写或打字错误而难以分析某些药物的试验。药品资源(如药品库)通常都是标准化的,但有些正在试验的药品在数据库中找不到。保持资源的更新和相互关联应该是一个重点,以此提高可访问性。
3.2 计算预测
评估预测的质量是具有挑战性的,因为许多研究没有同行评审,不进行实验评估,或依赖临床试验数据库。研究者通过从临床试验网站ClinicalTrials.gov (https://clinicaltrials.gov/)和Biorender (https://biorender.com/covid-vaccine-tracker)数据库中,确定最终候选药物列表和正在进行临床试验的药物之间的重叠来检验预测的质量。此外,研究者还提供了补充的体外筛选数据,如病毒靶点的IC50值和SARS-CoV-2细胞培养研究中的抑制指标。汇编这些数据的努力表明,大量的预测还没有经过试验检验。
评估病毒-靶向方法
研究者确定了53种通过对接模拟预测且目前正在进行试验的药物。Wu等鉴定了大部分药物(36种);然而,这些药物被预测用于多种病毒蛋白(例如,洗必泰用于11种病毒蛋白,甲氨蝶呤用于6种不同的病毒蛋白)。这表明他们的方法,没有产生特定的和可行的候选方案。排除本研究后,除氯喹(3CLpro和PLpro)和瑞德西韦(3CLpro和RdRp)外,其余药物均仅预测一种特定蛋白。通过使用3CLpro主蛋白酶的对接模拟预测,临床试验中排名前五的药物,在纳入的对接研究总数中,预测达鲁那韦(darunavir)、瑞德西韦(remdesivir)和洛匹那韦、奈非那韦、利托那韦分别为14.3%、19.0%和23.8%,表明对于每种药物,大多数研究都不能预测它们。Beck等人,采用DL方法鉴定了类似的药物,这些药物正在进行多项临床试验,包括利托那韦、洛匹那韦和瑞德西韦。然而,这些抗病毒药物还没有在病人身上显示明确的结果。对于利托那韦/洛匹那韦,只有4个试验完成,初步结果表明治疗后的结果没有差异。因此,需要进一步调查。瑞德西韦的一些试验已经完成,在患者和人类细胞系中的初步结果表明,它可以有效治疗SARS-CoV-2感染。抗病毒药物,一直是数据库中检索最多的药物,表明该方法的准确性较高。这些药物是实验筛选或临床试验的良好候选药物,与计算预测的可靠性无关。更有趣的候选药物,是这些方法确定的额外的药物;然而,这些药物很少得到实验验证,而且大多数都没有进入临床试验。在DL方法的新兴领域也观察到了类似的情况,其中大多数研究集中于证明其预测的准确性和开发基准数据集。基于DL和对接模拟的方法是很有前途的工具,以确定可重复使用的药物,因为它们能够在短时间内提供结果。虽然已经建立了对接模拟的标准工作流,基于DL的方法可能健壮地交付可测试的候选药物。然而,对接研究很少经过同行评审,并发现了非常不同的候选集,同时使用不同的分数进行评估和排名。因此,有必要通过系统的实验比较来验证这些结果。
评估宿主-靶向方法
宿主-靶向方法,通常涉及多种组学类型的整合和分析,并采用基于数据驱动的网络方法;因此,一个主要的限制是缺乏金标准的数据集,以及来自MERS-CoV(中东呼吸综合征冠状病毒)和SARS-CoV暴发的数据稀缺。在获得足够的SARS-CoV-2特异性数据之前,早期研究使用了初步数据或使用密切相关病毒的数据增强了分析。尽管预测的质量高度依赖于数据,但持续生成SARS-CoV-2特异性组学数据和等待临床研究结果有望改善预测。临床专家知识,对于根据毒性和药理特性等标准筛选药物预测,仍然至关重要。然而,由于可用数据有限,这些候选药物在试验中的有效性仍有待确定,并不能得出明确的结论。临床试验中药物的重叠程度一般较低,但主要基于知识图的集成方法预测的41种药物中有26种以上也正在进行临床试验。值得注意的是,在撰写本文时,注册临床试验的药物也被用作验证集,更多的预测药物随后被注册临床试验。研究注意到一些药物,可以通过基于特征和基于网络的方法预测,因此需要进一步的检查。六项研究中有四项预测了利巴韦林,从而为其预测疗效提供了机制基础。三项研究也预测了用于治疗类风湿关节炎的甲氨蝶呤。值得注意的是,进入临床试验后期的药物,不是根据计算机模拟来选择的,而是根据以往SARS或中东呼吸综合征暴发的临床经验来重新用途,并根据减轻疾病症状的已知效果来选择的。此外,在回顾的大多数研究中,这些预测并没有得到实验验证。在药物再利用和临床应用的计算工作之间的转换差距,是药物再利用和一般药物中被广泛承认的主要瓶颈。系统验证工作的结果,对于确定特别适合COVID-19背景下药物再利用的算法和数据集也很重要。鉴于在大流行中确定有效疗法的紧迫性,临床医生、实验生物学家和计算生物学家之间的密切合作有望解决这一差距。
5.1 一种统一的药物再利用策略
尽管计算预测的药物再利用和临床试验之间存在重叠,但没有迹象表明临床试验是基于计算预测进行的,尽管它们有很好的潜力。对于未来的大流行,计算工具应该能够提供有希望的候选病毒集,然后在试验或筛选中进行验证。因此,一个统一的策略是十分必要的。
5.1.1 标准化数据的可用性
新开发的方法通常依赖于相同的数据类型(图3a)。在未来的疾病爆发中,快速生成不同类型的数据是关键的初始步骤。值得注意的例子,是Gordon等人的交互数据和3CLpro结构的发表,它们都被随后的许多研究所使用。然而,从不同实验室获得的数据集的实验复制和不同数据类型的集对于提高鲁棒性至关重要,仍需要改进。
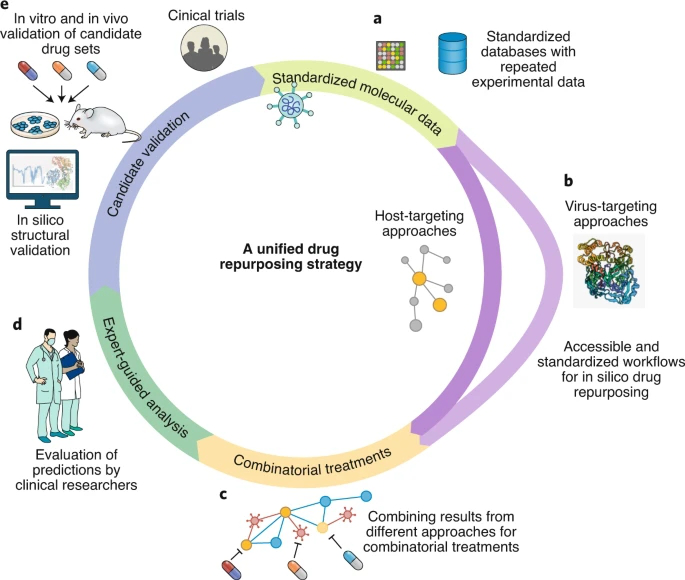
图3. 提出统一药物再利用策略的要素
图片来源于Nature Computational Science
5.1.2 工具可访问性
尽管有各种各样的计算工具和软件,但迄今为止对临床研究人员在COVID-19大流行期间的实际应用有限(图3b)。对于病毒靶向治疗,对接管道保持稳定,并开发了大量软件;然而,它们对应的输出显示出很大的可变性,这取决于所使用的算法,降低了可比性(标准化问题)。对于宿主靶向治疗,计算机模拟方法更具有方法论上的多样性,并开发了几种针对特定生物背景的策略。然而,在COVID-19大流行的背景下,计算工具和软件的普遍可用性非常有限。工具可访问性,允许研究人员使用开发的算法运行自定义分析(例如,对新获得的数据)。这将帮助非计算科学家使用这些工具,并继续验证例程,避免许多未验证的预印手稿,从而加速研究。
5.1.3 预测的整合
不同方法的结果并没有完全整合。在基于结构的再利用方法中,可以通过不同的对接工具或同源建模方法获得候选药物,从而得到一个再利用药物的集合(图3b)。对于宿主靶向治疗,一项研究使用类聚来整合不同算法的结果。另一项研究将模型的输出与基因集富集和专家知识的结果相结合,得出了最终的预测。值得注意的是,临床试验中的药物被用于开发方法,这两项研究预测与临床试验中测试的药物重叠比例最高。后者显示了集成方法的潜力,众所周知,它可以输出更稳健的结果。多种方法的整合可以显著增加对候选药物的再利用的信心,并指导临床研究人员通过药物选择过程。这需要一个精简的解决方案,考虑到工具的可访问性和标准化,就像存储支持meta分析的候选药物预测的标准化数据库一样。
5.1.4 组合治疗发展
计算识别协同药物组合是一个尚未开发的领域,它可以提供高度有价值的信息,以增加临床决策,因为它们已被证明比发现单一疗法更有效(图3 c)。到目前为止,病毒和宿主蛋白的靶向已经独立完成。在考虑副作用的同时,还缺乏寻找互补药物组的方法。结合病毒和宿主靶向药物是一种很有前途的策略,通过与病毒复制抑制剂结合,阻断SARS-CoV-2进入细胞所需的病毒和宿主分子机制,并破坏参与疾病进展的宿主通路。预测的组合治疗,可以大大减少后续体外验证的搜索空间。现有的筛选数据库,如美国国立卫生研究院开放数据门户或ReFRAME图书馆已经很少被使用,但是它们的潜力还没有被耗尽。通过在计算机模拟中扩展它们,可将计算机模拟和体外研究联系起来,并帮助确定有前途的组合治疗。此外,筛选结果有助于验证计算预测。特别是在对接仿真中,模型预测和参数可以很容易地以标准化的格式发布,供实验研究人员评估。对于宿主靶向治疗,Zhou等人的研究是组合疗法的一个例子。此外,一些联合疗法的试验已经注册,包括来自这两个类别的候选药物;其中,有10种药物被纳入了回顾研究的预测。然而,这些药物要么在招募阶段,要么报告的结果有限;因此,关于其有效性的数据尚无定论。
5.1.5 专家知识
由于对COVID-19复杂的生物学机制了解有限,需要专家知识或在工作流程的某些阶段(蛋白或途径选择或药物预测过滤)进行人工治疗(图3d)。专家审查的主要目的是发现不一致或矛盾的结果,同时仍然允许识别新的预测,这对筛选候选药物名单可能的副作用是至关重要的。为了说明这一点,抗疟药(羟基)氯喹引起了人们对其潜在毒性的关注。通过对接研究发现,洗必泰可作为靶向SARS-CoV-2蛋白的潜在药物;然而,洗必泰是一种广泛使用的消毒剂,其作用机制并不特异性于SARS-CoV-2,仅被批准用于外用或牙科。因此,利用专家知识仔细评估潜在的可重复使用药物,将有助于将有限的实验和计算资源分配给具有更大广泛应用潜力的安全有效药物。因此,计算和临床研究人员之间的密切合作是至关重要的,因为计算方法仍然局限于副作用数据和药物作用靶标的注释。
5.1.6 验证策略
药物再利用研究,通常通过构建它们自己的“地面事实”,来验证计算模型;这些数据包括预测化合物的体外筛选、使用动物模型的体内实验、正在进行的临床试验、电子健康记录、文献挖掘或专家知识(图3 e)。因此,这些标准的来源有相当大的异质性,目前正在努力解决这个问题。例如,美国国立卫生研究院开放数据门户等,新发布的数据库收集并持续更新SARS-CoV-2数千种化合物的体外筛选数据和其他SARS-CoV-2相关分析数据。在此,研究者鼓励未来的研究中,在计算机模拟中,可充分利用这些资源进一步验证或过滤。然而,除了一项研究外,还没有对COVID-19的药物再利用工作进行直接的后续试验验证。然而,大量的候选药物在试验上仍然无效;因此,候选药物的系统验证,将需要提供方法准确性的景观。由于这在实践中是不可行的,因此将预测与专家知识相结合就显得尤为重要。
展望与结论
综上,在这项工作中提出的战略,有望弥补以往研究的不足,并旨在为计算药物的重新用途提供指导,以加速研究,促进标准化,并在未来的大流行情况下,做出更快更准确的反应。
参考文献
Galindez, G., Matschinske, J., Rose, T.D. et al. Lessons from the COVID-19 pandemic for advancing computational drug repurposing strategies. Nat. Comput. Sci. 2021, 1, 33–41. DOI: 10.1038/s43588-020-00007-6