
引言
计算能力的提高与快速算法的发展使得量子化学方法可以应用于生物大分子的计算。与此同时对此类大型系统的大量电子结构信息进行处理也需要新颖的技术。反应性描述符是预测和合理化化学过程的替代方法。这些描述符是简便的理论量,通过对聚合电子结构结果进行数学处理而生成。例如分子轨道(MO)系数和能量。使用最多的是概念密度泛函理论(CDFT),该理论将不同的反应性理论统一在一个理论框架中。虽然电子结构信息可用于大型生物分子,但许多具有3D结构的酶并没有关于催化过程的理论描述,与分子电子结构的独特特征有关的问题经常出现。大多数用于反应性描述符计算的方法无法用于生物大分子。
最近的研究表明:1:采用半经验量子化学方法可以准确的获得多肽的化学反应性信息;2:对描述符的正确计算需要考虑HOMO-LUMO,而不仅仅是边界轨道。3:针对大分子进行适当调整的反应性描述子能够从理论上表征某些酶的催化机理。但是流行的后处理计算化学程序并不适合于大分子的电子结构的计算。UCA-Fukui是用于计算常见CDFT描述符的专用软件,但仅限于解析GAUSSIAN程序包的输出,尚未经过大分子测试。PYGlobal是另一个用于执行这些计算的应用程序,可以同时实现多个结构的全局数量的自动化功能以及简化统计分析,但是,PYGlobal不会计算与生物结构最相关的局部描述符。为了填补这一空白,我们开发了PRIMoRDiA,一种能够准确有效地计算大分子反应性描述符的软件。当前版本可输出9个全局描述符和14个局部描述符,可以按原子、按残差或作为高斯文件输出,所有输出格式都可以通过图形化软件包来处理,该软件包旨在探索大分子的电子和反应性数据。在这项工作中,我们详细介绍了PRIMoRDiA的操作方法及主要功能。
PRIMoRDiA 方法
1.PRIMoRDiA是用C ++编写的多线程共享程序。其可计算CDFT反应性描述符并适合大型系统。运行PRIMoRDiA时会自动生成其他信息,例如总电子密度,分子轨道和分子静电势,是常用的后处理软件不能做到的。
2.PRIMoRDiA代码可以分为三个功能块。(i) 用于解析和存储来自输出文件的分子信息;(ii) 生成反应性描述符;(iii) 控制所有过程和输出脚本以进行数据可视化。
3.HOMO-LUMO Band Fukui功能。处理大分子时,PRIMoRDIA实现了两种结合前沿分子轨道的方法。第一种方法是能带密度,第二种方法根据来自HOMO-LUMO间隙的能量差,为所考虑的每个分子轨道分配权重。
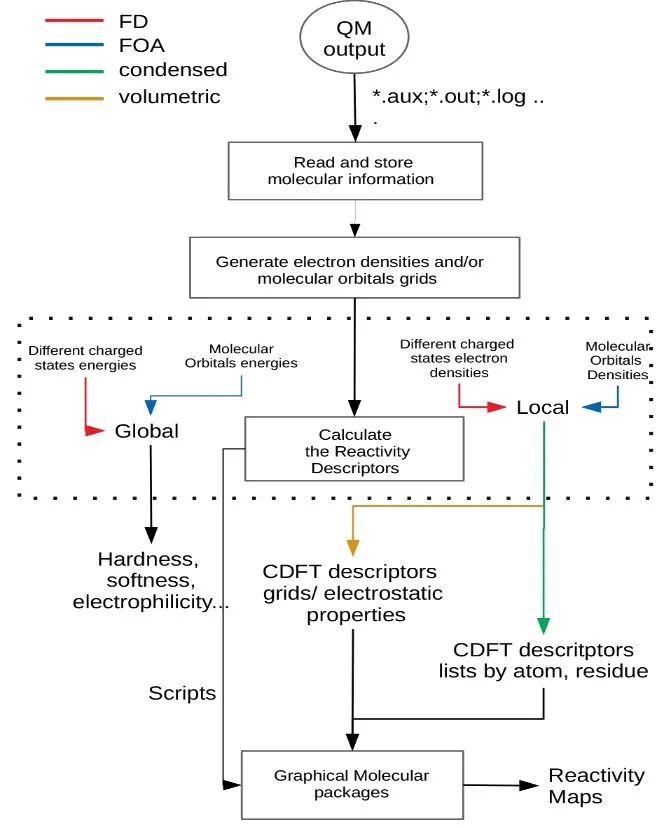
图1. 原始信息流程图及其主要特征
图片来源于JCIM
4.生物应用-酶促反应描述。为了测试PRIMoRDiA获得大型生物系统化学反应性信息的有效性,对酶促反应进行了预测。从MACIE数据库检索了参考数据,MACIE数据库是一个致力于对已发布的酶催化机制数据进行分类的网站。使用半经验哈密顿量和线性比例缩放算法结合量子化学计算,获得了描述符计算所需的电子结构,但是,后续的分子动力学等可能会增加总体计算时间,是PRIMoRDiA大规模应用的瓶颈。为了验证是否可以使用原始晶体学数据,生成了由不同处理级别(称为L1,L2和L3)产生的几何形状,这些几何形状具有高的复杂性和计算成本,用于电子结构计算中,随后是描述符计算。见图2。图3显示了描述符根据MACIE正确分类残基次数的百分比,每种静电稳定剂的正确分类都适用于L1以外的任何预处理,但是随着初级处理中复杂程度的提高,对其他类别的预测也逐渐变差。
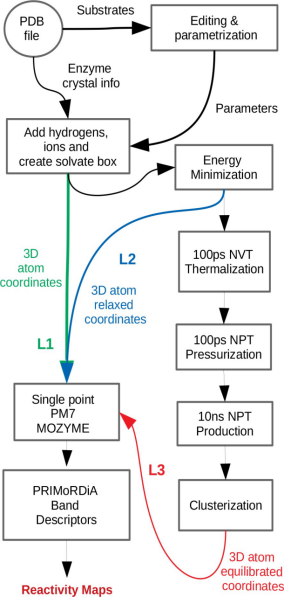
图2. 模拟流程图
图片来源于JCIM
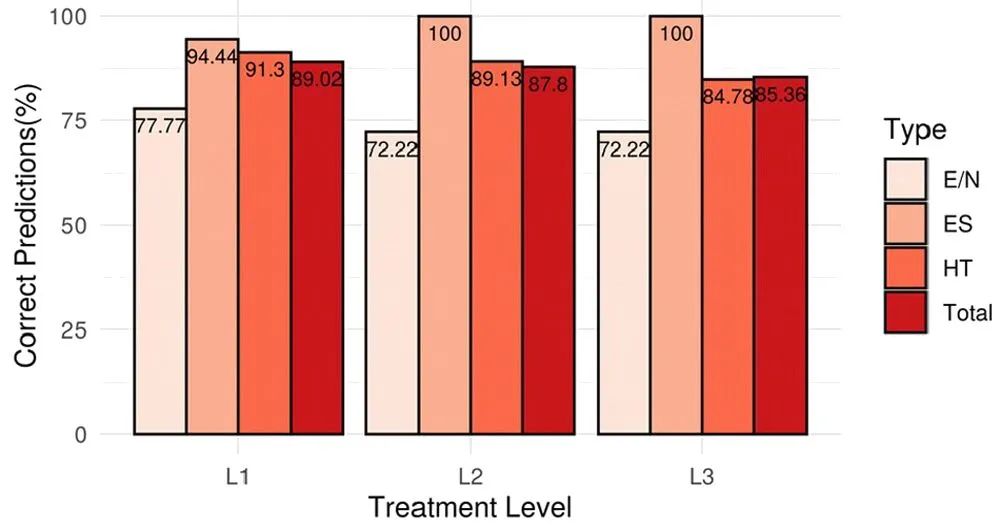
图3. 分类残基次数的准确性百分比
图片来源于JCIM
5.PRIMoRDiA还可生成脚本。这些脚本使用Pymol等图形软件包可生成有价值的信息和清晰的图形。这些图像将PRIMoRDiA处理的数学和数值数据转换为图形,可轻松一目了然地传达相关的化学信息。在大多数情况下,用分子动力学模拟优化结构会损害描述子的化学反应性预测,对蛋白质进行晶体学分析发现其活性位点具有过渡态,这表明,只要活性物质的精细结构,整个蛋白质的大规模运动可能与酶的催化特性无关。如图4所示。
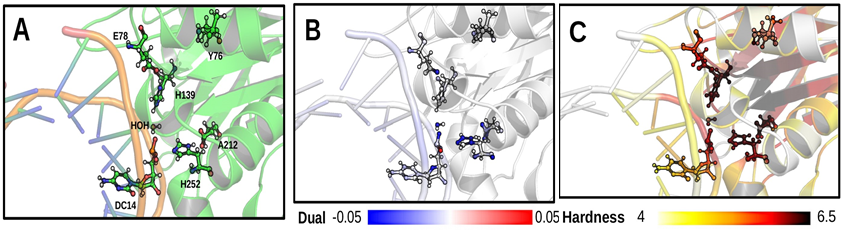
图4. L3处理水平的1dnk结构的反应图
图片来源于JCIM
6.SARS-CoV-2主要蛋白酶研究应用程序。 使用L1预处理对SARS-CoV-2主蛋白酶(PDB Code: 6LU7)进行研究,以查看其存在如何改变位点的反应性。在图5A,B中,残基CYS143易受亲核攻击,并且确实是在蛋白酶的第一催化步骤中后与配体反应的残基结合。HIS41失去亲核性,在配体结合后反应性降低,从质子供体变为质子受体。图5C,D中的局部图表明,静电稳定主要由残基HIS163和HIS164的主链完成。HIS41和CYS145中原子的局部硬度也略有增加,可能作为辅助来稳定系统。
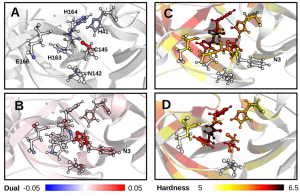
图5. SARS-CoV-2主要蛋白酶的反应性描述符
图片来源于JCIM
总结
本文介绍了PRIMoRDiA软件,通过使用CDFT反应性描述符,适当的计算方法以及脚本的自动生成,以清晰,高质量的方式呈现信息来有效地将大分子的电子结构转换为有用的和可表示的化学信息。此软件有望使用更低得成本研究大型系统中的化学反应性。除此之外,该软件是功能强大的解析器,能够从电子结构中检索信息。
软件下载地址
https://github.com/igorChem/PRIMoRDiA1.0v
参考文献
Igor Barden Grillo, Gabriel A. Urquiza-Carvalho, and Gerd Bruno Rocha, PRIMoRDiA: A Software to Explore Reactivity and Electronic Structure in Large Biomolecules, J. Chem. Inf. Model. 2020, 60, 12, 5885-5890. DOI: 10.1021/acs.jcim.0c00655.