

RealVS是一种可以显著提高top hits的精度的新方法,并能学习与生物活性相关的可解释的关键子结构。
背景介绍
虚拟筛选(VS)是一种用于药物发现的计算技术,可用于搜索最可能与药物靶点(如蛋白质受体或酶)结合的小分子库。VS大致分为两大类,即基于结构的(SBVS)和基于配体的(LBVS)。
SBVS将候选配体与目标蛋白对接并利用评分函数来估计配体与高亲和力蛋白结合的可能性,这一方法已经在许多工作中得到了成功应用。然而在大多数现实场景中,仅依靠SBVS无法获得满意的结果,需要了解蛋白质靶点的完整或局部三维结构。相比之下,基于配体的虚拟筛选(LBVS)不需要知道蛋白质靶点的三维结构,可以作为一种重要的虚拟筛选手段。同时,LBVS可以充分利用丰富的已知生物活性的配体样品,在大型化合物数据库中构建优良的药物先导物虚拟筛选模型。LBVS方法通常可分为三类,包括基于相似度的、基于药效团的和基于机器学习的方法,其中以基于机器学习的方法最为流行,在药物设计中得到了广泛的应用。
基于机器学习的LBVS方法的大体策略都是从一个庞大的数据库中精确预测所有化合物的生物活性。但是实验工作者比起关注整个化合物库的预测结果,更倾向于从大型数据库中预测的生物活性排名前k位的化合物中进行选择,以继续进行湿法药物发现实验。在药物先导物的虚拟筛选中,top-k精度的显著提高比传统LBVS方案更有价值。但目前,还没有专门关于这方面的研究。
主要内容
本文中,来自南京邮电大学的吴建盛团队提出了一种新方法–RealVS,可以显著提高从大型化合物数据库中进行药物先导物虚拟筛选的top-k精度。RealVS的特点如下:(1)通过迁移学习,从源域引入丰富的训练样本,弥补与药物靶点相关的非活性配体的不足;(2)采用自适应域对齐拟合训练模型所用化合物生成特征的分布与筛选化合物生成特征的分布,确保训练模型具有更强的泛化能力;(3)提出一种新的目标函数,同时优化分类损失、回归损失和对抗损失,通过优化分类损失可以筛选出大多数非活性配体;(4)引入图注意力网络,学习与配体生物活性相关的关键亚结构,提高模型的可解释性。大量基准数据集的结果表明,该方法在针对大型化合物数据库中的LBVS虚拟命中里显著提高了不同k值下的top hits精度,在实际应用中具有重要价值。相关的研究成果以“RealVS: Toward Enhancing the Precision of Top Hits in Ligand-Based Virtual Screening of Drug Leads from Large Compound Databases”为题发布在国际著名期刊Journal of Chemical Information and Modeling上。
RealVS网址
http://noveldelta.com/RealVS
网站首页:
模型组成
研究者提出的RealVS模型包含四个模块,如图1所示,即(1)图关注模块G;(2)回归模块R;(3)分类模块C;以及(4)适应模块A。回归模块R和分类模块C构成了一个筛选调节器,以筛选出无活性的配体,重点预测活性配体的活性。
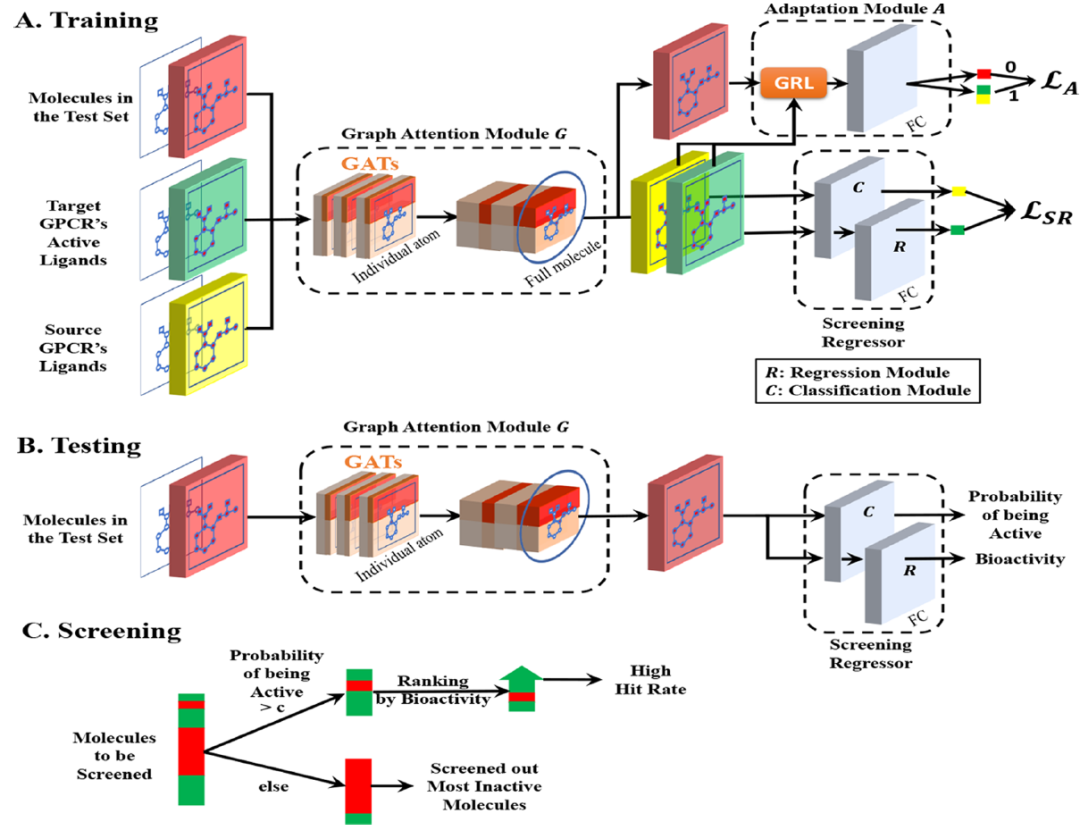
图 1. 本文所提出的RealVS的架构。图片来源于JCIM
与基于图分子预测方法的比较
研究者将结果与基于图分子预测方法进行了比较,包括图卷积网络(GCN)、图注意网络(GAT)、利用监督学习和语境预测预训练的图同构网络(GIN)、神经指纹(Neural FP)、Weave、消息传递神经网络(MPNN)、加权深度学习和随机森林(WDL-RF)、以及Attentive FP。
如表1所示,在所有数据集和k值上,RealVS均取得了最好的性能,对前5%,平均精度提高了15.27% (RealVS, 52.84%;第二名,45.85%),对前15%,平均精度提高了25.17% (RealVS, 62.46%;第二名49.89%),前25%则提高了30.88% (RealVS, 71.58%;第二名,54.69%)。特别是top-k精度的显著提高(提高32.55%;RealVS,60.64%;第二名,45.75%)的结果表明,即使在小的训练样本量下,RealVS仍然保持着较高的top-k精度。
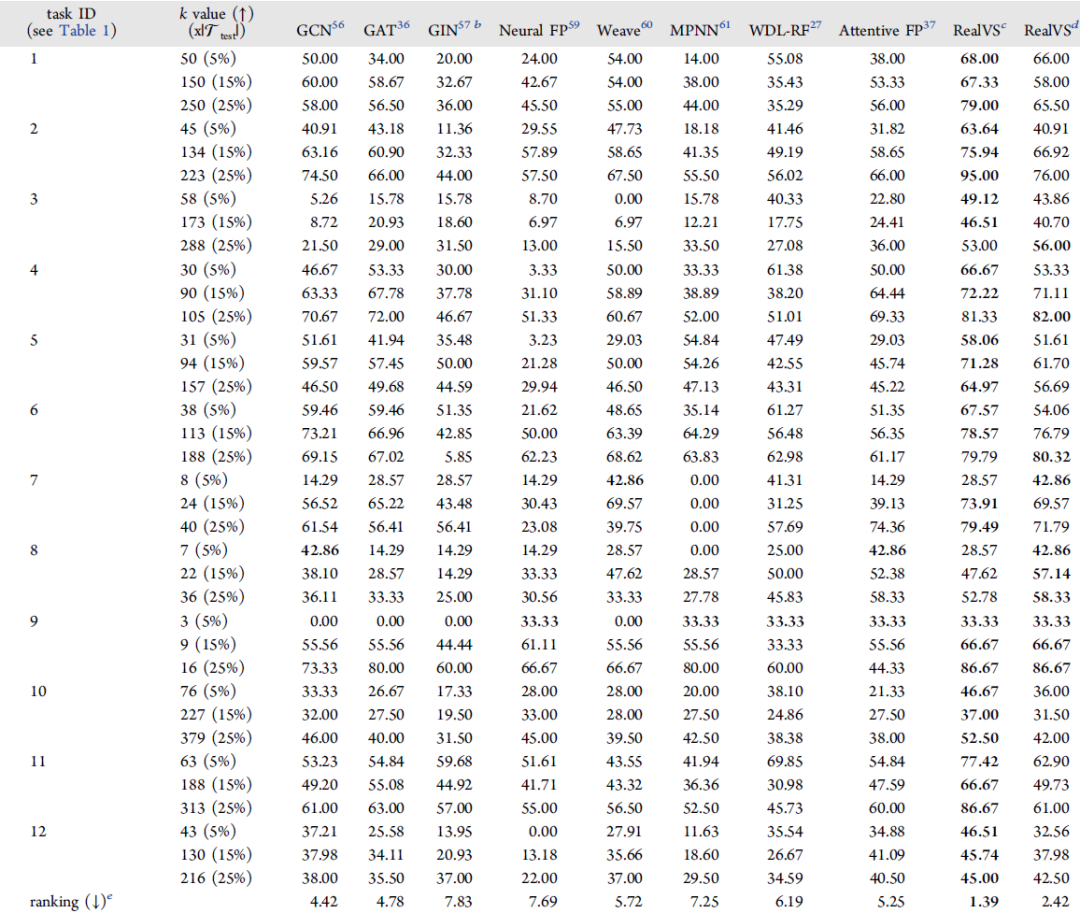
表 1. 5倍交叉验证下不同靶标GPCRs的Top-k精度和召回率比较。表格来源于JCIM
特征可视化与解释
与此同时,研究者对靶向高活性配体和筛选出非活性配体的能力进行了探索。在测试集中,研究者分别为人体溶脂、大麻素和腺苷受体选择了三种高活性配体和非活性配体(见图2和图3)。结果如下:
(1) RealVS更关注最有可能与靶标结合的原子,因此,预测高活性配体的等级较高。例如,RealVS将注意力集中在可能通过提供氢键与人溶质受体Q99500结合的羟基(“-OH”)上(见图2a);RealVS更关注氯离子(“Cl-”),氯离子可能通过提供阴离子与人大麻素受体P34972结合(见图2b);RealVS更关注“N”原子附近的“C”原子,该原子很可能与人腺苷受体P29274结合(见图2c)。
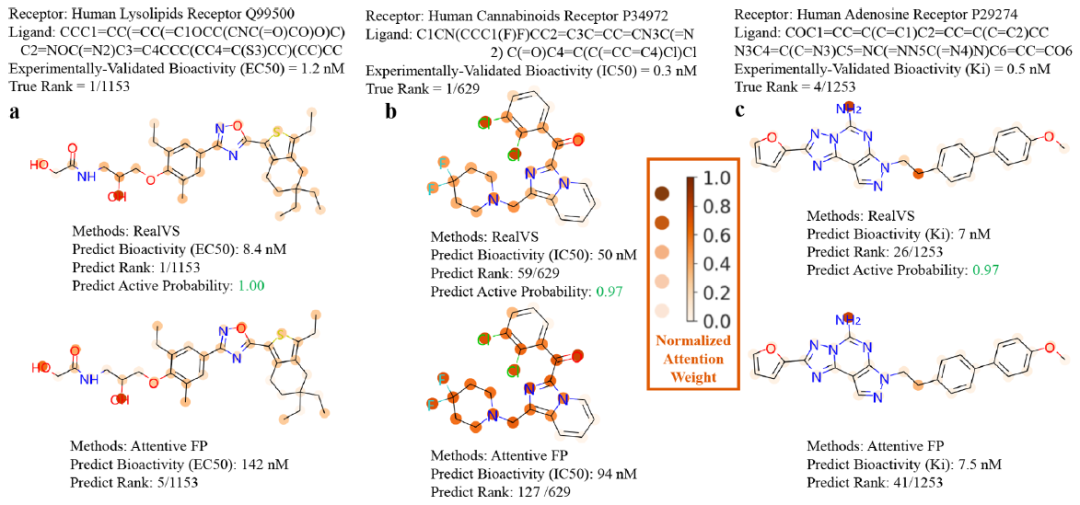
图 2. RealVS命中高活性配体能力的解释。图片来源于JCIM
(2) RealVS将注意力从看起来像但不会键合的原子转移,因此,即使回归模块预测高生物活性,分类模块也可以筛选出非活性配体。例如,RealVS将注意力从不容易与人类溶脂受体Q99500结合的“O”原子转移(见图3a);RealVS将其注意力转移到苯环外的所有原子,猜测配体可能不会与人体大麻素受体P34972结合(见图3b);RealVS将注意力从亚氨基(“-NH-”)和“O”原子转移,它们看起来像人的腺苷受体P29274,但不会结合(见图3c)。
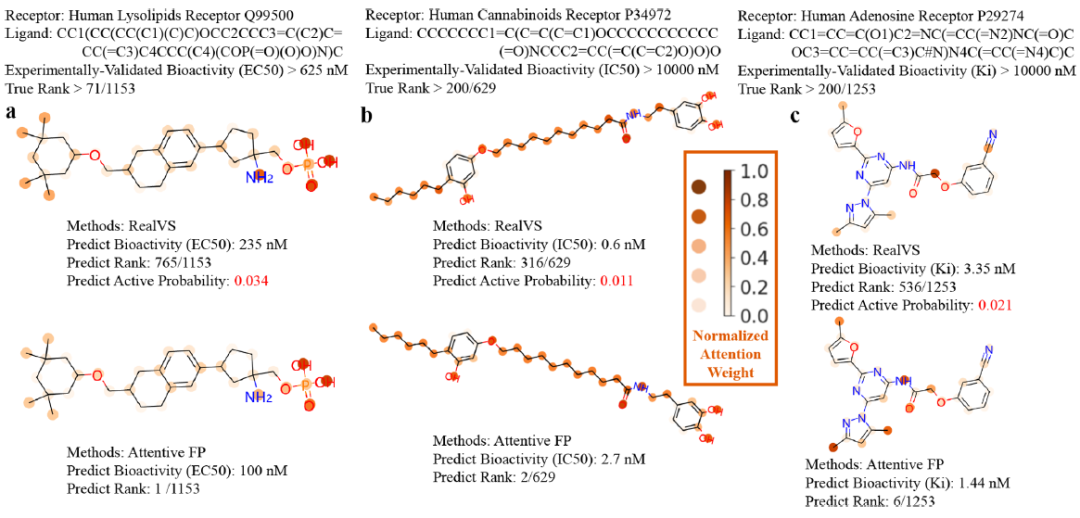
图 3. RealVS筛选非活性配体能力的解释。图片来源于JCIM
在PDBbind v2016中的研究
为了进一步验证RealVS的算法性能,研究者在PDBbind v2016基准数据集上进行了实验。PDBbind v2016数据集包含了114344个实验确定的结构和16179条实验确定的结合亲和力数据(Kd, Ki和IC50)。采用默认设置,将精炼集作为训练集,将核心集作为测试集。核心集包含聚集在57类蛋白质中的285个蛋白-配体结合亲和性数据。研究者使用了791个配体的SMILES和精炼集(不包括核心集)中同类型蛋白质的蛋白-配体结合亲和力作为训练集。
为了更好的比较,将活性最高的配体数量排在第一位(top-1 hit),并对所有57个数据集取Pearson相关系数(R)平均值来评估算法(图4a和4b)。可以看出,RealVS,在R和top-1 hit方面都优于其他方法,尤其是R指数(与基于配体的共识结果0.747相比),显示出了出色的预测能力。
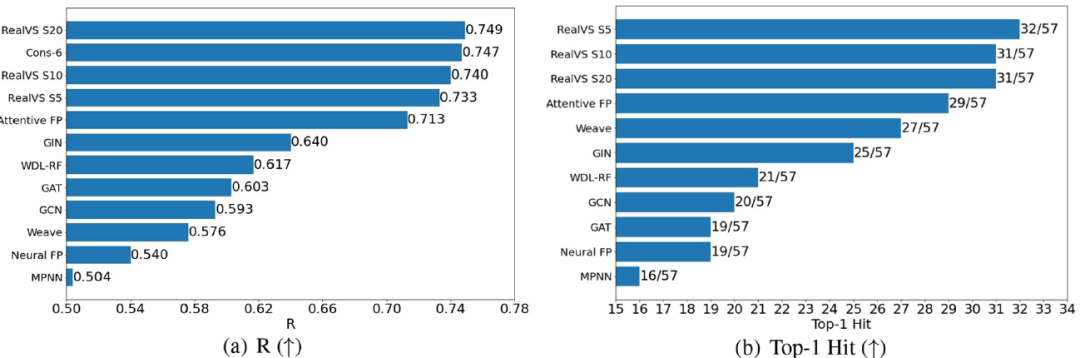
图4. 5倍交叉验证下PDBbind v2016精练集和核心集上R与Top-1 Hit的比较。图来源于JCIM
结论总结
本篇文章提出了一种新方法,RealVS,可以显著提高top hits的精度,并学习与化合物生物活性相关的可解释的关键子结构。
RealVS的动机包括以下几点:首先,通过从源结构域转移丰富的信息,减少了与药物靶点相关的非活性配体的需求。随后,采用域对齐方法拟合训练数据集和筛选数据库中化合物生成特征的分布,以获得较好的模型泛化能力。接下来,研究者提出了一个新的目标函数,同时优化分类损失、回归损失和对抗损失,其中大多数非活性配体往往被筛选出活性预测。最后,研究者采用图注意力网络来学习与配体生物活性相关的关键子结构,以提高模型的可解释性。大量基准数据集的结果表明,RealVS在药物先导物虚拟筛选的各种设置下,均显著提高了top-k精度(平均提高32.64%)。
参考文献
Yin, Yueming, et al. “RealVS: Toward Enhancing the Precision of Top Hits in Ligand-Based Virtual Screening of Drug Leads from Large Compound Databases.” J. Chem. Inf. Model. 2021, 61, 10, 4924–4939. https://doi.org/10.1021/acs.jcim.1c01021