一种基于几何结构软件和深度学习的新框架DeepPocket,用3D卷积神经网络对Fpocket识别的口袋重新评分后能发现蛋白表面的空腔。
背景介绍
基于结构的药物设计(SBDD)的关键步骤,是识别和验证受体靶点。一旦受体位点被识别出来,就可以设计出能很好地与靶标结合的小分子,并显示出所需的药理效果。预测配体分子在蛋白结构上的结合位置,是药物设计过程中必不可少的一步。这需要开发高度精确能够从给定的受体三维结构中检测配体结合位点的计算机模拟算法。
深度学习可以从数据中构建复杂的关系,这是传统机器学习无法比拟的。深度学习模型基于概念层次原理,将相互连接的神经元层堆叠起来,即通过从简单概念构建复杂概念来学习复杂概念。这些算法已被证明在计算机视觉和自然语言处理方面取得了巨大的进步。例如,卷积神经网络(CNN)在图像识别方面表现出了最先进的性能。
结合位点检测,可以通过三维蛋白质结构的体素化建模转化为计算机视觉问题。这样就可以在相同的任务中使用这些CNN。DeeplyTough是一种基于CNN的孪生神经网络,通过将口袋编码成描述向量,使用欧几里得距离来比较口袋的方法。DeepSite采用了类似于P2Rank的方法,它使用CNN对蛋白表面的所有点进行评分,并对所有得分高的点进行聚类,生成候选结合口袋。Kalasanty将整个蛋白结构通过一个受U-Net启发的基于CNN的细化模型,一步生成预测的结合位点。它赋予每个体素作为口袋一部分的概率。在结合位点检测方面,它的性能优于DeepSite。
主要内容
基于结构的药物设计包括潜在药物分子的开发,这些配体与给定受体的结合位点形成稳定的复合物。这样做的前提是,在蛋白质的3D结构上找到和功能性相关的结合位点。虽然已有多种检测结合位点的方法,但大多数方法在结合位点的准确识别和排序上都失败了。深度学习算法,在结构生物学各个领域的快速应用和成功,预示着这种算法可用于精确的结合位点检测。为此,来自印度国际信息技术研究所的U. Deva Priyakumar等人报告了基于几何结构软件和深度学习相结合的新框架DeepPocket。除此之外,研究者还提出了另一组包含蛋白质结构的数据集SC6K,用于配体结合位点(ligand binding site, LBS)的检测,该数据集于2018年1月1日至2020年2月28日提交到蛋白质数据库(PDB)。DeepPocket对各种结合位点数据集和SC6K的测试结果表明,它比目前最先进的方法具有更好的性能,对新结构具有良好的泛化能力。相关的研究成果以 “IDeepPocket: Ligand Binding Site Detection and Segmentation using 3D Convolutional Neural Networks” 为题发布在国际著名期刊Journal of Chemical information and Modeling上。
方法概述
DeepPocket是一个新的且全面的框架,能高效检测蛋白质的三维结构中的结合位点。研究者采用多步骤方法从输入的蛋白质结构中,获得最终的口袋位置和三维形状预测。首先,通过Biopython库从蛋白质结构中去除所有杂原子和溶剂分子来处理输入结构。然后,在结构上运行Fpocket,并计算每个预测口袋的中心。这些中心需要由CNN评分功能进行排序,再成为候选口袋中心。因此,在每个中心处放置固定大小的网格,然后使用CNN进行评分。最后,将排名靠前的中心通过CNN细分模型发送到最终的口袋结构。图1给出了研究者方法的pipeline。
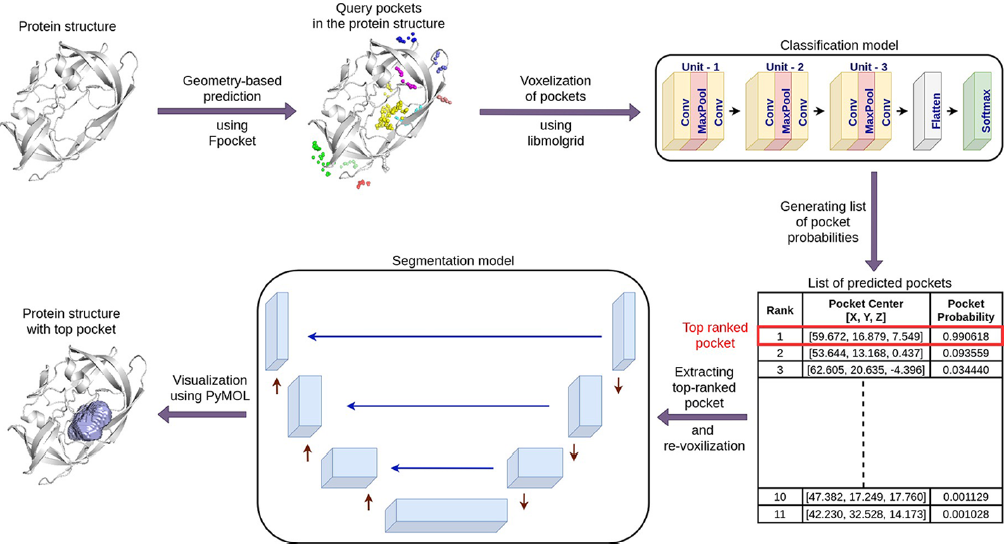
图 1. DeepPocket的使用流程。图片来源于JCIM
测试结果
为了对DeepPocket的泛化能力进行综合评价和测试,研究者在scPDB v.2017数据库上对分类和细分模型进行了10倍交叉验证,然后在COACH420、HOLO4k和SC6K数据集上进行了测试。对细分模型进行测试,重点是与目前最先进的口袋三维形状预测方法Kalasanty进行对比。最后,研究者验证了DeepPocket使用refined set在已建立的基准上识别结合位点残基的能力。
交叉验证,分类实验通过相应的交叉验证细分训练得到的10个分类模型的平均验证accuracy为0.943,AUC-ROC为0.966。Top-n和Top-(n + 2)验证集上10个模型的平均召回率分别为70.27%和87.77%。图2中绘制了从Top-n到Top-(n + 7)的验证集的成功率。研究者认为,从Top-n到Top-(n + 2)的17%的成功率,可能是数据集中尚未注释的推定或隐藏的结合位点的存在。此外还看到数据集中的大多数口袋都被预测在Top-(n + 7)排名中。
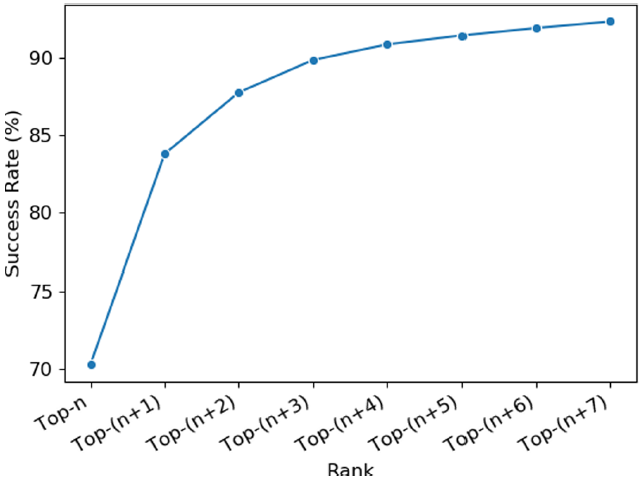
图 2. 在scPDB v.2017数据集中正确识别从Top-n到Top-(n + 7)级别的口袋的平均成功率。图片来源于JCIM
研究者在三个测试数据集COACH420、HOLO4k和SC6K上,比较了DeepPocket与其他最先进的方法在识别结合空腔方面的性能。Top-n和Top-(n + 2)的成功率结果见表1。除了COACH420的Top-n得分,DeepPocket在所有数据集上都优于其他所有最先进的方法。DeepPocket也是唯一一种不会在数据集中提供任何蛋白质口袋位置的深度学习方法。DeepPocket成功地将Fpocket检测到的85%的结合位点,排在了Top-n的排名中。
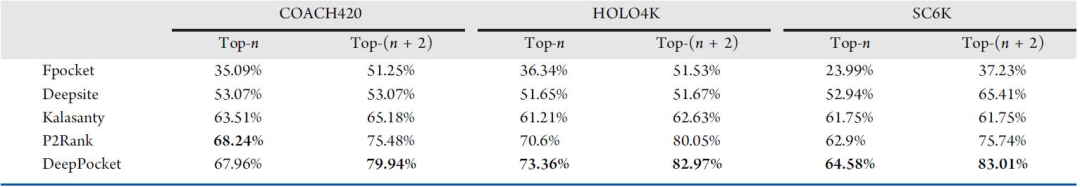
表1. DCA(与配体任一原子的距离)结果比较。表格来源于JCIM
表2报告了4Å阈值下的DCC(到结合位点中心的距离)成功率和两种方法的平均DVO(离散体积重叠)值。在DCC和DVO标准上,DeepPocket的表现都优于Kalasanty。DeepPocket在COACH420和SC6K上实现了惊人的DCC成功率,分别为81.31%和80.24%,这表明大多数预测口袋形状的中心位于口袋的真中心附近,而在HOLO4K数据集上,成功率较低(65.79%),但仍然相对较好。在对三个数据集进行测试时,DeepPocket也返回了大于0.6的平均DVOs,这表明了良好的细分精度。
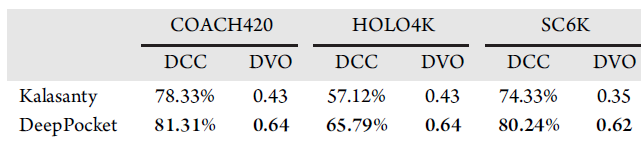
表2. DCC和DVO结果比较。表格来源于JCIM
三组数据的成功率曲线和DVO分布图,如图3所示。DeepPocket在COACH420和SC6K数据集上的成功率图与交叉验证实验中的成功率图非常相似,表明了良好的泛化性。这些结果可以得出结论,Kalasanty方法的优势——在结合位点的局部生成密度(表1),而DeepPocket的细分算法对整个结合口袋形状的解释相对较好。对HIV蛋白酶的位点检测案例显示,分类模型给出的排名第一的口袋中心和晶体结构中的一致,说明预测正确。
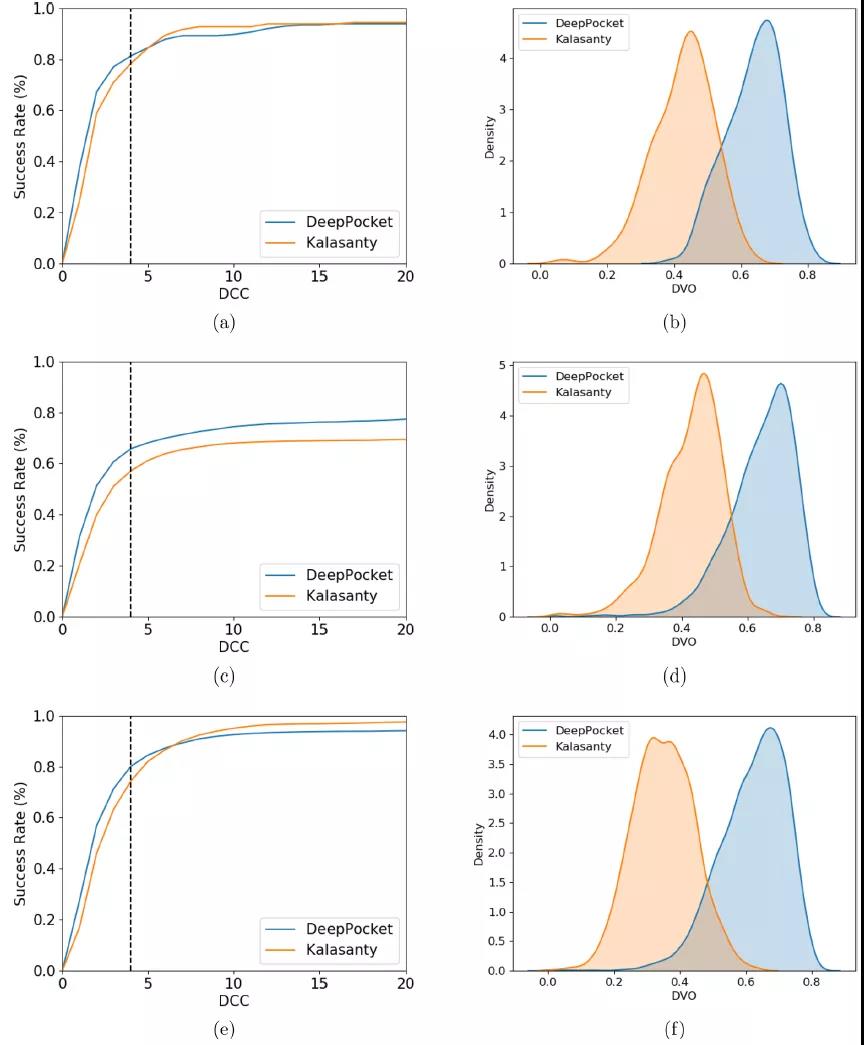
图3. DeepPocket和Kalasanty对测试数据集的细分结果。图片来自JCIM
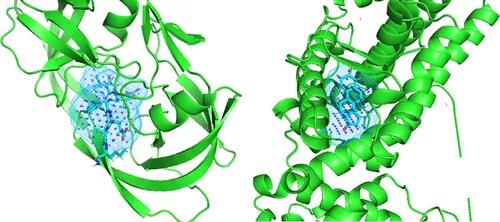
图4. DeepPocket检测到的HIV-1蛋白酶(PDB ID: 1K2C)和孕酮配体结合域(PDB ID: 1SQN)的顶部和细分口袋(“浅蓝色”体积)。图片来自JCIM
结论总结
本文介绍了一种方法DeepPocket,它遵循多步骤方法来识别蛋白质三维结构上的配体结合位点。主要包括三个步骤,提取候选口袋中心,使用3D CNNs分类模型对它们进行排序,最后使用细分模型对排名靠前的中心进行形状说明。DeepPocket的一个额外的优势是不会在任何提供的蛋白质结构上出错。因此,将DeepPocket纳入需要识别结合空腔的结构生物信息学和药物设计pipeline中将大有裨益。本文源代码地址:https://github.com/devalab/DeepPocket
参考文献
Rishal Aggarwal, Akash Gupta, Vineeth Chelur, C. V. Jawahar, and U. Deva Priyakumar, DeepPocket: Ligand Binding Site Detection and Segmentation using 3D Convolutional Neural Networks, Journal of Chemical information and Modeling, Article ASAP. DOI: 10.1021/acs.jcim.1c00799