主要内容
近两年来,新型乙型冠状病毒SARS-CoV-2在全球迅速蔓延,在不到100天内确诊病例超过150万例。高感染率给全球卫生系统造成了相当大的压力,截至撰写本文时,已导致2.04多亿人感染,430多万人死亡。
SARS-CoV-2大流行,促使人们开始在全球范围内寻找治疗方法,药物再利用是加速这一过程的主要策略之一,因为有希望的药物可能通过使用已批准的安全药物,直接进入II期及以后的临床研究。撰写此文时(2020年初),大多数SARS-CoV-2体外抑制研究依赖于高通量筛选(HTS)研究来测试特定FDA批准的药物,这些药物和化合物此前对不同的β-冠状病毒或特定的抗病毒靶点显示出抑制作用。此后,已在Huh7细胞中大规模筛选了1425种化合物,确定了11种活性IC50 < 1 μM的分子。许多化合物已经进行了体外测试,因而获得了一个不断增长的体外抗病毒活性分子数据库。机器学习模型,可以根据先前公布的数据预测最佳化合物,从而促进药物发现。来自美国Collaborations Pharmaceuticals Inc的Sean Ekins等人采用了几种机器学习方法,从最近的SARS-CoV-2体外抑制数据中开发预测模型,并使用它们从研究者的内部化合物库中选择FDA批准的化合物进行体外测试。从贝叶斯机器学习模型预测的化合物中,抗疟药lummefantrine被选中进行测试,并在基于细胞的检测中显示有限的抗病毒活性,同时通过微尺度热电泳显示与刺突蛋白结合(Kd =259nM)。这种机器学习和体外检测相结合的方法,可以扩展到用虚拟筛选来寻找对SARS-CoV-2参考WIV04毒株有活性的化合物。相关的研究成果以 “Machine Learning Models Identify Inhibitors of SARS-CoV-2” 为题发布在国际著名期刊Journal of Chemical Information and Modeling上。
与SARS-CoV-2刺突蛋白对接
研究者利用Discovery Studio (Biovia, San Diego CA),基于COVID-2 Spike受体结合域(RBD)和血管紧张素转换酶2 (ACE2)之间的晶体结构界面,选择了一个区域进行对接。使用CDOCKER在该界面的受体腔生成的对接位点(半径为9.7 Å)内,通过刚性对接在该界面生成lumefantrine的多个构象。对接参数设置为默认值(保留前10 的构象)。在配体最小化后,计算了化合物与受体之间的配体相互作用能。最小算法为“Smart Minimizer”,最大步长为1000步,最小均方根梯度为0.001,静电球形截止距离为12 Å。
数据管理
体外SARS-CoV-2数据,从5项药物再利用研究中经过初步整理,得到了包含63个分子的数据集,平均活性为15.94±22.45 μM。从不同研究中整理的外部测试集有30个分子,平均活性为34±42 μM。使用AC生成的贝叶斯模型对活性进行分类的阈值为6.65 μM,最终训练集的活性化合物比例为52%,外部测试集为37%。数据集的活性分布如图1所示。
图1. SARS-CoV-2数据集的常见活性/非活性结构特征。图片来源于JCIM
机器学习模型
本文涉及的机器学习模型是用AC以及其他几种可用的机器学习方法开发的。五折交叉验证比较结果显示仅使用训练数据实现的所有机器学习算法的不同预测统计量(表1)。在相同阈值6.65 μM下,AC优于所有其他方法,Rf紧随其后。这些机器学习模型可用于外部验证。
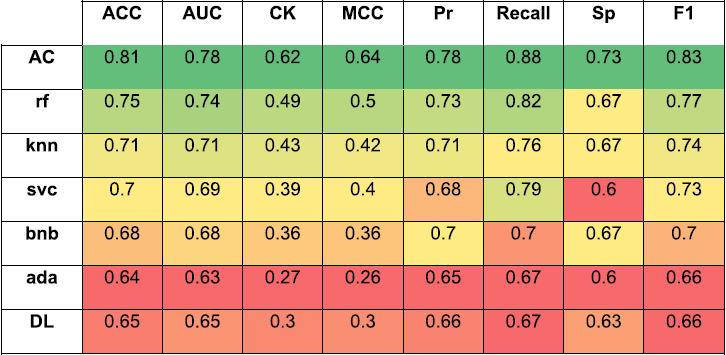
表1. 使用ECFP6指纹实现的所有SARS-CoV-2机器学习模型的五倍交叉验证统计。表格来源于JCIM
外部验证
机器学习模型对外部测试数据的性能,如表2所示。外部验证使用来自训练集之外的不同研究的数据来衡量模型性能。与其他机器学习模型相比,SVC和KNN的总体统计数据略好,在召回率和特异性之间达到了最佳平衡。
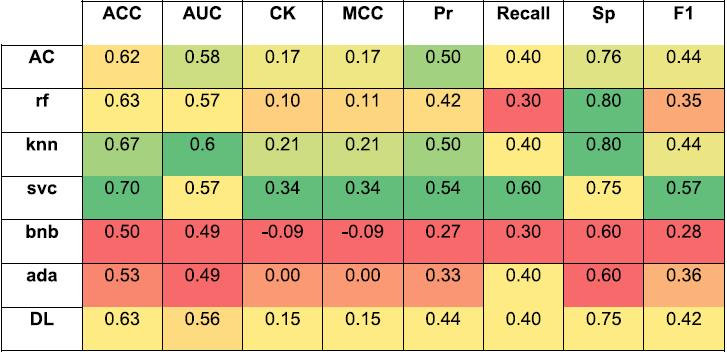
表2. 所有SARS-CoV-2机器学习模型的外部数据预测统计。表格来源于JCIM
化学空间
单独对模型训练集的主成分分析表明,使用分子指纹描述符分析时,SARS-CoV-2化学空间分布良好,活性分子和非活性分子混合良好(图2)。与主要由FDA批准的药物组成的Prestwick化学库(PwCL)相比,SARS-CoV-2数据属于具有分子描述符的大聚类,在使用指纹描述符时分布更为广泛(图2C,D)。
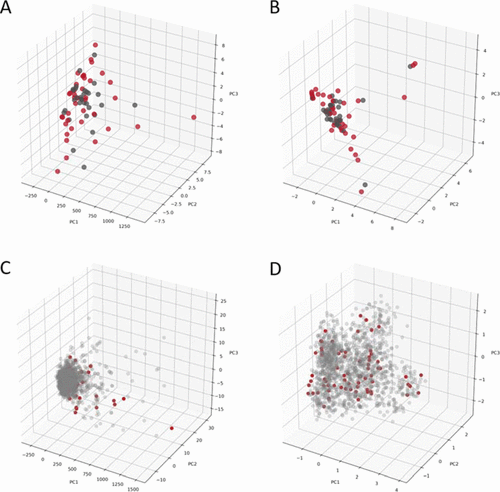
图 2. 具有分子描述符(A)和ECFP6的SARS-CoV-2集合的PCA (B)。红色球体有活性,灰色球体无活性。SARS-CoV-2集合和PwCL的PCA与分子描述符(C)和ECFP6 (D)。红色球体- SARS-CoV-2,灰色球体- PwCL。图片来源于JCIM
化合物的抗病毒活性测定
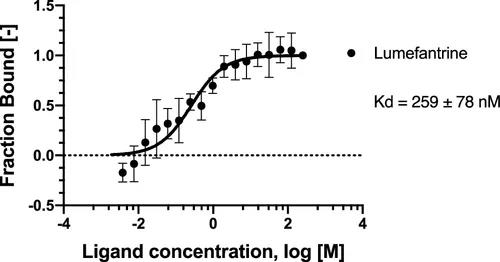
最初选择lummefantrine,是因为它是一种广泛可用的抗疟药。作者在Vero 76、Calu-3和Caco-2细胞中进行了测试。与细胞毒性相比,每个细胞系的IC50或EC90数据并不表明有用的体外活性(表3)。然而,Vero 76中性红分析数据显示,EC50远低于CC50。Budesonide、tiamulin fumarate和tetrabenazine也在Caco-2细胞中进行了测试,并显示了与细胞毒性相当的抑制作用。Tiamulin的EC90低于CC50。MST 测量lummefantrine对 SARS-Cov2 Spike RBD 蛋白的结合亲和力,结果如图3所示。
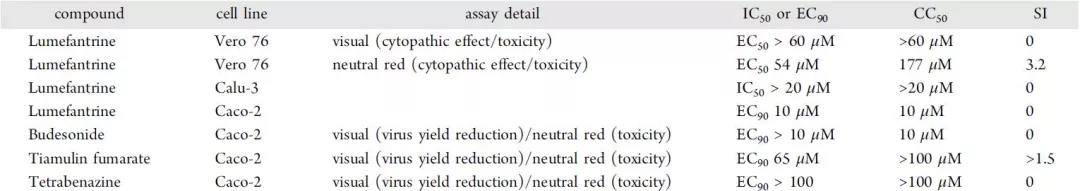 表3. Lumefantrine Vero 76、Calu-3和Caco-2的IC50、EC90和CC50值。表格来源于JCIM
表3. Lumefantrine Vero 76、Calu-3和Caco-2的IC50、EC90和CC50值。表格来源于JCIM
图3. Spike RBD与lummefantrine相互作用的MST结合分析。图片来自Journal of Chemical Information and Modeling
结论总结
本文证明了作者提出的机器学习模型,在内部交叉验证和外部验证中表现良好。机器学习使人们能够找到自己或他人验证的SARS-CoV-2的活性分子。这些机器学习模型还可以用来优先考虑未来的化合物,这些化合物的预测得分和可靠性都很高。这将有望反馈更可靠的预测,同时结合药物发现专业知识便可以帮助确定体外测试的化合物的优先级。
参考文献
Victor O. Gawriljuk, Phyo Phyo Kyaw Zin, Ana C. Puhl, Kimberley M. Zorn, Daniel H. Foil, Thomas R. Lane, Brett Hurst, Tatyana Almeida Tavella, Fabio Trindade Maranhão Costa, Premkumar Lakshmanane, Jean Bernatchez, Andre S. Godoy, Glaucius Oliva, Jair L. Siqueira-Neto, Peter B. Madrid, and Sean Ekins, Machine Learning Models Identify Inhibitors of SARS-CoV-2, Journal of Chemical Information and Modeling Article ASAP. DOI: 10.1021/acs.jcim.1c00683