引言
核酸(NA)在各种细胞过程中起着重要作用,是抗癌,抗病毒及抗菌领域中许多疾病的潜在药物靶标。DNA双链体、DNA G-四链体、RNA核糖开关以及mRNA中的延长重复序列这些优秀的靶标引起了人们对作用于核酸的药物的极大关注,与核酸有关的生物学及其药物化学研究深入详实。然而,在计算化学领域,由于在电荷,结合口袋和溶剂化效应等生化和物理性质上与小分子蛋白对接存在差异,导致小分子核酸对接的可用程序不多。因此,为更好地发展核酸对接领域,本文介绍了现有的核酸-配体对接程序及性能,并讨论了核酸-配体对接面临的五大挑战。

核酸与小分子是怎样结合的?
小分子与核酸之间的结合可以是共价的或非共价的。如图1所示,一般认为核酸-配体有三种相互作用模式:1)静电相互作用,2)沟结合,3)插层和π堆积。
静电相互作用
设计与核酸稳定结合的小分子时,通常会考虑静电相互作用。该方法旨在中和带负电的糖-磷酸骨架,带正电的离子(Mg2+,Ca2+)以及聚集在骨架周围的多胺。纯净的静电相互作用可利用核酸中大量的氢键供体和受体来补充氢键网络,从而利用该氢键网络来设计序列特异性的结合剂。
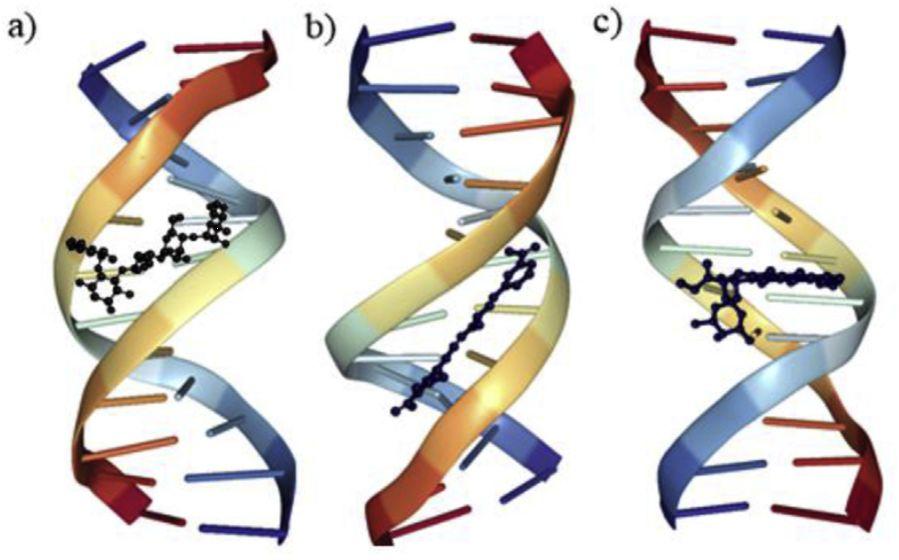
图1.核酸与配体的三种非共价结合模式:a)静电相互作用,b)沟结合,c)插层。
图片来源:EJMC
沟结合
Watson-Crick碱基对的整体几何结构导致在DNA和RNA螺旋内会形成大沟槽和小沟槽。通常,DNA的大沟比RNA的大沟更浅更宽因此有较多的氢键受供体位点,也为更大的分子(蛋白质和肽)提供了更大的口袋。DNA的小沟相对RNA的小沟更浅而窄,常用来小分子药物在水置换后的结合。DNA小沟结合剂被设计成抗癌药和抗生素药物。RNA的小沟与DNA相比有较低的静电势,但小分子通常与具有较大静电势的RNA大沟结合。单链RNA具有强聚阴离子的特性而被金属正离子和水分子包围,配体需具有大极性来破坏水合作用壳。RNA自身折叠的特性会形成如发夹和凸起等二级结构进而丰富了RNA的三级结构,最终形成多样的独特的结合口袋,能有选择性地结合小分子。
插层和π堆积
插层是指因在堆积的碱基对之间插入多芳香族分子从而破坏其功能的结合模式。插层剂在临床上用作靶向DNA的抗癌药。靶向DNA G-四链体的药物通过π-π堆积于DNA末端或附近结合,小分子在结合过程中对四链体的构象变化影响较小。而插层作用首先干扰核酸之间的内部π-π相互作用,会导致DNA大规模运动。
截止2018年6月,PDB数据库中收集了分子量介于70~1000Da之间的465个RNA-配体复合物和458个DNA复合物。这使基于结构的药物设计方法用于设计针对特定疾病的靶向核酸配体成为可能。
核酸分子对接的性能评估研究
分子对接可用于虚拟筛选得到潜在候选药物和提供配体与特定结合位点之间的相互作用信息。通常,如图2所示,分子对接可以分为两个部分:a)构象搜索,生成配体的几种结合模式,b)评分,应用打分函数估算结合自由能并选择有利的结合模式。
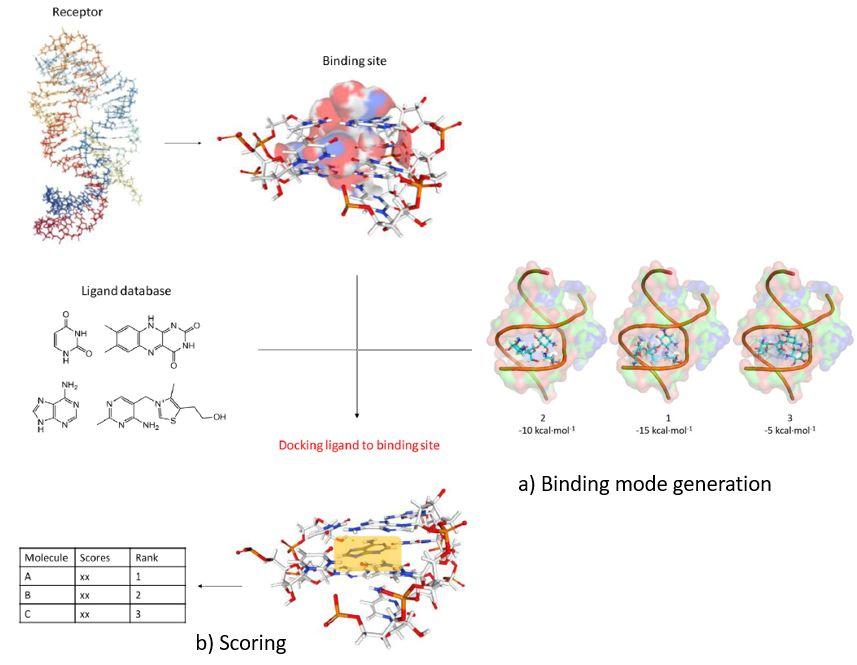
图2.分子对接的两个步骤:a)结合模式生成,b)结合模式评分和预测。
图片来源:EJMC
核酸分子对接程序
与蛋白质分子对接相比,小分子核酸对接方法相对不多。许多蛋白质都包含一个定义明确的疏水性结合位点,而核酸通常有很多具有高电荷密度和极性的暴露于溶剂的结合口袋。这些差异使得需要修改现有的蛋白质-配体对接程序来适应核酸-配体对接。表1中列出了本文中讨论的对接程序,本文涉及的打分函数包括DrugScoreRNA、LigandRNA、KScore、iMDLScore1和iMDLScore2。
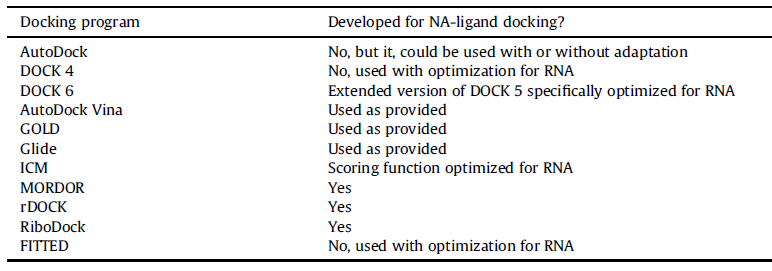
表1. 本文中讨论的对接程序
核酸分子对接用于预测结合模式
将天然配体对接至相应的大分子(自对接)或同一大分子的其他晶体结构(交叉对接)中时,可以基于对接准确预测的结合模式的能力来评估对接程序的性能。对接的配体构象与共结晶结构中配体构象之间重原子的均方根偏差(RMSD)可以量化衡量对接程序预测结合模式和预测结合亲和力的准确性。大多数对核酸-配体对接程序的性能比较研究都关注的是预测正确的结合模式而不是结合亲和力的成功率,而成功率又因数据集的大小质量不同而有所差异。
2004年,Detering和Varani表明DOCK和AutoDock可以在2.5Å范围内重现实验结合模式,在16种RNA-配体复合物中成功率为60%。其他研究者在相同的RMSD要求下进行了多项研究。在25种DNA-配体复合物的对接测试中,AutoDock的成功率为68%。AutoDock Vina在未对核酸进行特定处理的情况下,在56种RNA-配体复合物中成功率为29%,而Glide和rDock的成功率分别为18%和54%。在去掉水分子的情况下,60种RNA-配体复合物中,Glide和GOLD的成功率分别为60%和62%。AutoDock在自对接11个氨基糖苷化合物时,平均RMSD为3.25Å,作者考虑水分子在配体与RNA结合中的作用及大分子的柔性后,AutoDcok对接的平均RMSD提升为1.41Å。MORDOR是专为RNA-配体对接设计的程序,可在2.5Å内复现实验结合模式,在57个案例中成功率为74%。RiboDock用10种RNA-配体复合物进行测试时的成功率为50%(RMSD<2Å范围)。DrugScoreRNA用31种RNA-配体复合物自对接时成功率为42%。另一项研究中,LigandRNA能在42种RNA-配体复合物中找到2Å范围内的配体构象的成功率为36%,而DOCK6的成功率为36%,DrugScoreRNA为31%。当可用共晶结构数增加时,性能研究在更大的数据集上进行。ICM用96种RNA-配体复合物进行自对接的成功率为53%。作者开发的FITED用229种RNA-配体复合物进行自对接的成功率为77%,考虑氢键受供体等因素后,成功率提升为83%(RMSD<2.5Å)。当配体为插层剂时,AutoDock
在67种DNA-插层剂复合物中再现插层位点和小沟间的结合模式的成功率为80%(RMSD<2Å)。
核酸分子对接用于区分化合物活性
在一些研究中评估了对接程序预测结合亲和力的能力。ICM在对接一组48种RNA-配体复合物后,复合物的结合能实验值与计算值之间的线性相关系数为0.71。AutoDock在RNA-氨基糖苷复合物的数据集中的R2为0.84。打分函数KScore在15个RNA-配体复合物和9个DNA-配体复合物上获得的计算与实验结合能之间的线性相关系数分别为0.81和0.68。iMDLScore1在45种RNA-配体复合物上的线性相关系数为0.7,iMDLScore1为0.79。结合位点为DNA小沟时,与DOCK相比,AutoDock可以提高活性化合物库的富集度(富集值(f=1%)=86%)。对接软件区分活性和非活性结合剂的能力取决于所研究分子的种类。例如,AutoDock对氨基糖苷的表现较好,而DOCK对芳香族分子的准确性更高。
上述讨论可以提供对于不同对接程序及打分函数的见解,但有一定的局限性。首先,成功率很大程度上取决于数据集大小、复合物的选择及RMSD范围。其次,自对接的研究结果在指导发现新化合物的过程中可靠性有限。最后,少有研究报道预测的结合亲和力如何与实验值相关。因此,这些方法的一般适用性和预测能力仍然难以捉摸,需要更广泛的评估研究。
在真实世界中对对接程序的准确性验证是通过考察虚拟筛选能否发现新颖的结合剂来完成的。2010年,使用ICM对靶向于G-四链体的20000种化合物进行基于对接的虚拟筛选。最终选择了5种化合物经实验验证fonsecin B是G-四链体稳定剂。2011年,使用DOCK 3.5.54对靶向于腺嘌呤核糖开关(AR)的超过2500种化合物进行基于对接的虚拟筛选,考虑了配体与AR之间的大量氢键和π堆积,通过实验验证选择了5种得分最高的化合物,其中有4种在微摩尔范围内表现出与AR的亲和力,且共结晶得到的结合模式与对接预测的接近。
核酸分子对接面临的五大挑战
从蛋白到核酸的程序定义
多年来,被广泛运用的分子对接程序定义了药物化学家设计和优化与蛋白质结合的方式,其开发重点是蛋白质。研究核酸的药物化学家在进行对接研究时,首先考虑的是基于现有的蛋白小分子对接程序性能来对自己的体系进行修饰。有些对接程序在蛋白小分子复合物训练集上的准确性足够高所以不需要进行修饰,例如AutoDock。DOCK6则推出了将小分子对接进核酸的特殊功能,改进了构象采样方法,可以提高对接更大且更柔性的配体时的准确性(G-四链体结合剂和氨基糖苷类化合物较大且有更大柔性)。相应地,优化后的方法在蛋白质-配体复合物上的准确性会降低。DOCK中还添加了一个考虑金属离子和水的项目。有些程序则专门针对核酸-配体对接,如MORDOR、RiboDock(演变为rDock)。无论哪种方法都需要在开发对接程序时考虑核酸与蛋白的不同之处。
打分函数
与蛋白相比,核酸的极性大且某些非常规相互作用需要特殊处理,所以需要专门设计打分函数以预测RNA与配体的亲和力。RiboDock采用基于经验的打分函数,添加了一个解释带正电的碳(如胍盐)与带负电的基团(如羰基)之间相互作用的术语,以对不常见的静电相互作用建模。还引入了一个有利于π-π堆积的术语。DrugScoreRNA采用基于知识的打分函数能更频繁地观察到相互作用,LigandRNA也是基于知识的打分但观测重点在氢键的方向性。打分函数的主要开发挑战在于在高通量方式下计算核酸-配体结合时的熵能消耗。目前使用AutoDock生成的结合模式簇的大小来估计任何给定结合模式的振动熵。
RNA和DNA的柔性
大多数方法忽略核酸的固有柔性将其视为刚性受体,但RNA可能适用与配体结合时会运动而产生构象变化(图3)。通过这些运动,配体被封装在结合口袋内,几乎不暴露于溶剂中。
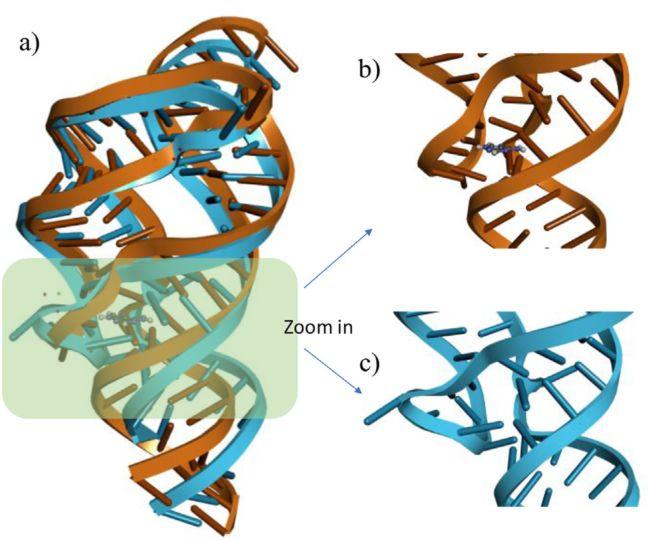
图3.嘌呤核糖开关在结合配体时的局部构象运动。
图片来源:EJMC
目前没有一项普遍适用的考虑核酸柔性的技术。为处理RNA的柔性,MORDOR使用分子力学最小化技术将RNA分子限制在实验得到的结构上从而诱导与配体的结合。然后,将配体放置于某个热点碱基上后便可以探索受体的表面。对接过程中的能量最小化步骤非常耗时,所以该方法不适用于大型分子库。但该方法在2.5Å范围内能达到超过70%的准确率。使用DOCK将化合物定位到靶标位置再用ICM的内部坐标探索构象空间也能处理核酸的柔性。另一种选择是通过一组预定义的RNA构象进行整体对接,这是通过组合一组AutoDock栅格来实现的,该栅格可对栅格中不同的RNA构象进行建模,从而对构象集合建模。还可使用NMR和分子动力学模拟结合的方法构建RNA整体动态构象,再将小分子对接到整体上来解决RNA构象柔性的问题。除此之外,DrugScoreRNA采用弹性势能网络来精准描述RNA-配体相互作用,该方法仅限于局部较小的构象变化。还有一种新颖的蒙特卡洛(MC)算法,从头将药物对接至DNA,完全考虑了两个分子的柔性,复合物的柔性由MC变量定义且无需事先选择结合位点。
金属离子和水分子
核酸的高电荷性质导致其会与水分子和金属离子产生强烈的相互作用。对于核酸-配体复合物,桥连水分子在稳定络合物方面起关键作用,能提供额外的氢键相互作用。金属离子结合位点起初是通过分子动力学模拟来识别的。后来研究Glide对接25种水合RNA-配体复合物时发现有水分子时的准确性显著提高,但自对接和交叉对接时需要的水分子可能不同。于是,开发了一种通过“可置换”的分子来模拟关键水分子的新颖方法。
通常,在核酸的结合位点发现诸如Mg2+和Mn2+之类的金属离子,将其作为RNA靶标一部分来考虑时,会改善预测的结合模式的准确性。但是,在大多数可硬的核酸对接方法中,尚未解决桥接水分子和金属离子的问题。
高电荷大分子
现实中,所有磷酸盐的负电荷都被溶液中的抗衡离子中和,但这些离子由于其位置不固定而很难在分子对接中被考虑进去。另一个关键点是当氢结合到诸如磷酸酯的各种集团上时对水分子极化的调节,而对接程序往往将所有水视为等效的。
总结
蛋白-配体对接方法虽然可以应用于核酸-配体对接,但结合模式预测准确度较低(很少高于60%)。低成功率的一个原因是没有考虑核酸的柔性,它是核酸与配体结合的关键结构因素。水分子和金属离子在结合过程中也很重要,开发新的对接程序或修饰蛋白-配体对接方法时必须考虑这些因素。尽管现有的这两类对接程序都显示出潜在的优越性能,但仍有改进的空间。除此之外也需要越来越多的共晶结构数据来支持更广泛的核酸对接程序评估。
参考文献:
Nicolas, M. et al. Challenges and current status of computational methods for docking small molecules to nucleic acids, European Journal of Medicinal Chemistry (2020), https://doi.org/10.1016/j.ejmech.2019.02.046