引言
新型冠状病毒(SARS-CoV-2)已在全球范围内爆发,药物研发刻不容缓。计算机辅助药物设计(CADD)作为加速药物发现的有效策略,能否在此次疫情中发挥作用?前面有大量计算相关研究,但都很少有实验验证的案例,华东理工大学李洪林团队和武汉大学病毒学国家重点实验室徐可合作,通过虚拟筛选发现对新型冠状病毒SARS-CoV-2有效的广谱抗病毒获选药物,成为CADD加速药物发现的成功案例。

先睹为快
靶点
hDHODH:二氢乳清酸脱氢酶(PDB ID:1D3G)
数据库
SPECS数据库(~280000个小分子)
计算方法
分子对接
Prime/MM-GBSA
相似度分析与聚类分析
计算软件
Schrödinger LLC、Pipeline Pilot 7.5
计算流程
李洪林/徐可团队应用分子对接和MM-GBSA研究相结合的有效虚拟筛选策略来发现二氢乳清酸脱氢酶(hDHODH)抑制剂。依次通过分子对接、Prime/MM-GBSA、相似度分析与聚类分析等方法,筛选了SPECS数据库中约28万个小分子,选出了47个化合物进行生物活性测试,其中18个化合物的IC50值在0.11~18.8μM,表明它们是一种新的hDHODH抑制剂。随后研究团队进行了结构优化最终发现了两种强效的DHODH抑制剂,S312和S416。这两种新型抑制剂表现出良好的广谱抗病毒药效,包括新型冠状病毒SARS-CoV-2,可作为流感病毒治疗的候选药物。
背景
新型冠状病毒(SARS-CoV-2)已在全球范围内爆发,危害程度也被定义为“大流行”。现有的直接作用抗病毒(DAA)药物由于病毒特异性不能立即应用于新的病毒,因此,宿主靶向抗病毒(HTA)药物通过阻断病毒复制和克服潜在的病毒诱变,在对抗广谱病毒方面具有许多优势。
基于结构的虚拟筛选
华东理工大学李洪林团队和武汉大学病毒学国家重点实验室徐可团队合作,针对抗病毒靶点二氢乳清酸脱氢酶(hDHODH)开展基于结构的虚拟筛选,依次通过分子对接、Prime/MM-GBSA、相似度分析与聚类分析等方法,筛选了SPECS数据库中约28万个小分子,选出了47个化合物进行生物活性测试,其中18个化合物的IC50值在0.11~18.8μM,表明它们是一种新的hDHODH抑制剂。随后研究团队进行了结构优化最终发现了两种强效的DHODH抑制剂,S312和S416。发现了两种强效的二氢乳清酸脱氢酶(DHODH)抑制剂,S312和S416,对各种RNA病毒,包括甲型流感病毒(H1N1,H3N2,H9N2),寨卡病毒,埃博拉病毒,特别是最近出现的新型冠状病毒SARS-CoV-2都显示出广谱的抗病毒作用,首次证实了DHODH抑制剂能够通过抑制病毒复制和调节免疫两种途径发挥广谱抗病毒药效。
体内活性验证
研究团队首次在动物体内证实DHODH候选药物可以100%保护小鼠免受致命流感的攻击,这与DAA药物一样好,甚至在DAA药物不再有效的感染后期也有效。研究团队进而提出DAA和HTA联合用药是一种很有前途的抗病毒治疗策略,并证明S312在治疗严重感染动物的晚期流感疾病方面比奥司他韦显示出更多的优势。在国内新冠疫情爆发期间,该研究团队迅速展开了两DHODH抑制剂S416和S312抗新冠病毒的活性测试,其中S416细胞试验抗病毒作用比瑞德西韦和氯喹分别强45倍和60余倍。到目前为止,在SARS-CoV-2感染的细胞中,S416被报道是最有效的抑制剂,其EC50为17nM,SI值>5882。
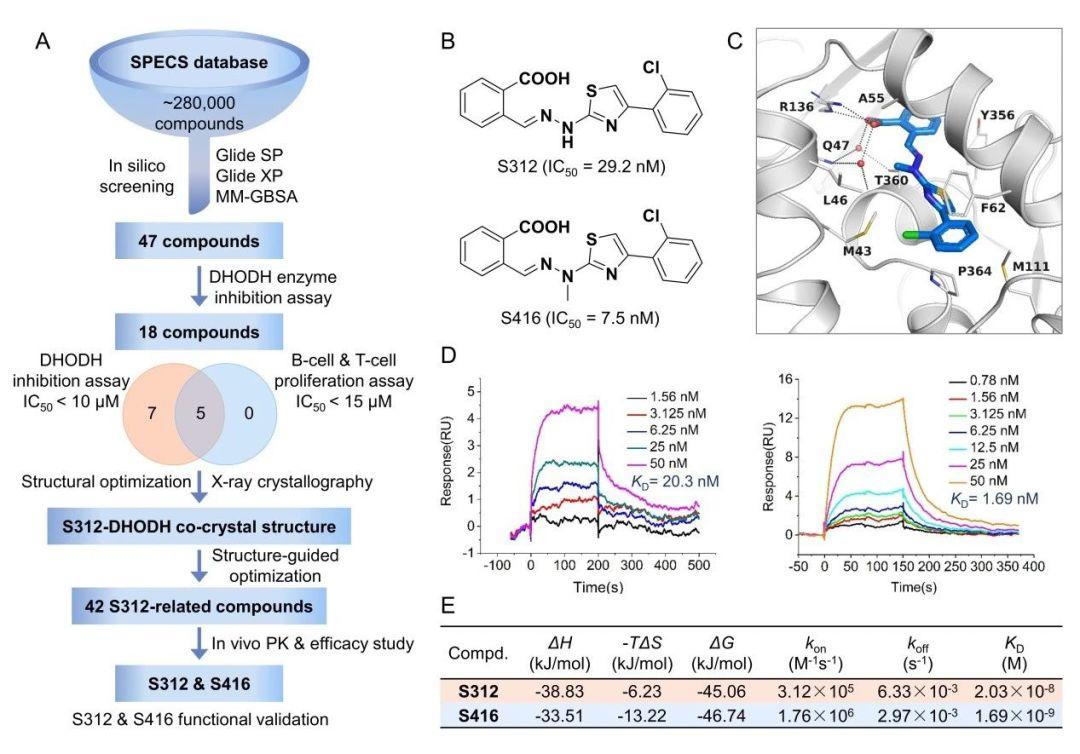
图1. 新型高效DHODH抑制剂的发现流程.
图片来源:bioRxiv
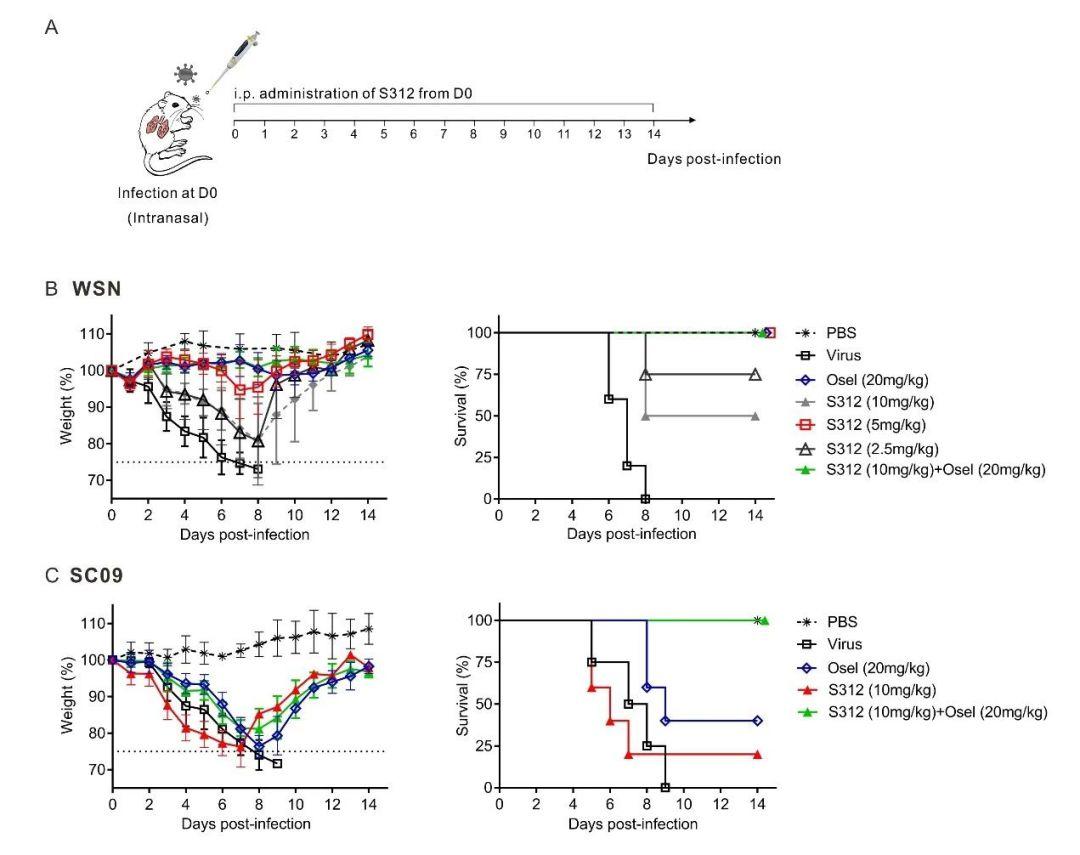
图2. S312在甲型流感病毒感染小鼠体内的抗病毒活性
图片来源:bioRxiv
参考文献
Novel and potent inhibitors targeting DHODH, a rate-limiting enzyme in de novo pyrimidine biosynthesis, are broad-spectrum antiviral against RNA viruses including newly emerged coronavirus SARS-CoV-2. Rui Xiong, Leike Zhang, Shiliang Li, Yuan Sun, Minyi Ding, Yong Wang, Yongliang Zhao, Yan Wu, Weijuan Shang, Xiaming Jiang, Jiwei Shan, Zihao Shen, Yi Tong, Liuxin Xu, Chen Yu, Yingle Liu, Gang Zou, Dimitri Lavillete, Zhenjiang Zhao, Rui Wang, Lili Zhu, Gengfu Xiao, Ke Lan, Honglin Li, Ke Xu. bioRxiv 2020.03.11.983056; doi: https://doi.org/10.1101/2020.03.11.983056