
引言
1. 介绍了抗生素耐药性(AR)相关的数据库;
2. 大多数用于识别AR相关基因特征的方法提供了一种基于相似性的方法来预测AR基因;
3. 深度学习已经被用于寻找基因组中的AR基因;
4. 确定了表型敏感性检测和基于基因组的AR预测之间的一致性;
5. 讨论了计算工具在管理AR中的作用。
背景
抗生素耐药性(AR)是细菌抵抗曾经可以抑制它们的药物的能力。细菌会对处方药物产生适应性变化从而在患者体内得以存活。多药耐药性(MDR)是对多种抗生素的同时耐药性,由于滥用和过度使用药物以及新药发现的减少,细菌耐药性正在逐步成为全球的主要威胁。使用抗生素治疗细菌性疾病与后续发展的抗生素耐药性之间有很强的相关性。由于过度使用抗生素,在选择性压力下,细菌会产生多种耐药机制。这种现象促进了耐药细菌的生长和脆弱细菌的死亡。多种抗生素耐药菌株的存在导致疾病治疗的有效性降低、死亡率增加。因此,开发针对大量AR细菌群的有效治疗药物已成为对抗抗生素耐药性的迫切需求。在当今时代,细菌的全基因组测序(WGS)不是很困难,但AR相关信息的汇编和挖掘仍然是一个重大挑战。在过去的20年里,已经开发了许多数据库、数据汇总工具和数据查询渠道,以研究细菌AR中的机制,并预测细菌AR基因和基因组。本文批判性地回顾了所有与AR相关的计算资源,以帮助科学界选择最合适的资源来满足他们的需求。其中包含用于直接或间接维护AR相关信息的数据库,文章根据内容类型对这些数据库进行分类,并描述了每种资源的效用和局限性以帮助用户找到定期更新的最佳资源。这些AR数据库被大量用于预测AR基因或基因组。此外,深度学习技术已被用于预测新序列基因组中的AR基因。本文推荐了相关工具并提供了详细的描述以便用户能够找到最适合他们的研究的工具,此外,还讨论了管理AR相关细菌性疾病治疗的替代解决方案。希望这些信息将帮助研究人员解决与AR相关的问题,同时也希望能为药物的发现做出贡献。
数据库
科学界正在积极地收集和汇编在过去二十年中获得的与AR相关的信息,以了解细菌抵抗药物的机制。本文将这些资源分为四类:AR基因数据库;酶专用数据库;生物体特定数据库;其他数据库(表1)。
表1. 与抗生素耐药性基因相关的数据库列表
图表来自 Drug Discovery Today
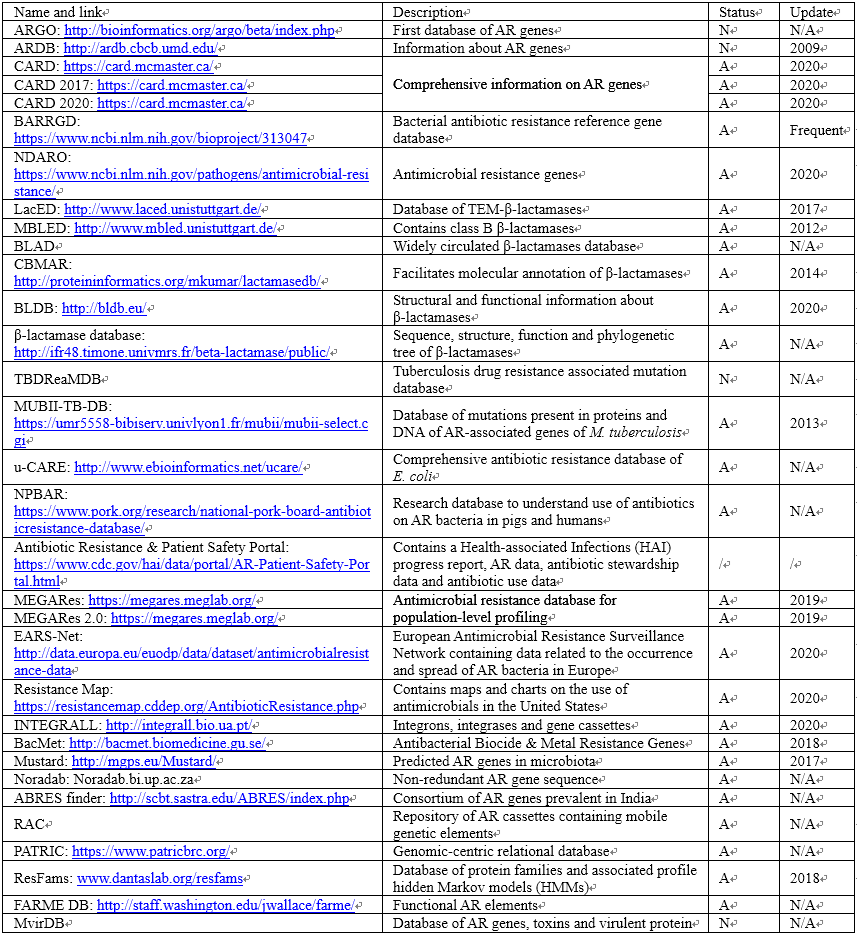
AR基因的数据库
线上抗生素耐药基因(ARGO)是2005年开发的第一个列出AR基因的数据库。它包含了555个β-内酰胺酶耐药基因和115个万古霉素耐药基因的综合信息。此数据库截止到2005年便不再更新。2007年,MvirDB数据库被开发出来以分类AR基因、有毒蛋白质,该数据库包含的AR基因有限,在开发后也没有再次更新。2009年,抗生素抑制剂综合性基因数据库(ARDB)建立,用于表征和鉴定具有抗生素耐药性的基因。该数据库已不存在,但所有的数据都可以在2013年开发的综合抗生素抑制剂数据库(CARD)中找到。该数据库包含抗生素耐药性基因及其相关蛋白质和表型的分子和序列数据,还包含与抗生素及其靶点相关的信息以及抗生素耐药性的理论,目前该数据库将定期更新;截止到2020,该数据库包含了3057个参考序列、3103个抗菌素(AMR)检测模型和1704个单核苷酸多态性(SNPs)。此外,许多预测和分析工具也包含在数据库中;此外还包含了新的残基和异构体的分析和统计总结。RGI等分析工具的存在,增强了该数据库在预测抗生素耐药性方面的效用。还有一些用于维护AR基因的数据库,如国家生物技术信息中心(NCBI)生物数据,细菌抗生素耐药性参考基因数据库(BARRGD)、由NCBI维护的与AR相关的抗菌素耐药性生物体国家数据库(NDARO)。NDARO数据库包含了关于抗菌素耐药性基因的大量信息,该数据库包含ARMFinderPlus,便于用户找到AR基因和抑制突变点。
β-内酰胺酶特异性数据库
负责降解或修饰抗生素的重要酶之一是β-内酰胺酶,这种酶水解了β-内酰胺抗生素的β-内酰胺环,从而消除了对细菌的伤害。β-内酰胺抗生素是用于治疗严重革兰氏阴性感染的广谱抗生素,大肠杆菌、肠沙门氏菌和肺炎克莱伯氏菌是最常见的革兰氏阴性细菌,可导致人类的多种疾病,因此,β-内酰胺酶被认为是AR领域中最重要的酶,并且建立了许多已专门用于这些酶的数据库。历史上,β-内酰胺酶的第一个数据库由拉希诊所运营,随后这些数据被转移到NCBI并可以在BAARGD中找到。在众多数据库中,乳酸乙酰胺酶工程数据库(LacED)包含TEM和SHV-β-乳酸乙酰胺酶的信息,是最古老的数据库。2012年开发的β-乳酰胺酶工程数据库(MBLED)包含了关于B类β-乳酰胺酶的信息。LacED和MBLED都是乳酰胺酶工程数据库的一部分,分别包含关于A类和B类β-乳酰胺酶的信息,但它们仅限于一类特定的β-内酰胺酶。因此,2013-2014年开发了甜乳胺酶数据库(BLAD)和综合甜乳胺酶分子数据库(CBMAR)。BLAD包含大约2000个基因序列,以及200个β-内酰胺酶的三维晶体结构及其结合配体的理化学性质,CBMAR提供了关于β-内酰胺酶的分子和生化功能的信息,并进行了详细的分类。β-乳酰胺酶领域最全面和最常更新的数据库是甜乳酰胺酶数据库(BLDB),它于2017年开发,每月保持更新,包含所有目前已知的β-内酰胺酶的序列以及生化和结构信息,该数据库包括各类β-内酰胺酶,它们的代表性突变体、动力学信息和三维结构。此外还有一个未发表的β-内酰胺酶数据库,其中β-内酰胺酶是根据其活性位点上的残基来分类的,此外,结构信息、动力学信息和最小抑制浓度等信息也包含在内。数据库中的分子标号直接与NCBI数据库链接,结构信息与蛋白质数据库链接,数据库中包含的结构和动力学信息可以帮助我们掌握β-内酰胺酶的活性位点残基,从而研究它们对不同抗生素的识别和特异性。
生物特异性数据库
结核病是最致命的传染病之一,而病因细菌结核杆菌经常通过特定基因的突变来获得耐药性,为了提供关于细菌耐药性全面的信息,2009年开发了结核病耐药性突变数据库(TBDReaMDB),该数据库在开发新的测试药物方面具有巨大的效用,可以帮助快速识别M.结核病菌株的敏感性特征。在此基础上更新的数据库MUBII-TB-DB于2014年开发,该数据库包含了一组AR相关结核基因的蛋白质和DNA中的突变信息,使用该数据库进行查询序列简单快捷,该数据库可用于识别结核杆菌突变体并帮助进行的抗生素研发,此外,还可用于其他微生物突变体的鉴定,因此也可用于多种耐药微生物的监测和控制。为了研究大肠杆菌的耐药性,2015年开发了综合抗耐药库(u-CARE),它含有52种抗生素,以及大约107个基因及参与大肠杆菌药耐药性的SNPs和转录因子。此外,还有2017年建立的国家猪肉抗生素耐药性数据库,目的在于帮助了解某些抗生素对猪及其环境中存在的AR细菌的影响,以及这些细菌向其他猪和人类传播的渠道。
其他数据库
还有其他几个数据库可以以某种方式处理AR字段。其中,PSA是一个有四个交互式数据集的web应用程序,包含AR数据(AR数据描述了具有耐药表型的细菌)、医疗保健相关感染(HAI)数据、门诊抗生素使用数据和住院抗生素管理数据。对于病原体中出现的耐药性造成的治疗困境,应用PSAweb可以对这类抗药菌株进行研究。MEGARes于2017年开发,对抗菌素耐药性的人群水平分析非常有用。此外,欧洲抗菌素耐药性监测网络(EARS-Net)数据库包含与AR细菌相关的数据。ResFams和FARMEDB是包含基因组的抗生素耐药性数据库,其中ResFams是2015年建立的具有AR功能及其相关特征的蛋白质数据库,功能抗生素耐药元件数据库(FARMEDB),也被称为元基因组元件(FARME)数据库,包含来自元基因组数据的DNA和蛋白质序列。此外INTEGRALL于2009年开发,提供了DNA序列和基因排列的详细信息等数据,2011年开发的RAC(耐药盒存储库)数据库包含一组基因盒,这个基于web的平台上的数据将有助于揭示整合子在细菌相互作用和适应性反应中的作用。此外,多重抗抗生素抗炎剂(MARA)是2018年4月开发出来一个包含革兰氏阴性细菌的移动元素和移动AR基因的数据库,该数据库能够实现提交序列中抗性基因和相关移动元件的比较分析。除了抗生素之外,抗菌生物杀菌剂和金属还通过共同选择对细菌群落中AR的发展和维持做出了重要贡献,抗菌生物灭活和金属抑制基因数据库(BacMet)开发于2014年,最后更新于2018年,可通过共同选择促进AR的发展或维持。该数据库包含753个经实验证实的和155512个预测的耐药基因,以及111种化合物,包括58种抗菌生物杀菌剂和23种金属。抗生素耐药性领域相当广泛,生物信息学家还提供了其他一些不同的资源,如ARGMiner是一个基于网络的管理系统,包括其基因名称、耐药机制、抗生素类别、流动性证据和临床重要菌株。Mustard是一个AR决定因素和策划基因集的数据库,该数据库于2017年开发,包含了来自人类肠道微生物群中20个家族的6095个AR决定因素。Noradab抗生素耐药性数据库创建于2018年,它包含从ARDB和CARD数据库中收集的抗生素耐药基因序列;抗生素耐药性基因发现器(ABRESfinder)是一个在印度流行的AR基因联盟,它共包含37种抗生素,377个基因家族和36,467个基因。同样,PATRIC是一个2011年开发的以基因组学为中心的关系数据库,包含了致病菌的所有基因组数据。另一个MvirDB数据库建于2007年,整合了其他数据库中管理的AR基因、DNA数据。对于AR预测基因的工具,包含前文提到的一些工具,还有诸如CARD包括分析分子序列的工具,以及基于同源性和SNP模型的电阻预测的RGI软件等。图2所示为对抗抗生素耐药性的计算资源的时间轴。
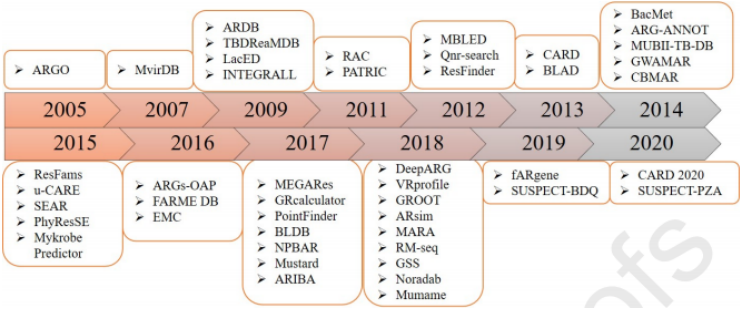
图2. 对抗抗生素耐药性的计算资源的时间轴
图片来源于Drug Discovery Today
AR基因的预测
ARG-annot是一种检测细菌基因组中AR基因的生物信息学工具,于2014年建立,该工具可用于检测现有的耐药性基因决定因素、AR相关的突变点,以及细菌基因组中抗生素耐药性基因决定因素。DeepARG是一种基于深度学习方法的工具,该工具可用来预测元基因组数据中的AR基因,开发于2018年,可以作为命令行工具和web服务使用。DeepARG的作用并不局限于AR基因,它可以用于训练任何一套基因,因此它可以用于创建新的深度学习模型。成对比较模型(PCM)是一种基于3D的解释抑制基因的通用方法,它使用一种特定的方法来构建结构模型并评估其相关性。Mykrobe预测器于2015年开发,用来分析特定细菌的完整基因组,并在几分钟内预测抗生素或药物的耐药性。ARIBA是2017年开发的一种工具,可以通过配对序列来识别AR相关基因和SNPs,该工具具有快速、高效和准确的特点。表2列出了用于帮助表征AR的所有计算工具。
表2. 可用于抗生素耐药性研究的工具和软件的列表
图表来源于Drug Discovery Today

AR相关突变的预测
ResFinder开发于2012年,用于识别全基因组数据中获得的AR基因和突变染色体。该工具可以在https://cge.cbs.dtu.dk/services/ResFinder/上访问。细菌耐药性相关突变的全基因组评估(GWAMAR)突变检测工具于2014年开发,通过对细菌全基因组序列的比较分析来检测耐药性相关突变。2016年,科尔蒂娜等人开发了“异常变异代码”工具,可以在github上免费获得,他们的代码在分子动力学模拟中使用位置相互信息来预测影响β-内酰胺酶耐药性的局部和异构突变。2018年,格里洛特等人开发了一种基于分子条码方法的深度测序工具RM-seq,该工具有助于检测和估计细菌群中抗突变性的功能。PointFinder开发于2017年,用于检测与病原细菌染色体点突变相关的抗菌素耐药性。此外,有一些工具利用分子动力学技术来识别突变,如PZA是一种用于治疗结核病的药物,但对吡嗪酰胺的抑制作用有时会由于结核病人的吡嗪酰胺酶蛋白(PncA)的突变而发生,此工具提供的结构分析可帮助患者疾病的治疗。
对AR的基因注释的工具
VRprifle是一项基于网络的服务,开发于2018年,旨在帮助探索AR基因。该工具可用于帮助实时定义致病菌中与疾病相关的基因簇,也可满足细菌基因组可变区域重新注释不断增加的需求,电阻体分析是与参考基因相似的复杂过程,测序数据庞大,分析工作流程复杂。GROOT开发于2018年11月,该方法使用了索引,可以用于分类元基因组样本中的抗药基因。PhyResSE是2015年开发的另一种网络工具,用于描述结核病系和其他谱系中来自全基因组测序数据的AR,也可用于识别多耐药结核复合物(MTBC)耐药介导变异及分类系统发育谱系。GRcalculator于2017年开发,用于提高临床研究中药物反应研究的价值和可靠性,使用这种方法可计算、分析和可视化药物反应数据;GRmetricsR也可用于离线数据分析和可视化,在测量细胞对药物的反应方面具有优势,此外该工具可以用于药物疗效和效力的比较。ARsim模拟工具在2018年开发,用于模拟细菌生长和抗生素耐药性,以决定特定抗生素是否适用,以寻找对抗抗生素耐药性的新方法。
基于元基因组的工具及AR基因相关资源
片段抗抗生素基因iENntifier(fARGene)是2019年4月开发的一种工具,可以用来直接从元基因组数据中识别和重建AR基因,包含了许多已开发和优化的耐药基因模型。此外,该工具使用户能够创建和优化他们的抑制基因的模型。然而,如果用户对元基因组数据中的基因特异性突变点感兴趣,可以使用2018年9月开发的一种名为Mumame的工具。此外本文提供了AR基因搜索的相关资源(见表3),如抗菌素耐药性搜索引擎(SEAR)于2015年建立,目的是从原始序列数据中检测抗素耐药性基因。
表3. 可用于抗生素耐药性研究的资源
图表来源于Drug Discovery Today
总结与展望
抗生素耐药性的不断增加是全球面临的主要威胁,根据世卫组织的数据,到2050年,耐药疾病每年可能导致1000万人死亡,到2030年,AR可能迫使多达2400万人陷入极度贫困。目前,每年至少有70万人死于耐药性疾病。即使在当前全球大流行的COVID-19中,AR也发挥着重要作用,使病毒不断产生耐药性,加重了疾病。据报道,10%的新冠患者出现了继发性细菌感染,从而造成了器官损伤或死亡。此外,一些研究表明,几乎所有的COVID-19患者都以抗生素作为其治疗方案的一部分,但抗生素的广泛使用也在推动耐抗生素细菌的进化。几十年来,抗生素一直是对抗传染病的主要救世主,但耐药性的出现已经将肽疗法转向了肽治疗。研究表明,细胞穿透肽和抗生素的结合可以有效地对抗抗药性病原体,但肽的半衰期仍然是一个主要的问题,这限制了它们克服耐抗生素疾病的应用。此外,疫苗接种也是防治传染病侵袭的一种策略。另一方面,包含AR基因、蛋白质、酶等信息的数据库的建立,以及从高通量测序数据中了解AR的新工具的发展,有望对人类疾病的消灭和经济的发展做出巨大的贡献。本综述中讨论的资源有助于促进药物开发过程中的临床前研究。此外,不断发展的算法和软件可以作为辅助帮助对抗生物体的耐药性。通常,每种病原体都表现出一种特定的耐药机制,并随着时间和抗生素的使用而演变。这些复杂的机制使得人们难以准确地预测表型抗菌素耐药性,因为基因的表达取决于多个因素。尽管研究人员已经使用各种机器学习和深度学习方法进行研究,但往往是专门针对特定的细菌的。此外,每种机器学习技术都有其缺陷,新的算法也一直在发展用于更准确的表型抗生素耐药性的预测。我们希望对AR的准确和快速的基因识别将为基于基因序列的现在医学的发展提供帮助。
参考文献
L. Maryam, S. S. Usmani, G. P. S. Raghava, Computational Resources in the Management of Antibiotic Resistance: Speeding Up Drug Discovery, Drug Discovery Today, 2021, in Press. DOI: 10.1016/j.drudis.2021.04.016.