
引言
深度学习的质量和效率,在很大程度上,取决于被学习对象的表示。特别是,增强的药物学习,依赖于适当的分子表征(MolRs)。通过直接从分子的底层图形中,学习它们自己的优化表示,基于图形的MolRs从头学习,显著提高了对药物和物理化学性质的深度学习,优于那些基于传统的分子描述符(MolDs)和指纹特征(FFs)。因此,对MolRs的更广泛的探索,弥补了基于图的方法和其他方法的不足,从而增强了对药物性质的深度学习。许多MolDs和FFs,都来源于人类专家知识,用于全面描述分子的结构、物理化学、拓扑、结构和子结构特征,这些是有价值的特征生成和药物特性深度学习的先验知识。但关于如何将这些先验特征,转化为更合适的表征,仍然存在疑问。一般来说,良好的表征是任务非特定先知,它捕获了多个潜在解释元素的后验分布,能够对这些元素进行解纠缠和聚类,支持任务函数的平滑和灵活的局部泛化。例如,破坏-构造学习方法,能够识别高难度的细粒度图像。在破坏构造学习中,输入图像被分割成局部区域,然后对局部区域,进行洗刷以暴露有区别的局部特征,然后重构以揭示语义跨区域的相关关系,从而在三个基准数据集上,实现了最先进的(SOTA)性能。因此,适当的特征生成,可以通过广泛分析不同的MolDs和FFs,相对于已知化学空间中的大量分子的内在相关性来建立。此外,利用卷积神经网络(CNNs)的共享权值架构,将一维无序向量转换为二维(2D)聚类特征映射(Fmaps),可以实现高效的学习(参数保存)。这种特征生成方法的发展,可以通过对MolRs、开源工具、相关度量、化学多样性和特征的量化,以及维数缩减法的广泛研究来促进。这篇文章中,研究者开发了一种新的分子特征生成方法MolMap,用于将MolDs和FFs映射为鲁棒的2D Fmap,捕捉分子特征的内在相关性(本文中所使用的的数据集总结如表1)。
深度学习背景
目前,基于四个MolR类,可进行药物性能的深度学习。
1. 第一个是基于图的特征表示,其中的图卷积网络(GCNs)或图注意力网络(GATs),已被探索用于直接从分子的底层图进行从头学习,获得SOTA在制药相关任务中的性能。
2. 第二种是基于字符串的表示,其中CNN和递归神经网络已被用于从化学结构的字符串表示的嵌入中学习(例如,典型简化的分子输入行输入系统(SMILES))。
3. 第三种是图像表示,CNN已被用于,从基于规则的二维化学数字网格或Kekulé图像渲染中学习。
4. 第四种是基于知识的表示,深度学习模型已经开发出来,用于从先验的人类知识衍生的模型或FFs中学习。
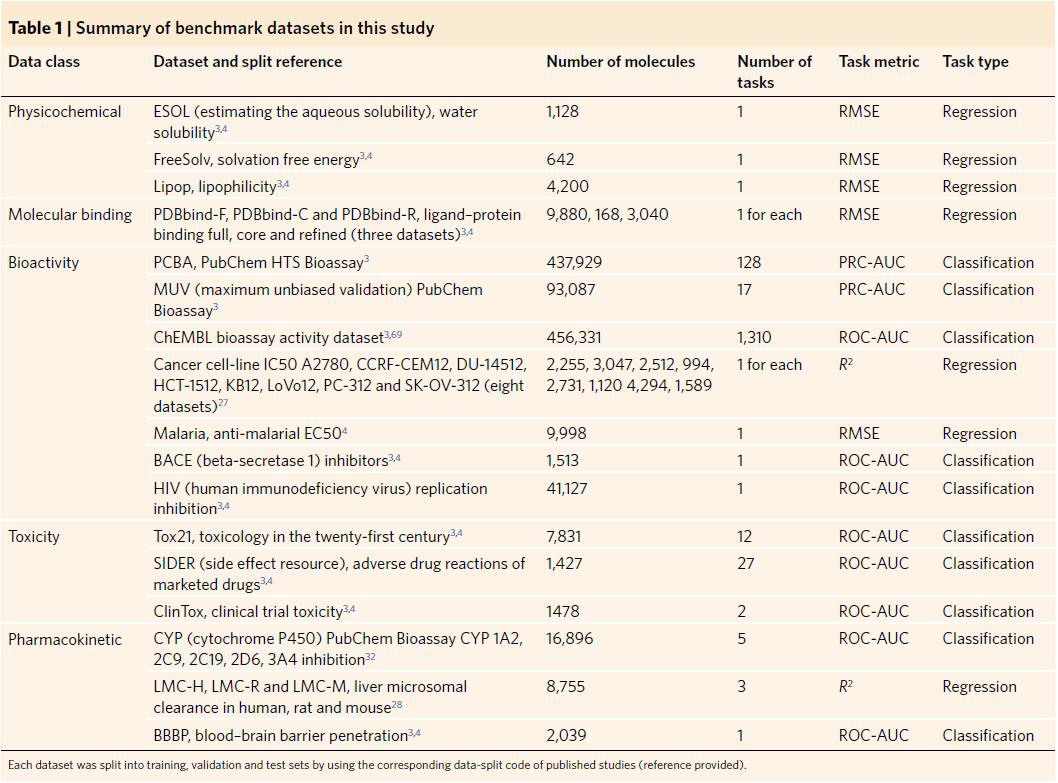
表1. 本研究基准数据集总结
表格来源于Nat. Mach. Intell.
虽然在不依赖人类直觉的情况下,探索较低级的表示方式是更好的,但广泛的MolDs和FFs知识基础,对于从人类知识的角度学习MolRs和药物特性,是非常有用的。特别是,MolDs和FFs的子集显示出高度的相关性,这为合适的MolRs提供了独特的线索。一些MolDs或FFs是通过设计相关的(例如,MolWeight和MolExactWeight),而一些不相关的MolDs显示出高度的内在相关性。化学筛选收集的调查显示,极性表面积与氢键受体和供体的数量有关。这些相关的MolDs或FFs的聚类以及它们在2D fmap中的投影,使特征模式聚类,能够通过共享权CNN架构进行高效学习。为了协调学习MolDs和FFs,最好使用MolDs和FFs的通用相关度量。余弦相关与在某些基于FF的分子研究中广泛使用的谷本系数,和在一些基于MolDs的分类任务中广泛使用的欧氏距离一样,一直表现得相当好。因此,余弦相关可以用于基于MolD/FF的特征生成。要从CNNs的MolDs和FFs中学习,需要将高维MolDs和FFs投影到2D Fmap中,这需要一个流形学习算法,且信息损失最小。最近开发的统一流形近似和投影(UMAP)工具,基于黎曼几何和代数拓扑算法,已经证明了这一任务的竞争力。
MolMap Fmap
利用MolMap包(图1),研究者生成了阿司匹林及其类似物N -乙酰邻氨基苯甲酸的MolD和FF Fmap(图2)。尽管这些分子在结构上高度相似,但它们的MolD Fmap包含小范围的明显不同的图案,它们的FF Fmap包含明显不同图案的区域。这些模式(例如,图2中的紫色和浅蓝色虚线框)可以被典型的CNN过滤器捕捉到。在MolD Fmap中,不同的MolD类别主要集中在不同的区域。在FF Fmap中,PharmacoErGFP FFs与其他FFs基本分离,而MACCSFP和PubChemFP FFs互补分布在同一区域。此外,相关的MolDs或FFs形成集群。例如,例如,三种药物相似(QED) MolDs的定量估计(MolQedWeightsMax、MolQedWeightsMean和MolQedWeightsNone)聚在一起(图2),其他各种相关的MolDs也聚在一起。这些结果表明,MolMap fmap在分子和结构特征上,具有独特的表征和内在的相关性。
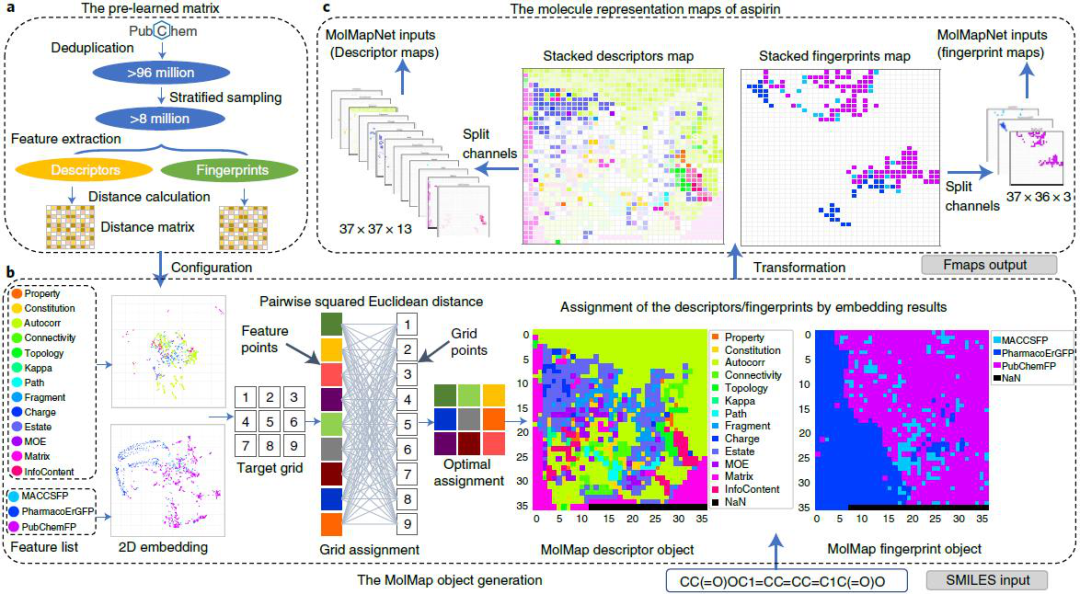
图1. MolMap特征展现流程表
图片来源于Nat. Mach. Intell.
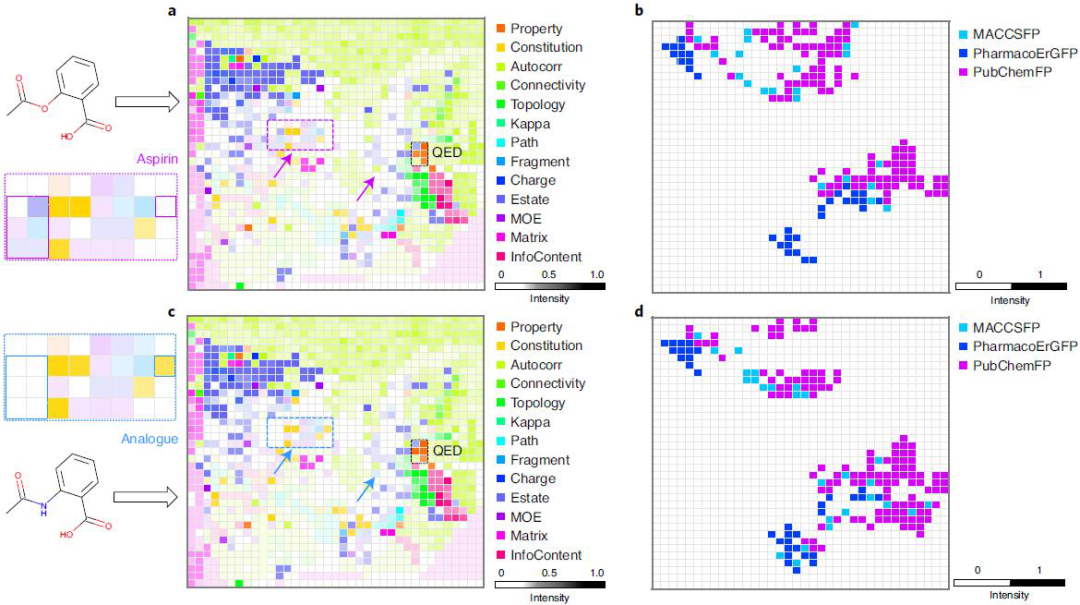
图2. MolMap多通道描述符和指纹Fmaps
图片来源于Nat. Mach. Intell.
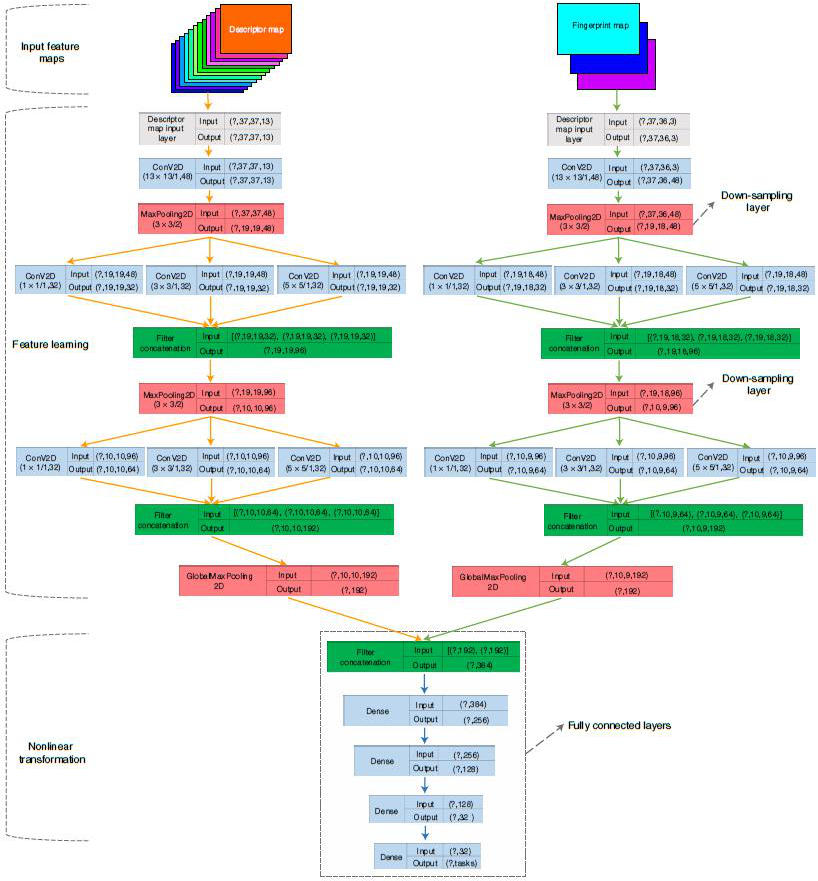
图3. MolMapNet深度学习架构
图片来源于Nat. Mach. Intell.
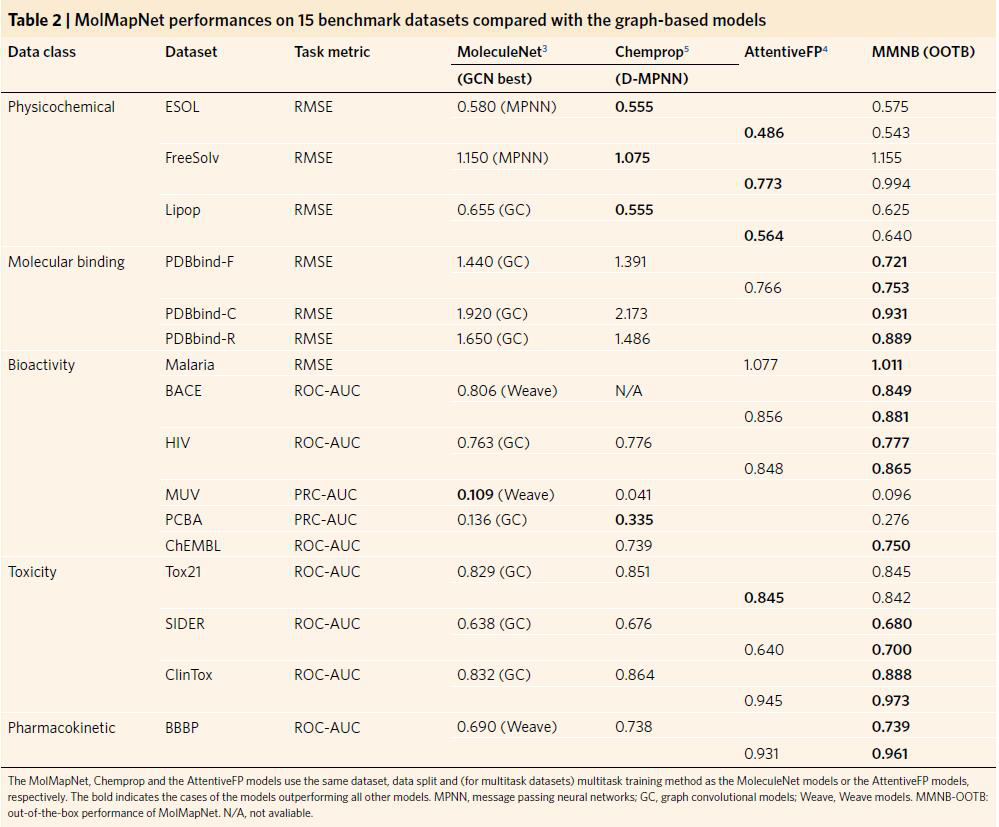
表2. MolMapNet在15个基准数据集上的性能与基于图的模型进行的比较
表格来源于Nat. Mach. Intell.
相对于基于SOTA图的GCN/GAT模型的
MolMapNet的深度学习性能
GCNs/GATs在许多基准数据集上,都达到了SOTA性能。在这些GCN-/GAT测试的数据集中,有13个药物(3个分子结合,6个生物活性,3个毒性,1个药代动力学)和3个物理化学数据集,具有可用的数据分裂代码。因此,研究者在这16个数据集上开发了MolMapNet OOTB模型(图3),并使用相同的数据分割、评价度量和(针对多任务数据集)多任务训练方法,将其与发布的GCN/GAT模型(表2)的性能进行比较。在12个药品数据集中的9个上,MolMapNet优于分子网模型和定向消息传递神经网络(D-MPNN)模型,但在所有3个物理化学数据集上,MolMapNet的表现低于GCN模型(表2)。在8个药物数据集中的7个上,MolMapNet的表现也优于AttentiveFP模型,但在所有3个理化数据集上,MolMapNet的表现低于GAT模型。这些结果表明,MolMap Fmaps是非常合适的MolRs, MolMapNet在学习SOTA的药物特性方面,是非常有用的。MolMapNet在物理化学数据集上表现不如GCN/GAT模型,部分原因如下:MolMapNet从MolDs中学习,其中一些MolDs是计算出的物理化学性质(例如,计算出的logP、clogP)。物理化学性质的MolMapNet学习,受到物理化学性质值的计算MolDs的内在误差的影响(例如,药物的clogP值与实验值之间的相关系数为0.955)。GCN/GAT从头学习算法避免了这些内在错误,因此对学习物理化学性质更有优势。在12个基准数据集上,MolMapNet OOTB模型与D-MPNN模型和AttentiveFP模型相比,在10个不同的数据分割随机种子上,进一步评估了MolMapNet OOTB模型的性能。除物理化学性质预测任务外,MolMapNet对不同种子的预测均表现出更好的表现,其差异与D-MPNN和AttentiveFP模型相当或更小。在三种物理化学性质预测任务中,MolMapNet的表现大多优于D-MPNN和AttentiveFP模型,但仍表现出与D-MPNN和AttentiveFP模型或两者相似的变化模式。
相对于基于化学图的CNN模型的
MolMapNet深度学习性能
基于化学图的CNN模型,在没有化学知识的情况下,对药物和理化性质的深度学习表现良好。其中一些CNN模型端到端的学习深度高达19层,而MolMapNet是一个层次更少的CNN。MolMapNet对这些CNN模型的差异性能,部分反映了MolMap Fmaps在学习药物特性方面的差异能力。值得注意的是,基于图像的19层CNN KekuleScope模型在8个癌细胞基准数据集上,记录了出色的性能,这些模型的性能可以与MolMapNet模型进行比较,因为这些模型的数据集和数据分割代码都是可用的。因此,在这8个基准数据集上开发MolMapNet OOTB模型,并使用相同的数据分割和评价指标,与KekuleScope模型(表3)发布的性能进行比较。MolMapNet在所有8个数据集上都优于KekuleScope模型(预测值和观测值之间的Pearson平方相关系数R2 = 0.583-0.734 versus R2 = 0.427-0.622)。
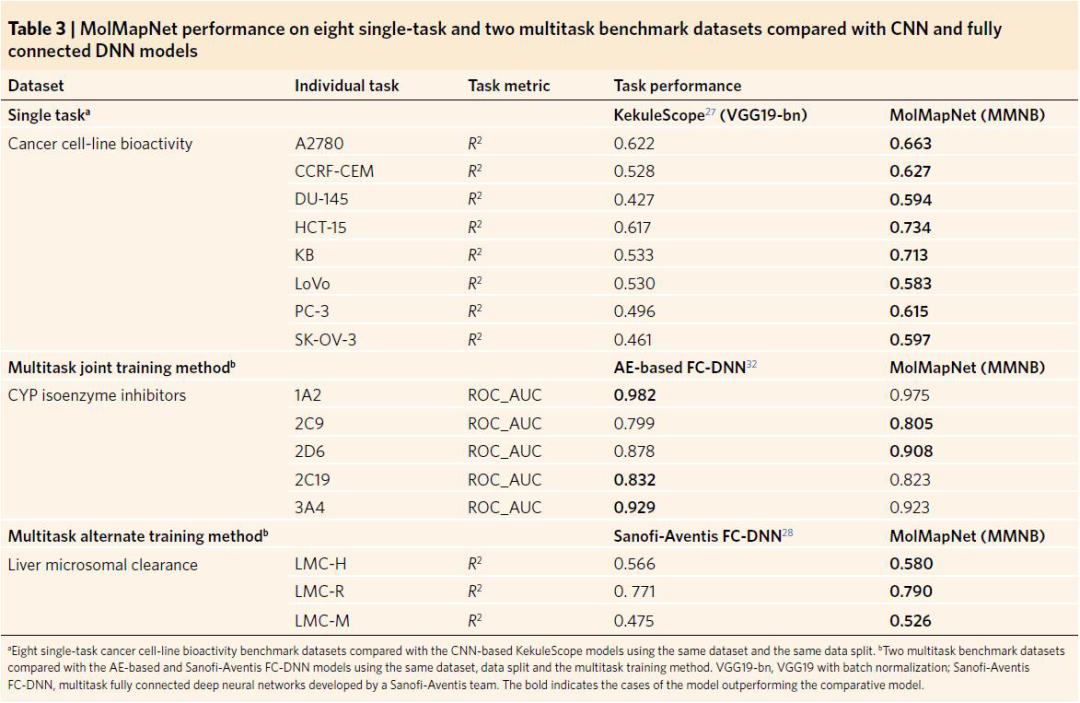
表3. MolMapNet在单任务和多任务基准数据集上的性能
表格来源于Nat. Mach. Intell.
相对于基于分子描述符的
多任务全连通深度神经网络模型的
MolMapNet深度学习性能
研究表明,多任务全连接深度神经网络(FC-DNNs),在预测药物性能方面比单任务FC-DNNs表现更好。特别是,基于自动编码器(AE)的模型和赛诺菲-安万特FC-DNN模型,在两个多任务基准数据集(CYP同工酶抑制剂数据集和肝微粒体清除数据集)上获得了出色的性能。在多任务学习中,MolMapNet可能在多任务学习中更合适的MolRs竞争。因此,研究者在这两个多任务数据集上,开发了多任务MolMapNet OOTB模型,并分别采用相同的数据分割、评价度量和多任务训练方法,与基于AE和Sanofi-Aventis模型的公布性能,进行了比较(表3)。在CYP450数据集上,MolMapNet在五项任务中的三项上表现低于基于AE的FC-DNN模型,但三项表现不出色的任务的受试者工作特征曲线下面积(ROC-AUC)值与基于AE的FC-DNN模型具有可比性。在肝脏微粒体清除任务上,MolMapNet在所有三个任务上都优于Sanofi-Aventis模型。总的来说,MolMap Fmaps和多任务MolMapNet体系结构,在多任务学习药物性能方面是有竞争力的。
MolMapNet深度学习模型的
单路径和双路径相比较
图4显示了MolMapNet OOTB模型,在MoleculeNet数据分割和AttentiveFP数据分割的11个基准数据集上的性能比较。对于回归任务,在5个回归数据集中的3个上,仅MolD-单路径(MolMapNet-D)模型的表现,与联合MolD和FF双路径(MolMapNet-B)模型相当或更好。对于分类任务,仅FF单路径(MolMapNet-F)模型,在6个分类数据集中的4个上的表现与MolMapNet-B模型相当。有趣的是,与MolMapNet-D模型相比,MolMapNet-F模型在回归任务中表现稍差,但在分类任务中表现稍好。有趣的是,MolMapNet-D模型的输入Fmap是定量MolDs(例如分子量),而MolMapNet-F模型的输入Fmap是分类FFs(0或1)(图2)。因此,MolMapNet-D模型往往更适合于回归任务等连续预测器,而MolMapNet-F模型更适合于分类任务等分类预测器。总的来说,MolMapNet-B模型利用了这两种输入类型(MolD和FF fmap),因此在回归和分类任务中具有高度的竞争性。
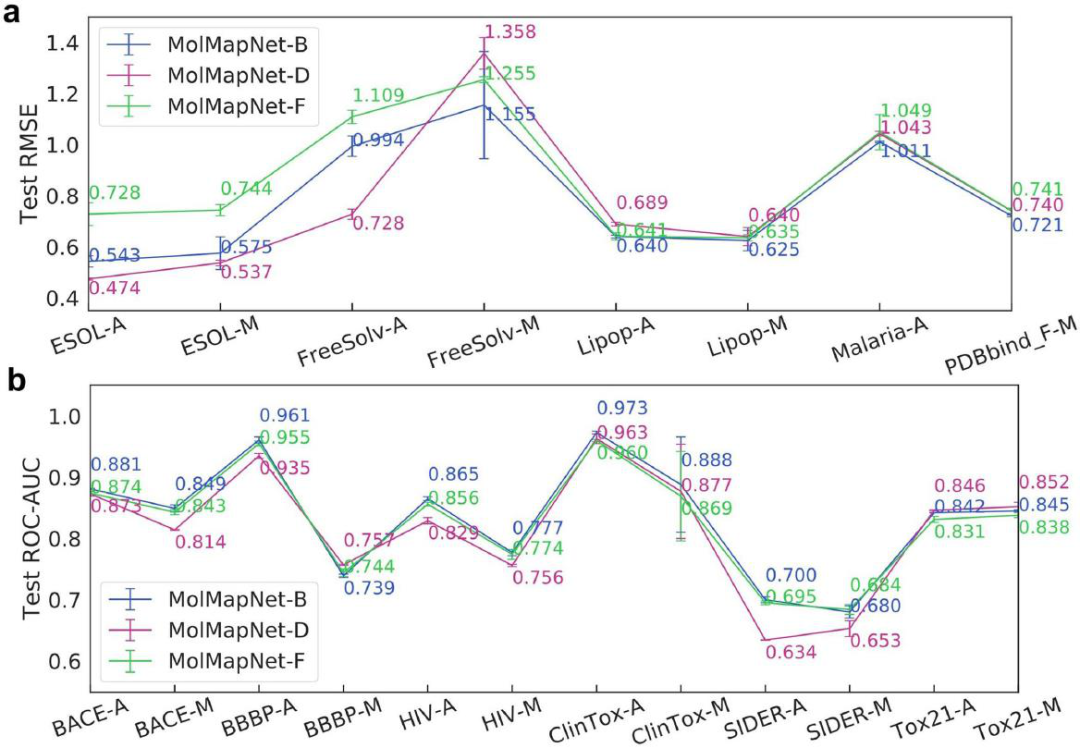
图4. 单路径MolMapNet-D、MolMapNet-F和MolMapNet-B模型在基准数据集上的性能
图片来源于Nat. Mach. Intell.
相对于k-近邻模型的MolMapNet深度学习性能
MolMapNet OOTB模型的表现,部分归因于预训练的MolMap Fmap。为了探讨这种预训练对分类性能的影响,研究者对有和没有预训练的FF Fmap模型进行了评估。具体而言,MolMapNet-F OOTB模型与k-近邻 (kNN)模型的性能进行了比较,kNN模型与MolMapNet-F模型,在5个分类基准数据集(BACE、BBBP、HIV、ClinTox和SIDER)上,建立的相同的三个FF集以及分子网数据分割。MolMapNet-F OOTB模型的ROC-AUC值分别为0.843/0.851、0.744/0.677、0.774/0.728、0.869/0.806,BACE、BBBP、HIV、ClinTox和SIDER数据集的ROC-AUC值分别为0.684/0.630。相比之下,对于BBBP、ClinTox、HIV和SIDER数据集,分子网和Chemprop模型中最好的ROC-AUC值分别为0.806、0.738、0.776、0.864和0.676(表2)。因此,MolMap预训练有利于增强药物特性的学习。值得注意的是,所有kNN模型都表现良好,其中BACE模型优于GCN和MolMapNet OOTB模型。这些表现部分归因于,通过三个MolMap选择的FF集所选择合适的MolRs。之所以选择这些FF集,是因为它们的MolMap Fmap与其他FF集相比,呈现出不同的、更密集的聚集模式,从而促进了增强学习。
优化的与OOTB MolMapNet深度学习模型
通过粗粒度网格搜索优化MolMapNet-B模型的4个超参数:用于增强特征表示的UMAP特征生成参数,批量大小用于改进收敛性和性能,第一个卷积层的核大小为更有效的接受域,以及密集层的宽度用于改进了多任务分类。首先,分别对每个超参数进行优化。对ESOL、BACE和Tox21数据集的UMAP优化表明,减少邻居数量,通常通过提高局部特征分布的精度,来提高性能。对ESOL和FreeSolv数据集的批量大小优化表明,减少批量大小可以加速收敛并提高性能。在BACE数据集上的核大小优化表明,增加核大小可以通过更有效的接受野增强学习。密集层宽度优化表明,节点数量的增加提高了多任务性能,扩展了信息处理能力,超出了主要为单个任务定制的OOTB设置。其次,在8个数据集(ESOL、FreeSolv、Malaria、BACE、HIV、MUV、PCBA、ChEMBL)上使用MoleculeNet和AttentiveFP数据拆分,对4个超参数进行了综合优化。在优化前,12个MolMapNet-B模型中只有5个优于SOTA GCN/GAT模型。优化后的9个模型在相同的数据分割下,优于SOTA GCN/GAT模型。其中,FreeSolv模型的RMSE从1.075降低到0.916,降低了14.7%;MUV模型的精确回忆曲线 (PRC-AUC)值下面积从0.109增加到0.158,增加了44.9%。对于三个表现不佳的MolMapNet-B模型,它们的MolMapNet-D对应模型实质上更好,其中两个模型在相同的数据划分中优于SOTA AttentiveFP GAT模型(在ESOL数据集上RMSE为0.477对0.486,在FreeSolv数据集上RMSE为0.728对0.773)。
MolMapNet对新化合物的泛化能力
MolMapNet对从ChEMBL数据库中,提取的216和179种新的BACE高效和低效抑制剂(nace数据集)进行评估。NBACE和BACE数据集之间的分子相似性模式通过TMAP可视化,NBACE和BACE数据集的化合物谷本系数为0.23和0.61。谷本系数<0.7,通常表示遥远的相似性。采用NACE数据集对经BACE训练的MolMapNet-F模型进行性能测试,并与D-MPNN和AttentiveFP模型进行比较。MolMapNet-F模型的灵敏度和特异性分别为70%和84%,而GCN D-MPNN模型的灵敏度和特异性分别为48%和81%,AttentiveFP4模型的灵敏度和特异性分别为63%和63%。
MolMapNet学习了
深层潜在特征和重要的输入特征
为了探究MolMapNet学习的深层潜在特征和重要输入特征,研究者首先分析了在ESOL数据集上训练的MolMapNet-D溶解度模型和AttentiveFP数据分割。主成分分析(PCA)对全局最大池化层(全连接层之前)的潜在特征进行分析,这些潜在特征是根据溶解度值进行聚类的。因此,面向任务的聚类是一种深层潜特征。根据排列算法和均方误差(MSE)度量计算的重要度得分,得到重要的输入特征。来自训练集和测试集的重要输入特征是相关的(Pearson r = 0.92)。将最重要的输入特征E-state、QED、电荷和拓扑指数聚在一起。E-state指数编码了与溶解度预测相关的拓扑环境和电子相互作用。QED描述符量化药物相似性,并显示口服药物的溶解度和渗透性。电荷描述符。反映了影响溶解度的离子相互作用。接下来,研究者分析了在BACE数据集上训练的MolMapNet-F BACE抑制剂模型的重要输入特征。排在前几位的重要FFs包括5个PubChemFP FFs和7个MACCSFP和PubChemFP FFs,这些FFs在BACE强效抑制剂中常见,但在低效抑制剂中较少。重要性得分前50名的FFs被映射到每个分子的单个原子和键上,可以用不同的颜色方案直观地显示出来,以揭示深度学习模型认为重要的子结构。在分析典型的2-氨基喹啉抑制剂及其结构上的近邻2-氨基苯并咪唑抑制剂时,结果表明,高效阻垢剂BACE_276的疏水碳链是BACE活性的重要亚结构,这与构效关系研究的结论一致。前50个重要特征。可用于提示BACE抑制支架。通过这种方法,收集到的26种临床试验BACE抑制剂药物中,有25种被鉴定为高效抑制剂,而剩余的药物PF-04976081由于是一种新型的分子支架,缺少一个可识别的高度重要的亚结构。因此,研究者的分析表明,MolMapNet可以很好地捕获对药物相关特性重要的摩尔体,从而增强对这些特性的预测。
展望与结论
药物特性的准确学习和预测是一项具有挑战性的任务,特别是对于低数据案例和新颖的预测任务。适当的MolRs,对于增强学习和预测能力是至关重要的。基于图形的MolRs从头学习,已经取得了显著的进展。在这些显著的进步之上,对MolRs的更广泛的探索,有助于为增强学习和预测能力提供更多的视角。特别是,通过对广泛的人类先验知识基础进行更广泛的学习,适当的MolRs可能来自于MolDs中丰富的结构、结构和物理化学性质以及FFs中多种多样的子结构,从而促进了对药物特性的深度学习。MolMap等新工具促进了MolDs和FFs的特征生成,并将其转化为2D Fmap,捕捉深度学习应用的分子特征的内在相关性。
在这些Fmap的基础上,共享权CNN架构可以用于增强学习和预测药物特性。为了减少技术障碍并支持更广泛的应用,研究者希望将深度学习模型开发为OOTB工具。因此,研究者开发了基于CNN的深度学习MolMapNet模型,用于药物属性的OOTB深度学习,在26个基准数据集的大部分上,与已建立的模型具有很强的竞争力。深度学习模型探索了,更广泛的表示和特征生成策略(例如,基于图的DNN指纹),并不断取得进展。对这些和已建立的策略的集体探索,使更强的深度学习和预测药物和其他分子特性成为可能。
工具下载地址
https://github.com/shenwanxiang/bidd-molmap
https://codeocean.com/capsule/2307823/tree
参考文献
Shen, W. X., Zeng, X., Zhu, F. et al. Out-of-the-box deep learning prediction of pharmaceutical properties by broadly learned knowledge-based molecular representations. Nat. Mach. Intell. 2021, ASAP. DOI: 10.1038/s42256-021-00301-6.