
引言
全细胞建模与仿真,是21世纪的重大挑战之一,更是系统生物学的终极目标。利用现有的实验确定的信息,进行的详细的全细胞模型及其模拟,可用来探索未知的、未观测到的生物系统区域,从而进一步扩展了人类现有的生物知识的极限。
在组织建模、细胞建模、神经生物学等方面,数学建模,即生物过程的数学表示,已被证明是湿实验室实验的一个非常成功的替代方法。而设计和模拟一个广泛的生物全细胞模型,是一个非常耗时的过程。当前,尽管存在一些基于随机模拟的方法,如E-Cell、虚拟细胞、GEPASI和原始细胞的布朗动力学模拟等,但这些仅限于小的假设模型。但是,只有像Markus Schwehm在2001年预测的那样,将问题并行化并利用现有的高性能计算(HPC)系统,大量模型和仿真数据的极端调节,才能进行全细胞仿真。因此,必须以这样的方式构建、设计和处理全细胞模型,以便在合理的时间内,合理地利用高性能计算系统,来执行并行的全细胞模拟。
细胞功能,是由称为功能模块的不同相互作用的分子群来执行的。有时,多个功能模块,共同参与完成某一细胞功能。组成每个功能模块的相互作用分子,被分配到特定的细胞区域或隔间,它们在其中发挥功能,而它们从指定区域,穿越到细胞内其他区域的概率非常低。这一观察帮助人们得出结论,如果能够最小化功能模块之间的相互依赖性,那么每个功能模块都可以被独立地模拟。因此,全细胞建模,可以看作是每个单元,只包含一个功能模块的亚细胞建模的总和。
由于全细胞模拟的计算时间,取决于许多因素,包括分子的数量、细胞的大小和模拟的持续时间,因此,正确地对整个细胞建模是很重要的,这样才能有效地利用现有的高性能计算架构。Markus Schwehm预计,在196个CPU的开普勒簇上,模拟大肠杆菌的全细胞周期大约需要24天。在大肠杆菌的细胞周期中,在4000万个细胞质分子中,发生了1016个生化反应。植物和动物细胞的成分,大约是大肠杆菌的1000倍。因此,与大肠杆菌细胞相比,这些细胞更复杂,它们的模拟计算也更昂贵。
全细胞模拟,需要处理大量的模型和模拟数据。因此,在小的理论模型中能够很好执行的方法,在实际的大的全细胞模型中变得难以管理,除非利用现有的HPC系统,同时将问题并行化处理。因此,最大限度地利用高性能计算系统,是使全细胞模拟可行的绝对前提。这篇文章中,研究者提出了一种基于随机模拟的方法,通过在合理的时间内有效地利用现有的高性能计算系统,可以模拟整个细胞的大量分子。
此文中,研究者首先描述了用于模拟的整个细胞的数学模型。随后,研究者介绍了并行实现的计算方法和细节。最后,研究者提出了,优化全细胞模拟的方法。(由于篇幅原因,我们这里将详细介绍后面两个部分,第一部分可详见原文)
计算细节
在此,研究者选择了单细胞细菌大肠杆菌,展示了他们的模型。与含有3748个蛋白质的大肠杆菌(K12)的蛋白质位点图(PLG)相同,研究者设计的细胞,由3748个蛋白质分子组成。研究者主要目标,是模拟由这3748个分子单独组成的所有功能模块。尽管大肠杆菌细胞由大约188个蛋白质模块组成,但研究者演示了不同数量模块的结果,以便对模拟器进行深入的性能分析。为了可视化和易于理解,生成了一个进程监控日志(PML)文件,该文件允许用户在使用分子可视化软件PyMOL进行模拟之前,可视化细胞模型的整个设置。每个子单元类似于一个虚拟容器,它模拟分配给它的功能模块。研究者提出的方法,分别执行每个虚拟子单元,边界条件因子单元之间的不同而不同,这取决于它是驻留在单元中边缘还是中心。在研究者的CUDA实现中,在内核模块中设置了一些标记,可以检测分子从一个亚细胞到另一个亚细胞的遍历。因此,每当需要在核之间传递消息时,模拟数据的当前状态,就从GPU转移到CPU。然后CPU相应地更新数据结构,并将其传输回GPU进行进一步处理,主要用于下一个模拟时间瞬间。
研究者对这里讨论的所有模拟,都使用了固定的参数集。模拟的总步骤为1000步,分子之间的碰撞被认为是非弹性的,即COR(回弹系数) = 0。研究者对全细胞预聚类进行CPU模拟,串行实现了所有3748个分子。然后,进行并行仿真,研究者生成了空间定位的PLG簇。
结果讨论
2.1 负载均衡
研究者将集群划分为不同数量的核,如图1所示,这样每个核的工作负载都是相似的。当使用两个GPU核时,研究者将其中一个核分配到,最大的包含1693个蛋白质的簇中,而剩下的核分配到另外三个包含1481个蛋白质的簇中。类似地,当使用四个GPU核时,研究者将两个核分配到最大的簇中,每个核分别处理847和846个蛋白质的计算。由771个蛋白质组成的簇被分配到第三个核,而第四个核处理另外两个共包含710个蛋白质的簇。按照相同的步骤,研究者在8、16、32、64、128和256个GPU核之间平衡工作负载。同时,研究者从最大的四个簇的3174个粒子中,移除294个随机粒子,并将剩下的2880个粒子,分配给所有GPU的2880个CUDA核。
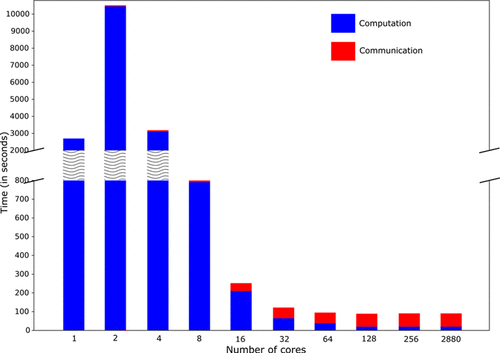
图1. 绘制模拟3748个大肠杆菌分子所需的计算和通信时间图
图片来源于JCIM
2.2 观察
从图1可以看出,利用GPU的两核模拟PLG集群系统所需的计算时间,几乎是单核CPU所需的3.9倍。当开始使用两个或更多的GPU内核时,内核之间的通信,会根据分子的运动而产生。因此,巨大的内存传输(CPU到GPU,反之亦然)开销和核间通信,是导致两个GPU核相对于单个CPU核计算时间,要求如此之高的两个主要因素。对于4个GPU核,计算时间与CPU仿真时间相近。四个GPU核改进后的数据处理和计算速度隐藏了内存延迟,因此相应的时间要求低于两个GPU核。当使用8个或8个以上的GPU CUDA核时,由于在GPU上的数据处理和计算速度上开始大幅提高,获得了良好的性能。随着计算单元数量的增加,分配给核的分子数量也越来越少。这导致了属于不同核的分子之间更多的相互作用,从而导致越来越多的核间通信。虽然2核、4核和8核的通信时间较短,但通信时间从16核开始增加,但在128核左右趋于稳定。因此,对于当前的小区配置和所选的仿真参数,在128核GPU上进行仿真时得到了最优的性能。为了便于理解,研究者在每个时间步结束时,收集模拟数据或每个分子的轨迹,并使用一种化学文件格式存储它,称为XYZ文件格式,扩展名为a.xyz,它存储了分子的笛卡尔坐标,可以很容易地在PyMOL的视频中显示出来。研究者对老鼠和人类这两种高等生物,进行了可扩展性分析。褐家鼠(大鼠)的PLG由9554个蛋白、652738个蛋白-蛋白相互作用(protein-protein interactions, PPIs)和598个紧密连接的簇组成。同样,对于智人(人类),PLG由41550个蛋白,8943744个PPIs和711个簇组成。
图2和图3分别为模拟大鼠9554和人41550分子,所需的计算和通信时间。对于大鼠来说,计算和通信时间都稳定在256个GPU核左右,而对于人类来说,则稳定在1024个GPU核左右。正如预期的那样,核需求随着蛋白质数量、它们之间的相互作用以及PLG的增加,从低等生物到高等生物而增加。
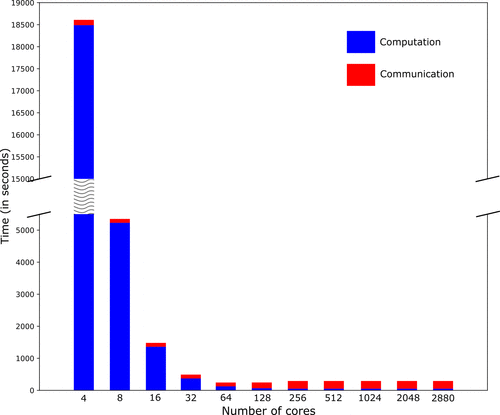
图2. 模拟褐家鼠(大鼠)9554个分子所需的计算和通讯时间图
图片来源于JCIM
计算优点
亚细胞内动力学所需的计算时间,总是少于亚细胞间动力学(图1),因为在亚细胞内动力学中,分子的运动受到模拟子空间的限制。因此,GPU核之间不需要进行信息传递。当PLG簇之间的连接数量非常少(可以忽略)时,就可以完全避免这样的串扰,只限制它们各自亚细胞内分子的运动。研究者的模拟器也可以处理这种场景,这减少了通信时间要求,并提供了更高效的计算模拟。
此外,研究者的方法非常灵活,可以很容易地用于对高等生物进行并行的整体模拟。从上述例子中可以明显看出,对于任何生物,研究者都可以计算其PLG、空间定位簇,并利用研究者提出的计算框架并行执行其细胞动力学。
对于开放系统,可能需要动态的PLG,这取决于进入/退出系统的蛋白分子浓度。研究者所提出的模拟器,可以使用基于哈希的元胞字典数据结构,来处理这种情况。首先构建整个细胞的PLG,然后将其分割成紧密相连的簇。最初,关于每个集群及其相应组件的信息,存储在元胞字典(CD)数据结构中。在模拟过程中,每当一个蛋白质分子从系统中退出时,它对应的细节就会从CD中删除。或者,在进入时,分子的细节在相应的簇/亚细胞的CD中更新。因此,PLG的动态特性取决于,由CD处理的进入/离开系统的蛋白分子浓度。
在这里进行的并行全细胞动力学,和图1中提到的计算时间分析,证明了对PLG进行聚类,并利用现有HPC系统的单独计算单元并行模拟这些聚类。事实上,研究者所提出的方案,适用于任何生物的任何类型的生物网络,并且整个方法保持不变。并行全细胞仿真所面临的巨大计算开销的一个恒定因素始,终是CPU到GPU的内存传输开销,当将仿真框架从CPU计算转移到并行GPU计算时,不能忽视这个因素。在并行单元模拟过程中,研究者可以控制的另一个昂贵的计算因素,是核心间通信的数量。因此,应该尽可能减少集群间连接的数量,因为:即使尽最大努力微调集群过程,由于蜂窝网络的高度互联性,仍然不能完全消除集群间连接的数量。然而,在一个特定的时间步长,没有任何集群间的移动,不需要从GPU到CPU的内存传输,反之亦然,这是在研究者的模拟器中使用的。
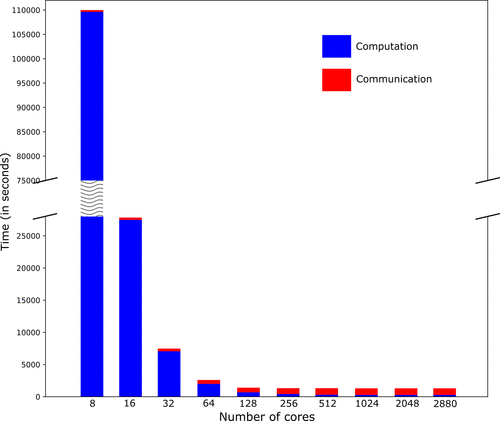
图3. 模拟41550个智人(人类)分子所需的计算和通信时间图
图片来源于JCIM
范例研究
图4中展示了用研究者的模型在16个亚细胞中模拟全细胞蛋白动力学的范例。研究者用PyMOL生成了仿真轨迹并将其可视化。在开始模拟之前,研究者将整个细胞分为16个不同大小的亚细胞。每个亚细胞由不同数量的蛋白质分子组成,它们用不同的颜色标记,如图4所示。在HPC系统的单个计算单元上,分别对每个子单元进行了模拟。最初,所有的蛋白质分子都处于静止状态,它们被分配到三维亚细胞空间的随机笛卡尔坐标中。当模拟开始时,力作用在分子上,分子开始移动。随着模拟的进行,系统定期更新。这里不允许亚细胞间运动,因此,蛋白质只能在它们分配的细胞间隔内运动。分子倾向于非弹性碰撞,因此,模拟中的蛋白质分子也会非弹性碰撞并粘在一起(如图4中黄色圆圈所示),其速度也随之改变。随着模拟的进行,碰撞的分子可能会再次分解成单独的分子,这取决于作用在碰撞物体的团簇上的力。
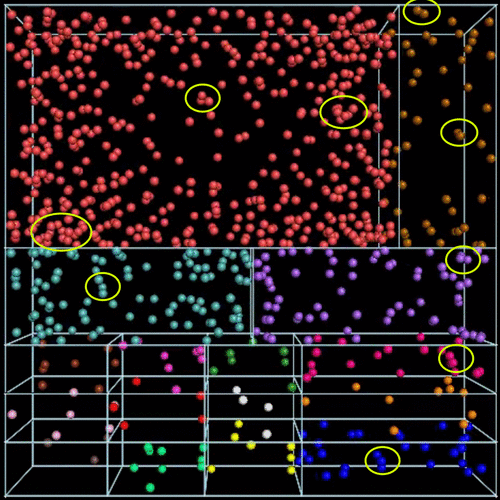
图4. 从细胞初始状态开始的90次迭代后,描述平行全细胞蛋白动力学的示例模拟
图片来源于JCIM
展望与结论
在此,研究者提出了一种有效地利用现有并行硬件架构,并利用整个细胞的动态特性的并行全细胞模拟框架,从而引导人们走向细胞动力学的新概念。研究者选择了力场,运动方程和一个积分算法,并提出一个算法,来检测和解决碰撞。然后,提出如何利用现有的高性能计算系统,来进行最优的并行全细胞模拟。
研究者观察到,对于大肠杆菌,当至少使用了128个核的GPU时,得到了一个最佳的模拟时间,并且计算和通信时间都变得稳定。对于大鼠和人类,分别用256和1024个GPU核,实现了计算和通信的稳定性。此外,可以通过尽可能减少核间通信和计算时间,来达到最优的全细胞模拟时间。以空间定位的生物网络的形式,聚集所有可能的细胞信息,通过最小化集群间连接的数量,聚集它们紧密相连的子组件,在单独的GPU核上模拟每个集群,通过CD数据结构有效地处理核间通信,为利用高性能超级计算架构,进行并行全细胞建模和仿真,提供了新的研究视角。
在本文中,模拟是在没有溶剂的情况下进行的。将BD(布朗动力学)应用到仿真工具中,产生了充当溶剂作用的随机力。研究者展望,未来该方法应该进化到,支持任何种类的溶剂以及各种参数,如溶剂的粘度和溶液的温度等。这样,就可以利用一个合适的力场,进行全细胞模拟和特征值分析,以确定时间步长与细胞动力学之间的关系。通过所有这些循序渐进的实现,未来将能够进行全细胞模拟,准确地模拟真实的活细胞,从而进一步拓展,现有生物学知识的极限。研究者相信,此处提出的计算框架,只要有足够的实验数据,对于任何生物的生物网络都是有效的,并且可以扩展到任何CPU-GPU架构。
参考文献
Barnali Das and Pralay Mitra. High-Performance Whole-Cell Simulation Exploiting Modular Cell Biology Principles. J. Chem. Inf. Model. 2021, ASAP. DOI: 10.1021/acs.jcim.0c01282.