
先睹为快
作者
日本武田制药Michiko Tawada
研究对象
O-连接的N-乙酰葡糖胺水解酶:O‑GlcNAcase (OGA)
计算方法
同源模建(Homology Modeling)
分子对接(Docking Calculations)
基于形状的相似性搜索(Shape-based similarity search)
虚拟筛选(Virtual Screening)
WaterMap分析(WaterMap Analysis)
分子动力学模拟(Molecular Dynamics (MD) Simulation)
计算软件
ROCS version 3.2; Maestro version 10.4
计算流程
首先基于已知OGA抑制剂的结合构象,应用ROCS筛选了包含2000000分子数量的化合物库,初步选出约100000个化合物;然后,将化合物与人类OGA蛋白对接,选出与蛋白有相互作用的7万个化合物;接着,根据设置的理化参数过滤化合物,保留了55000个化合物;随后剔除假阳性化合物(PAINS),筛选出53000个化合物;最后,将化合物分为2681个簇,并对每个簇中排名靠前的2681个化合物进行生物活性分析。
引言
众多周知,Tau蛋白的过度磷酸化是神经退化性疾病的主要特征。然而,Tau蛋白在体内除了被磷酸化修饰还可被O-GlcNAc 糖基化修饰。研究发现,如果提高Tau蛋白的O-GlcNAc水平,可有效降低Tau 蛋白的磷酸化程度。O-GlcNAcase(OGA)是生物体内唯一水解蛋白质O-GlcNAc 糖基化修饰的酶,因此,靶向OGA的抑制剂研究逐渐成为开发神经退化性疾病药物的热点话题。
目前报道的OGA抑制剂(图1)多是糖衍生化的底物类似物,由于结构中多羟基取代特征,小分子不易透过血脑屏障。在2019年,经过结构修饰获得的MK8719抑制剂能突破血脑屏障并具备了较好的药代动力学特征,将进行下一阶段的临床试验。然而,具有高效价和更好的药代动力学特征的非糖类化合物亟需开发以帮助表征OGA。
近十年来,目前已经报道了几种OGA-抑制剂复合物晶体结构,由于人类OGA的晶体结构在之前尚未报道,作者以细菌OGA为模板蛋白同源模建了人类OGA的蛋白结构。基于蛋白结构,可以通过对接小分子库筛选化合物;基于配体结构方法,通过晶体结构配体的结合构象,可以确定配体的空间和静电特性,应用药效团或形状相似性筛选类似物。为了提高虚筛的命中率,作者结合了多种方法的虚拟筛选手段;并通过药物性质和中枢神经系统(CNS)渗透的物理化学参数对化合物进行过滤,获得了苗头化合物;对已鉴定的苗头化合物进行优化,以改善其药代动力学和亲脂性;最终获得了具有较高的亲和力和中等的亲脂性的新型OGA抑制剂化合物5i,有望成为tau蛋白介导的神经退行性疾病的治疗药物。接下来,我们将详细讲述作者的研究过程和结果。
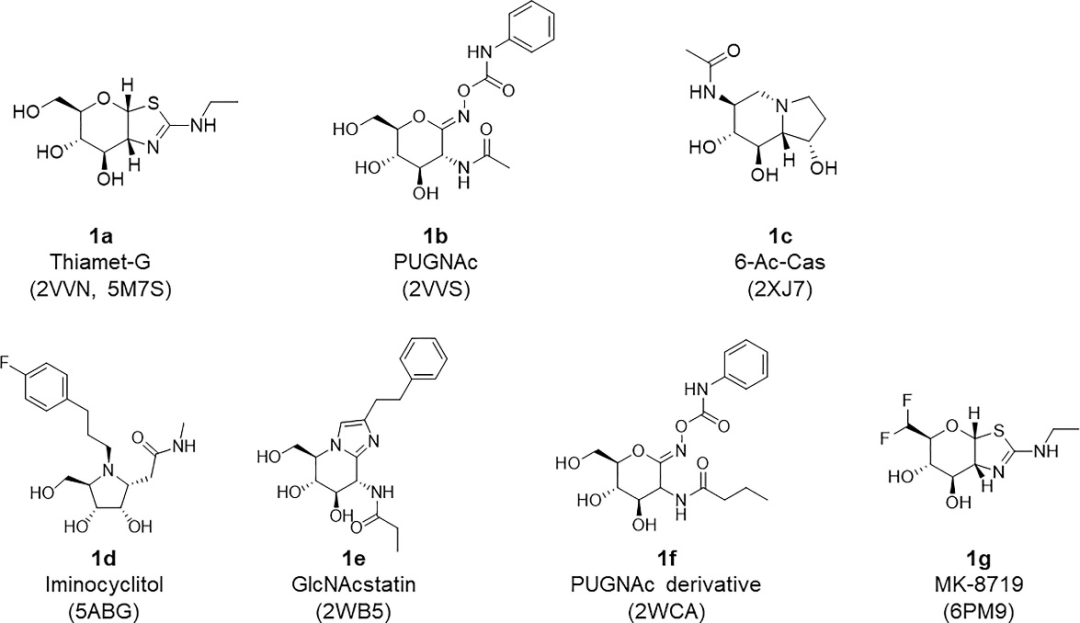
图1. 已报道的OGA抑制剂的化学结构
图片来源于JMC
研究内容
苗头化合物的获得
虚拟筛选的工作流程如图2所示。虚筛的第一阶段,作者基于化合物1a−1f(图1中)的结合构象,应用ROCS(形状相似性搜索的标准方法之一)筛选了包含2000000分子数量的化合物库,初步选出约100000个化合物;第二阶段,将化合物与人OGA蛋白对接,优先选择出与蛋白有相互作用的7万个化合物;第三阶段,设置理化参数(分子量≤350,AlogP≤5,氢供体数≤4,拓扑极性表面积≤125,氢受体数≤8,可旋转键数≤6)过滤化合物,保留了55000个化合物;第三阶段,随后剔除假阳性化合物(PAINS)筛选出53000个化合物;第四阶段,根据分子相似性将化合物分为2681个簇,并对每个簇中排名靠前的2681个化合物进行生物分析。

图2. 虚拟筛选的工作流程
图片来源于JMC
在10μM浓度下测定了虚筛所得2681个化合物的OGA抑制活性,鉴定出13个抑制率超过50%的化合物。随后的剂量-反应分析使作者最终鉴定出化合物2(IC50 = 41 nM),并证实其可以选择性抑制OGA。作者构建了化合物2与OGA的对接模型(图3),比较不同立体异构体、R-型和S-型以及可能pH范围内存在的质子化状态下的结合模式,其中N-1和N-4质子化的S-异构体表现出最合理的结合模式。在这种对接模式下,嘧啶的N-1和邻近的-NH与Asp285的羧基形成双齿氢键;甲基哌啶基团与Tyr219和Trp278形成类似三明治的π-π堆积;哌嗪基团插入到活性口袋的入口处。为了进一步阐述配体结合位点和蛋白质的性质,作者应用Schrödinger中的WaterMap工具对化合物2-OGA的对接模型进行结合热力学计算。在这种方法中,水分子置换到活性位点并分析自由能变化,可提供蛋白口袋相关性质的丰富物理见解。根据WaterMap计算结果(图3c),三个最不稳定的水分子1、2和3,聚集在由Tyr219和Trp278组成位置。根据Tyr219和Trp278周围的疏水侧链推测及SiteMap表明该位点是疏水的。在SiteMap绘制的表面图中,该位点主要被疏水表面覆盖;尽管这个疏水口袋显示出倾向于选择配体的疏水取代基,但是由WaterMap计算的水分子的性质表明杂原子也是可以被接受。在Asp285附近观察到ΔG值低于−1 kcal/mol的四个最稳定水分子,该位置的水分子显示出较大的焓增益,水分子1′和4′与Asp285形成氢键,并且与化合物2中的氨基嘧啶基团的氮原子有很好的重叠;在口袋入口处的残基Val255和Tyr286周围观察到具有中等自由能值的水分子,这表明口袋入口允许各种取代基;附着在手性碳上的甲基对结合亲和力没有显著贡献。
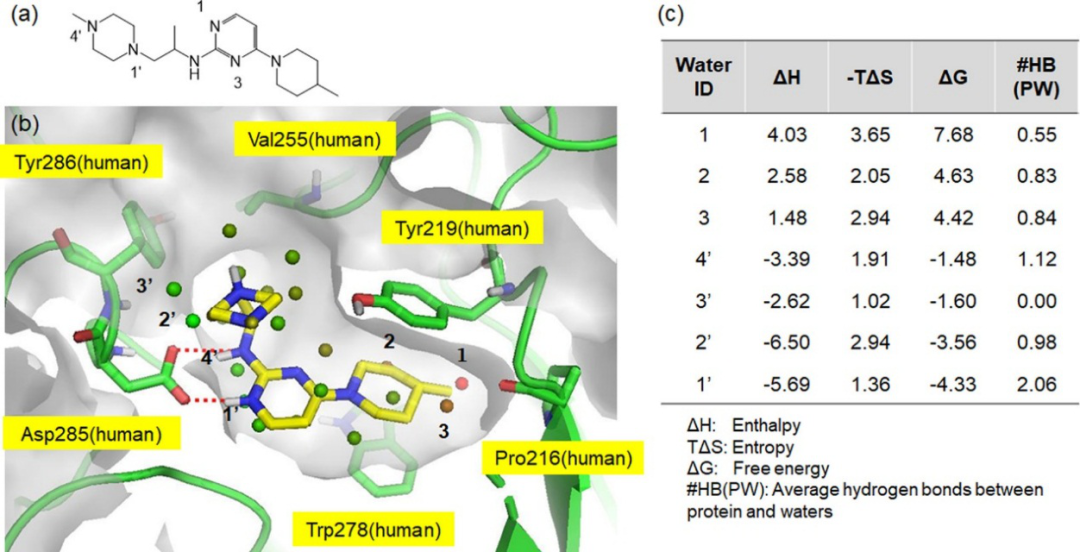
图3. 化合物2与OGA的对接模型
图片来源于JMC
先导化合物的发现和优化
基于WaterMap计算结果,作者将化合物2中哌嗪环替换成了苯乙基,并且去掉了手性碳原子的甲基得到了化合物5a,其仍保持对OGA的抑制作用。作者基于5a的化学结构进行了后续理化性质的优化,重点考虑了配体亲脂性效率(LLE)。为了降低配体的亲脂性,引入了六元环胺,如吗啉和甲基哌嗪;引入极性取代基对哌啶进行了进一步的修饰,羟基或甲氧基的引入没有产生有益的效果;另一方面,根据WaterMap计算数据展示苯乙基可以进行不同的修饰,用含氮杂环化合物修饰得到化合物5i(IC50=46nm;LLE=5.2),其具有理想LLE值的强效OGA抑制剂的特征。通过对化合物5i在细胞中的药效、选择性和药代动力学进行了详细的表征,结果表明5i是有效性的OGA抑制剂,并具有良好的血脑屏渗透性。
化合物5a/i的结合模式分析
作者解析了化合物5与细菌OGA的X射线晶体(图5a)。与人类OGA同源模型相比,化合物5a在晶体结构中表现出类似的蛋白质-配体相互作用模式;氨基嘧啶与Asp344(对应人类OGA中的Asp285)形成双齿氢键;甲基哌啶基与Tyr282和Trp337形成π-π堆积,与蛋白形成紧密的疏水作用;两种结构之间的差异在于位于门控区的Asp242(对应于人类OGA中的Asp174)的构象;值得注意的是,在上述晶体结构中还观察到小分子自身嘧啶核和末端苯环之间形成分子内π-π堆积,这种堆积作用有助于保持5a的刚性构象。将苯环替换为吡啶环(见5f和5g),其特点是π-电子密度较低,降低了π-π堆积效应。
作者基于5a-OGA的晶体结构构建了5i与人类OGA的对接模型(图5b)。除了5a中观察到的与Asp285、Tyr219和Trp278(分别对应于细菌OGA中的Asp344、Tyr282和Trp337)的常见相互作用外;还发现末端甲基咪唑与蛋白口袋入口处的Val254和Tyr286形成了CH-π作用;与化合物5a相比,由于烷基连接长度减少,未形成分子内π-π堆积作用;但与Val254和Tyr286的CH-π作用弥补了亲和力,仍保留对OGA的抑制效力。作者进一步采用分子动力学方法(MD)拟了5i与人OGA同源模型的复合物,证实所预测的结合模式和关键相互作用的稳定性。
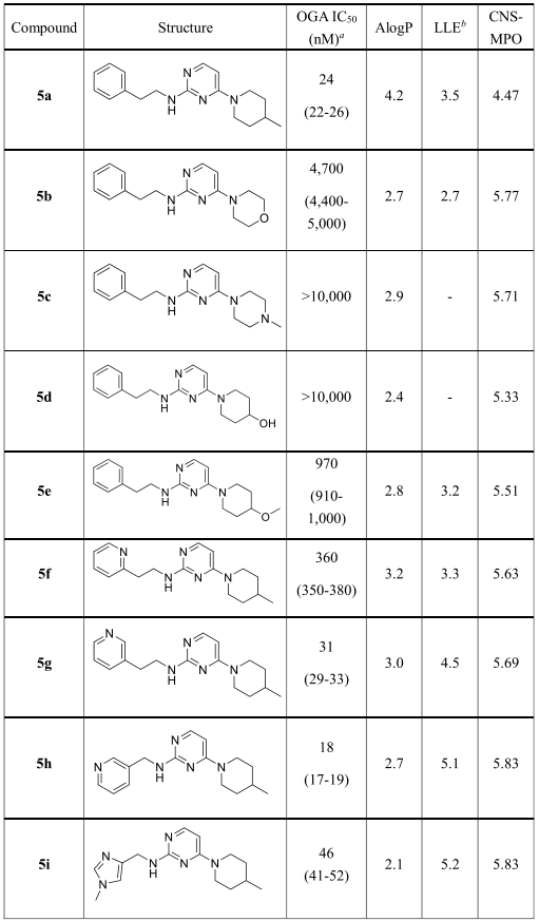
图4. 化合物5a−i的OGA抑制活性和配体亲脂性效率
图片来源于JMC
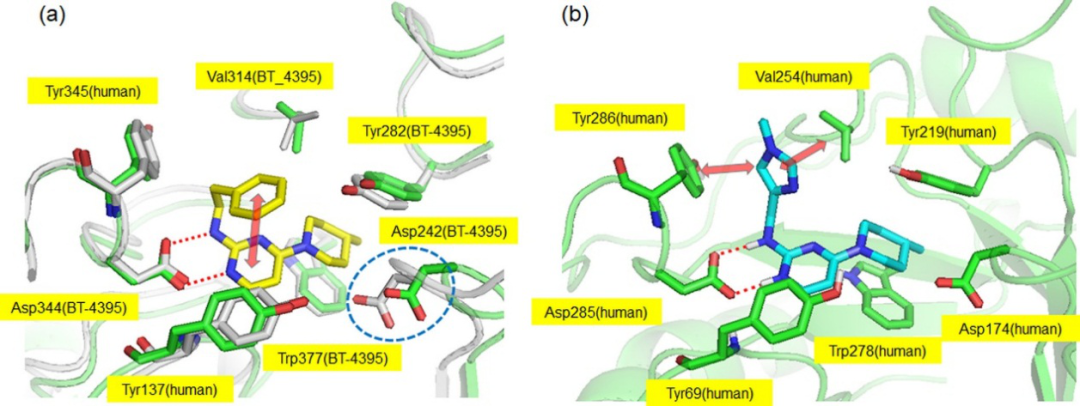
图5. (a) 化合物5a-OGA的晶体结构;
(b)化合物5i-OGA的对接模型
图片来源于JMC
结果与讨论
作者结合了多种方法的虚拟筛选手段,并通过药物性质和中枢神经系统(CNS)渗透的物理化学参数对化合物进行过滤,获得了苗头化合物;对已鉴定的苗头化合物进行优化,以改善其药代动力学和亲脂性;最终获得了具有较高的亲和力和中等的亲脂性的新型OGA抑制剂化合物5i。其可作为阐明OGA在神经退行性疾病中作用的体内化学工具,有望成为tau蛋白介导的神经退行性疾病的治疗药物。
参考文献
Michiko Tawada, et al. Discovery of a Novel and Brain-Penetrant O‑GlcNAcase Inhibitor via Virtual Screening, Structure-Based Analysis, and Rational Lead Optimization. J. Med. Chem. 2021, 64, 1103−1115.